摘 要: 酶作为生物催化剂参与很多重要的生理过程, 同时也是一类重要的生物分子。酶的活性分析对于疾病诊断和治疗具有重要意义。基质辅助激光解吸电离飞行时间质谱 (MALDI-TOF MS) 具有操作简单、分析速度快、灵敏度高和易于实现高通量分析的特点, 已被广泛用于各种组学研究和生物分子的检测, 在酶的检测和活性分析中亦发挥了重要作用。该文综述了国内外利用MALDI-TOF MS分析酶活性和进行药物筛选的策略, 总结了各种方法的优缺点, 提出了MALDI质谱技术在酶活性分析领域存在的问题和挑战, 并对其发展前景进行了展望。
关键词: 基质辅助激光解吸电离飞行时间质谱 (MALDI-TOF MS) ; 酶活性; 均相; 非均相;
Abstract: Enzymes as biocatalysts, participate in many physiological processes, which are important biomarkers. The activity analysis of enzymes is of great significance for diagnosis and treatment of many diseases. Matrix-assisted laser desorption ionization time-of-flight mass spectrometry ( MALDI-TOF MS) has been widely used in various omics studies and biological molecules detections because of its good performances in sensitivity, throughput, speed and operation. It also has played an important role in enzymes activity analysis. The methods of MALDI-TOF MS for enzyme activity analysis and drug screening are reviewed in this paper, the merits and disadvantages of each method are discussed, the problems and challenges existed are put forward, and finally the perspectives for MALDI-TOF MS in enzyme activity analysis are outlooked.
Keyword: MALDI-TOF MS; enzymes activity; homogeneous; heterogeneous;
酶分子产生于活细胞,其是具有特异识别底物和催化功能性质的蛋白质或RNA分子,亦是一种极其重要的生物催化剂。人体和哺乳动物体内至少含有5 000种酶,在凝血过程、蛋白质代谢、组织再生和免疫防御等许多生物活动中扮演着重要角色[1,2,3,4]。酶活性的改变与糖尿病[5]、阿尔兹海默症[6]、癌症[7]、艾滋病[8]等许多疾病密切相关。酶活性抑制剂可以调控生物过程,用于疾病的治疗,例如:人类免疫缺陷病毒 (HIV) 患者接受HIV-1蛋白酶抑制剂治疗,可以抑制疾病的发展,延长寿命。因此,发展高灵敏、高效的酶活性检测技术和方法,同时有效筛选酶活性抑制剂对于疾病的诊断和治疗具有重要意义。

传统的免疫分析法是通过检测酶的浓度间接确定酶活性。由于酶的浓度并不能直接代表酶的活性,因此常出现假阳性结果[9,10]。针对底物设计的荧光探针,制备过程相对复杂,且光学信号易重叠,不适合多种酶的同时检测[11,12]。质谱法是通过检测酶的底物和产物在酶作用下的信号变化来监测酶活性,方法准确性更高,而且适合多种酶的同时检测。近年来,利用电感耦合等离子体质谱 (ICP-MS) 技术将镧系元素标记在多种蛋白酶底物上,应用于多种蛋白酶的测定,取得了满意的结果[13,14]。液相色谱-串联质谱 (LC-MS) 结合多反应监测 (MRM) 技术亦被用于定量描述酶活动,被认为是“金标准”方法[15,16]。与ICP-MS和LC-MS相比,基质辅助激光解吸电离飞行时间质谱 (MALDI-TOF MS) 具有样品用量少、分析速度快、高耐盐和高通量的优势,在分析过程中可以简化复杂样品的分离、除盐和标记步骤。MALDI质谱离子化和MALDI靶板的修饰及生物芯片技术相结合,可以实现微量样品的在线分离和富集,以及高效的酶活性分析和药物筛选。本文综述了国内外利用MALDI-TOF MS检测酶活性和药物筛选的方法。这些方法主要分为两类:一种是基于非均相酶催化反应,将酶的底物固定在固体表面上,此表面与溶液相中的酶进行反应,再用MALDI-TOF MS进行检测;另一种是基于均相酶催化反应,即底物和酶在溶液相中反应,再用MALDI-TOF MS检测底物和产物。两种方法的原理示意图如图1所示。
图1 酶活性分析方法示意图
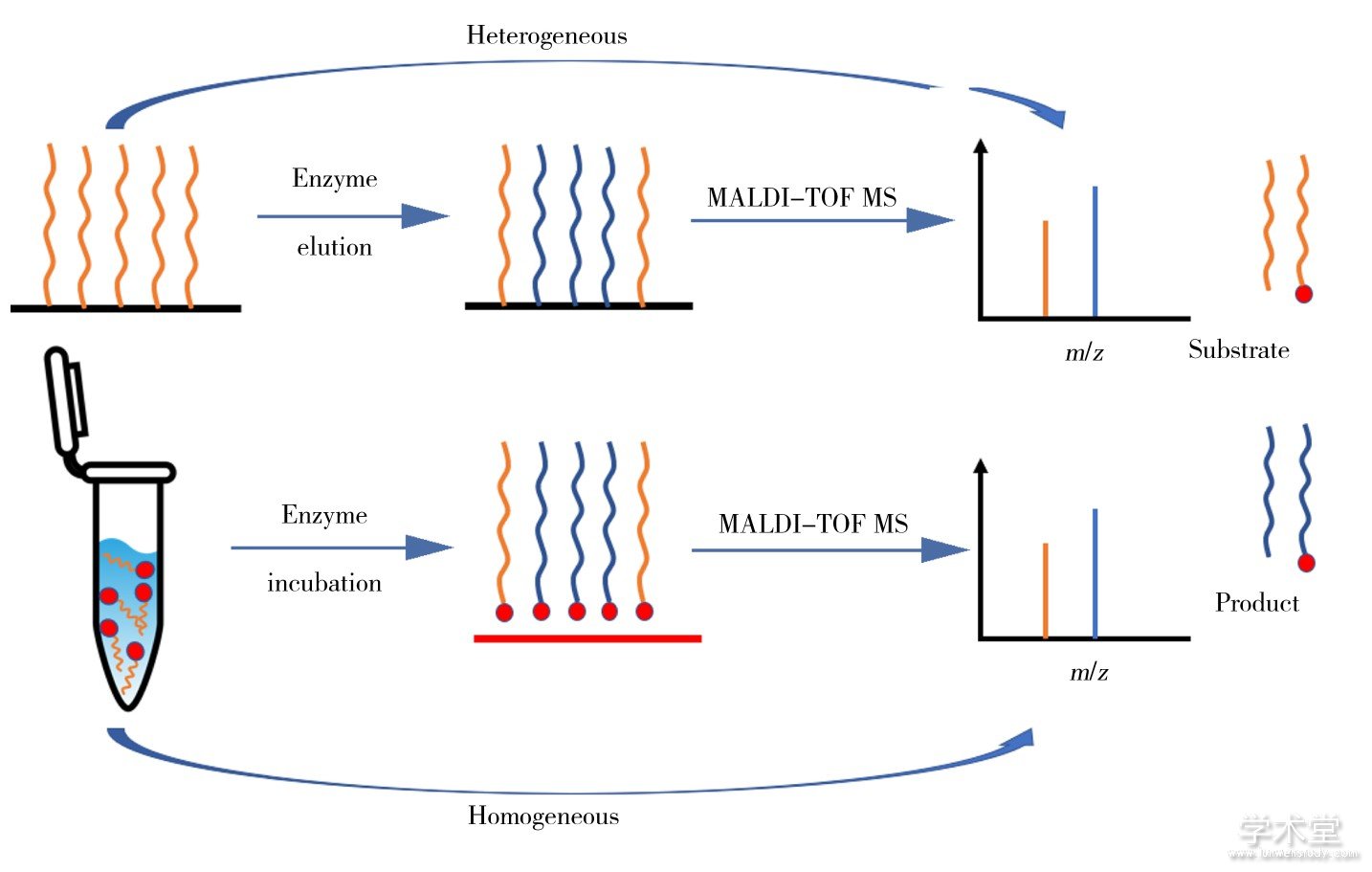
Fig.1 Schematic diagram of methods for enzyme activity analysis
1 、基于非均相酶催化反应
1.1 、通过共价作用固定底物
Su等[17]在金基底上通过Au-S作用自组装单分子层 (SAMs) ,此生物芯片非常适用于MALDI-TOF MS,因此称为SAMDI (Self-assembled monolayers for desorption ionization) 技术。如图2[18]所示,马来酰亚胺基团可与半胱氨酸残基反应从而固定蛋白激酶底物多肽,而寡聚乙二醇的设计能有效地抑制单分子层与蛋白质之间的非特异性作用,确保单分子层只通过固定的配体与蛋白质进行特异性相互作用。这种多肽芯片可用MALDI-TOF MS表征。蛋白激酶活性通过底物肽和产物肽的信号来监测。不同蛋白激酶底物的氨基酸序列不同,因此在一个芯片上固定多种多肽底物,可同时分析多种蛋白激酶。此方法评价了激酶抑制剂浓度对于激酶催化作用的抑制效果。
图2 单分子层合成与酶活性表征的方法[18]
![图2 单分子层合成与酶活性表征的方法[18]](http://www.xueshut.com/uploads/allimg/200114/36-200114151412457.jpg)
Fig.2 Strategy for synthesizing monolayers and characterizing enzymes activities by MALDI-TOF MS[18]
除了分析蛋白激酶,Kuo等[19]还将该方法运用于其他酶活性检测。通过分析CHRF巨核细胞 (Mk) 细胞裂解液中赖氨酸脱乙酰酶 (KDAC) 的活性,验证了该方法的可行性。利用类特异性去乙酰化酶抑制剂研究表明,分化晚期的巨核细胞表现为6种烟酰胺腺嘌呤二核苷酸 (NAD+) 依赖型的去乙酰化酶 (SIRTs) 活性明显下降,而另11种二价离子依赖型的去乙酰化酶 (KDACs1-11) 活性变化不大。该研究建立的平台可用于识别细胞裂解物中多种酶活性的变化。
除了将肽段作为金-硫单分子层的固定化底物外,Ban等[20]还设计了直接在生物芯片上制备寡聚糖阵列的方法,以快速有效地研究其相关蛋白的生物活性。该方法首先将含有苯酚末端的硫醇烷基链组装到金表面,酚基作为亲核试剂与第一个糖结合,再利用糖的末端高效地合成不同的寡聚糖阵列,并检测了β-1, 4-半乳糖基转移酶的特异性,如图3所示。此外,Kim等[21]通过将带有生物素标记的DNA底物固定在自组装的单层链霉亲和蛋白层上,制备了寡核苷酸阵列。该阵列在3’端具有正常匹配和错配碱基对的复合物,还包括该位点的一些删除和插入。用连接酶和三磷酸腺苷 (ATP) 或三磷酸类似物核苷处理阵列,再用基质辅助激光解吸电离质谱分析,比较了5'-探针链的腺化和连接的产物量。由此揭示了连接酶特异性,并发现使用ATP类似物可以提高酶的特异性。该方法可广泛应用于以核酸为底物的酶活性分析。Choi等[22]报道了一种基于在金上自组装单分子层的氯霉素 (CAP) 乙酰转移酶 (CAT) 活性的测定方法。在乙酰辅酶A存在下,CAT将单分子层上的CAP转化为乙酰化的CAP。利用MALDI质谱技术,通过观察CAP分子质量的变化,直接监测其转化过程。
图3 在芯片上合成低聚糖的方法[20]
![图3 在芯片上合成低聚糖的方法[20]](http://www.xueshut.com/uploads/allimg/200114/36-200114151353a3.jpg)
Fig.3 Strategy for the on-chip synthesis of oligosaccharides[20]
以上方法的特点在于将酶底物固定在惰性表面上,通过清洗去除干扰分子,很大程度上简化了样品前处理过程。这种策略基于MALDI-TOF MS的特点提供了一种简单、快速、高通量、免标记的检测方法。然而,此方法通过共价键 (Au-S) 固定底物,用MALDI-TOF MS检测时需经过Au-S键断裂过程。因此可能会导致底物和产物测定不完全,影响定量效果。Hu等[23]提出了一种肽编码的酶活性分析微板。将蛋白酶特异性的底物肽段直接固定在微板上,与目标蛋白酶作用后,从微板中释放出编码区域,以单氨基酸差异的相应肽段为内标直接定量分析。编码区域可作为唯一的“蛋白酶ID”用于识别相应的蛋白酶,裂解产物的量用于蛋白酶活性分析。以胰酶和糜蛋白酶为模型蛋白酶进行了多重蛋白酶试验,结果显示肽编码微板具有良好的选择性。该方法为多种蛋白酶的鉴定和活性定量分析提供了有力的工具,但主要针对蛋白水解酶,不适用于蛋白激酶等其他酶。
1.2、 通过非共价作用固定底物
除了在金-硫单分子层上利用含特殊官能团的硫醇烷基链来固定特殊肽段或寡聚糖来制备阵列,酶底物的固定化过程还可通过非共价作用完成。Sanchez-Ruiz等[24]将疏水自组装单层膜置于金板上作为支架,然后将一系列用脂质修饰的不同寡糖通过疏水-疏水相互作用固定在疏水单分子层板上,形成寡聚糖阵列 (如图4) 。基于酶作用后产物的不同,此阵列使区分不同的糖基水解酶活性成为可能,提高了聚糖阵列的结构多样性。随后,Beloqui等[25]利用该方法快速检测了环境样品中糖基水解酶活性和特异性。与其他方法相比,基于胶体的系统需要优化。此方法疏水表面和糖的标记更易制备,而且平面非多孔表面使酶易于接近反应表面和提高稳定性。该方法适于环境样品和复杂体液的功能分析。此外,该课题组[26]还对单分子层的材料进行了改良,选择商用的ITO涂层玻片,用3-氨基丙基三乙氧基硅烷 (APTES) 对其硅烷化,然后与硬脂酸偶联,为固定脂质标记的生物分子创造了疏水支撑层。这种单分子层基底可获得更高的表面覆盖度和疏水性。聚糖与疏水层结合能形成良好的斑点形态,为酶活性分析提供了良好的基础。然而MALDI-TOF MS检测技术对待测物的质谱响应性要求高,因此质谱响应信号直接影响定性、定量检测的准确性。Hu等[27]用两亲性的磷脂标记蛋白酶底物多肽,通过疏水相互作用固定在硬脂酸改性的ITO载玻片上,组装成不同半胱天冬酶的肽底物阵列,通过对酶裂解前后的肽段进行鉴定能够直接分析多种酶活性。由于磷脂在负离子模式下的离子化效率高,可得到很好的质谱响应信号。因此,该方法用于质谱成像获得了良好的可视化结果,具有多重酶活性分析和可视化特点,为酶的活性分析、耐药性鉴定和抗癌药物疗效评价提供了有力手段。
图4 利用疏水相互作用固定底物和酶活性表征的过程[24]
![图4 利用疏水相互作用固定底物和酶活性表征的过程[24]](http://www.xueshut.com/uploads/allimg/200114/36-20011415131H54.jpg)
Fig.4 The process of substrate immobilization by hydrophobic interaction and enzymes activity characterization[24]
2、 基于均相酶催化反应
2.1、 免标记法
相比于非均相酶催化反应,均相酶催化反应更有利于底物和酶的接触性。Ritorto等[28]用MALDI-TOF MS技术在体外分析了42个人泛素分解酶 (DUB) 对所有泛素拓扑异构体 (M1、K6、K11、K27、K29、K33、K48和K63-linked chains) 的活性以及特异性,并筛选了DUB抑制剂。此方法以同位素标记的N15-泛素为内标,对底物裂解的泛素单体进行定量,得到了良好的线性关系 (如图5) 。为减少干扰,在用MALDI-TOF MS检测之前对酶进行了去除。该方法免标记、快速,适合于大量筛查酶活性。
2.2、 反应后产物标记法
检测复杂样品如血液、细胞或组织中酶活性时,酶底物或产物的信号会被其他物质所干扰,因此开发对特定底物或产物进行分离富集以及选择性定量分析,并能够对多种酶进行同时分析的方法,对于酶活性评价和酶抑制剂筛选起着重要作用。Yang等[29]合成一种末端含有酰肼的氟化物探针,此探针与α-葡萄糖苷酶水解后的糖反应,并通过与氟化金单分子层表面的氟相互作用而被固定,然后用MALDI-TOF MS进行检测并测定酶活性。采用此方法测定了两种酶抑制剂的IC50值,证明该方法适用于酶抑制剂的筛选。然而在蛋白激酶催化过程中,由于反应混合物中含有丰富的非磷酸化肽,会对磷酸化肽的信号产生,因此Lv等[30]在酶催化反应之后,利用自制的二氧化钛纳米颗粒填充的毛细管柱,从复杂混合物中分离富集磷酸化肽,提高其信号强度。通过合成与底物肽分子量相似的肽段作为内标,定量分析了磷酸化效率。通过测定临床治疗慢性骨髓白血病的一种药物 (Abl的抑制剂) 的IC50值,验证了MS方法的有效性。然后利用该方法筛选了实验室合成的6个该药物类似物,得到的结果与酶联法吻合。Deng等[31]开发了用于MALDI-TOF MS定量分析多种蛋白激酶活性的同位素二甲基标记底物的方法。采用稳定同位素二甲基标记法,在一个MALDI靶点上实现了在单个和多重底物体系中,对不同反应时间下蛋白激酶A (PKA) 活性的监测。此外,通过完整的标记和精确的定量,可以准确得到动力学常数。该方法还成功地应用于激酶抑制剂的筛选。因此,该方法能够高通量、简单、快速、经济有效和准确地定量激酶和激酶抑制剂。Gregorius等[32]还提出了将金属标记肽用于多重酶活性测定。蛋白水解反应后,利用络合稀土金属离子使胰蛋白酶酶解反应后生成的剩余底物和肽产物形成金属螯合物进行标记。使用MALDI质谱对简单肽混合物进行定量,或使用LC-MALDI质谱对复杂底物混合物的标记肽进行定量,其准确度高,动态范围宽 (至少2个数量级) ,可用于监测随时间变化的产物形成和底物消耗过程。由于该方法具有多路复用能力和准确性,在生物化学和生物技术领域具有广泛的应用前景。
图5 DUB活性表征过程[28]
![图5 DUB活性表征过程[28]](http://www.xueshut.com/uploads/allimg/200114/36-20011415124aR.jpg)
Fig.5 The process of DUB activity characterization[28]
2.3、 反应前底物标记法
除了对酶与底物反应后的产物、剩余底物进行标记富集外,Ling等[33]报道了一种芘连接的底物肽,用于MALDI-TOF MS定量测定蛋白酶活性。该方法选择了一个序列为GGGGRG的胰蛋白酶特异性肽段与芘结合,形成了芘连接肽探针Py-GGGGRG。在胰蛋白酶存在下,Py-GGGGRG探针可以特异性水解成Py-GGGGR。芘的引入大大提高了肽段的电离效率,由于底物和产物的分子量相似,可以直接通过底物和产物的强度比例进行定量。并且利用芘的疏水性和共轭结构,用聚苯乙烯涂层的MAL-DI板可选择性地从复杂混合物中捕获芘肽段。该方法的线性范围宽,检出限低,已成功地用于尿液中胰蛋白酶活性的定量测定和胰蛋白酶抑制剂的筛选。此外,还利用探针Py-GGGGGGYG对糜蛋白酶进行了验证。该方法简单、高通量、定量准确,在多种蛋白酶活性测定和特定肽序列筛选抑制剂方面具有很大的应用潜力。众所周知,MALDI-TOF MS技术由于基质小分子干扰,在小分子区域酶底物或产物的测定方面受到限制。Wang等[34]报道了一种蒽醌-谷胱甘肽 (Aq-ECG) 探针,利用MALDI-TOF MS快速定量分析谷氨酰基转肽酶 (GGT) 的活性 (如图6) 。该探针是一种水溶性分子,由芳香蒽醌 (Ag) 和谷胱甘肽 (ECG) 通过一步硫醇-烯反应共价连接。蒽醌部分的作用不仅是在GGT催化裂解后提高目标离子的分子量,使目标离子出现在较高的m/z区域,避免基质离子的干扰,而且能显着提高肽的电离效率。此外,磁性氧化石墨烯可以选择性捕获蒽醌部分,将目标物与复杂混合物分离。使用一个蒽醌连接的甲基化谷胱甘肽作内标,可以定量分析GGT活性。利用该探针在健康人和肝癌病人血清样本中能检测到GGT,结果与酶联免疫吸附法测定结果一致。该方法还成功地应用于不同肿瘤、正常细胞内源性GGT含量的检测以及抗癌药物丁酸钠对GGT活性抑制作用的研究。利用该分子探针,开发了一种简单、灵敏、经济的方法,可以准确定量地测定GGT活性并筛选其抑制剂或抗癌药物。该方法也可改变探针部分,广泛应用于其他蛋白酶活性分析。
图6 Aq-ECG和Aq-ECA的合成路线 (A) 以及基于Aq-ECG的酶活性分析方法流程图 (B) [34]
![图6 Aq-ECG和Aq-ECA的合成路线 (A) 以及基于Aq-ECG的酶活性分析方法流程图 (B) [34]](http://www.xueshut.com/uploads/allimg/200114/36-200114151225216.jpg)
Fig.6 Synthetic routes of Aq-ECG and Aq-ECA (A) , and schematic flowchart of Aq-ECG based method for monitoring enzyme activity (B) [34]
表1中对上述主要的方法进行归纳总结,列举了不同方法的基本原理、适用范围和优缺点。
表1 酶活性分析方法性能的比较

3、 结论与展望
综上所述,由于MALDI质谱的高通量、高耐盐性等优点使得其在酶活性分析和酶抑制剂筛选中具有独特的优势。与其他传统分析方法相比,MALDI质谱能够直接检测酶催化反应的底物和产物的分子量信息,并且特别适用于多种酶的同时分析,在定性和定量分析方面均有广泛的应用前景。然而MAL-DI-TOF MS的重现性较差,用小分子有机基质时,由于结晶不均匀,会导致响应差异。基于MALDI-TOF MS的酶活性分析还需要提高底物或产物的离子化效率以及重现性,以提高酶活性分析的灵敏度以及定量准确性。因此,开发更简单、快速、效率高的酶活性分析MALDI-TOF MS平台在临床检测中具有重要意义。
参考文献
[1] Lopez-Otin C, Matrisian L M.Nat.Rev.Cancer, 2007, 7:800-808.
[2] Thornberry N A, Lazebnik Y.Science, 1998, 281:1312-1316.
[3] Puente X S, Sanchez L M, Overall C M, Lopez-Otin C.Nat.Rev.Genet., 2003, 4:544-558.
[4] Leung D, Abbenante G, Fairlie D P.J.Med.Chem., 2000, 43:305-341.
[5] Havale S H, Pal M.Bioorg.Med.Chem., 2009, 17:1783-1802.
[6] Yan R, Bienkowski M J, Shuck M E, Miao H, Tory M C, Pauley A M, Brashier J R, Stratman N C, Mathews W R, Buhl A E.Nature, 1999, 402:533-537.
[7] Friedl P, Wolf K.Cancer Res., 2008, 68:7247-7249.
[8] Haim H, Salas I, Sodroski J.J.Virol., 2013, 87:1884-1889.
[9] Wu J, Fu Z F, Yan F, Ju H X.Tr AC, Trends Anal.Chem., 2007, 26:679-688.
[10] Cravatt B F, Wright A T, Kozarich J W.Annu.Rev.Biochem., 2008, 77:383-414.
[11] Soderstrom C I, Spriggs F P, Song W, Burrell S.J.Immunol.Methods, 2011, 371:106-113.
[12] Xia Z Y, Xing Y, So M K, Koh A L, Sinclair R, Rao J H.Anal.Chem., 2008, 80:8649-8655.
[13] Lathia U S, Olga O, Vladimir B, Mark N.Anal.Biochem., 2010, 398:93-98.
[14] Yan X W, Yang L M, Wang Q Q.Angew.Chem.Int.Ed., 2011, 50:5130-5133.
[15] Percy A J, Parker C E, Borchers C H.Bioanalysis, 2013, 5:2837-2856.
[16] Jeongmin J, Boram L, Taeho L, Kwang-Hyeon L.Rapid Commun.Mass Spectrom., 2015, 28:2405-2414.
[17] Su J, Mrksich M.Angew.Chem.Int.Ed., 2002, 41:4715-4718.
[18] Min D H, Su J, Mrksich M.Angew.Chem.Int.Ed., 2004, 43:5973-5977.
[19] Kuo H Y, De Luca T A, Miller W M, Mrksich M.Anal.Chem., 2013, 85:10635-10642.
[20] Ban L, Mrksich M.Angew.Chem.Int.Ed., 2008, 47:3396-3399.
[21] Kim J, Mrksich M.Nucleic Acids Res., 2010, 38:e2.
[22] Choi I, Kim D E, Ahn J H, Yeo W S.Colloids Surf.B, 2015, 136:465-469.
[23] Hu J J, Liu F, Ju H X.Anal.Chem., 2015, 87:4409-4414.
[24] Sanchez-Ruiz A, Serna S, Ruiz N, Martin-Lomas M, Reichardt N C.Angew.Chem.Int.Ed., 2011, 50:1801-1804.
[25] Beloqui A, Sanchez-Ruiz A, Martin-Lomas M, Reichardt N C.Chem.Commun., 2012, 48:1701-1703.
[26] Beloqui A, Calvo J, Serna S, Yan S, Wilson I B, Martin-Lomas M, Reichardt N C.Angew.Chem.Int.Ed., 2013, 52:7477-7481.
[27] Hu J J, Liu F, Ju H X.Angew.Chem.Int.Ed., 2016, 55:6667-6670.
[28] Ritorto M S, Ewan R, Perez-Oliva A B, Knebel A, Buhrlage S J, Wightman M, Kelly S M, Wood N T, Virdee S, Gray N S, Morrice N A, Alessi D R, Trost M.Nat.Commun., 2014, 5:4763.
[29] Yang H, Chan A L, La Vallo V, Cheng Q.ACS Appl.Mater.Interfaces, 2016, 8:2872-2878.
[30] Lv S, Luo Q, Li X C, Wu J H, Liu J N, Xiong S X, Feng Y Q, Wang F Y.Analyst, 2010, 135:2858-2863.
[31] Deng Z Z, Ye M L, Bian Y Y, Liu Z Y, Liu F J, Wang C L, Qin H Q, Zou H F.Chem.Commun., 2014, 50:13960-13962.
[32] Gregorius B, Jakoby T, Schaumloffel D, Tholey A.Anal.Chem., 2013, 85:5184-5190.
[33] Ling L, Xiao C S, Wang S, Guo L M, Guo X H.Talanta, 2019, 200:236-241.
[34] Wang S, Xiao C S, Guo L M, Ling L, Li M, Li H M, Guo X H.Talanta, 2019, 205:120141.