摘 要: 第二大类CRISPR-Cas系统具备多重基因编辑修饰功能,仅由单效应蛋白介导完成干扰过程,包括靶向编辑双链DNA的Cas9、Cas12系统,及识别并切割单链RNA的Cas13系统.综合介绍第二大类CRISPR-Cas系统的结构与功能基础、作用机制,及其在构建肿瘤模型、肿瘤免疫治疗、耐药性研究、核酸检测等方面的应用,为进一步开发CRISPR-Cas工具并应用于临床医学研究提供参考.
关键词 : CRISPR-Cas; 肿瘤模型构建;耐药性研究;肿瘤免疫治疗;核酸检测;
Abstract: The Class 2 CRISPR-Cas system has the function of multiple gene editing and modification.This system completes the interference progress by single effector proteins,including Cas9 and Cas12 systems for editing the double-stranded DNA,and Cas13 system for recognizing and cutting single-stranded RNA.In this article,systematically summarize the Class 2 CRISPR-Cas system's structural and functional characteristics,the interference mechanism,and the application in tumor model construction,drug resistance research,tumor immunotherapy,nucleic acid detection,etc.Thus,this article will further provide important reference information for developing new CRISPR-Cas tools in clinical research.
Keyword: CRISPR-Cas; tumor model construction; drug resistance research; tumor immunotherapy; nucleic acid detection;
肿瘤有良性肿瘤与恶性肿瘤之分.恶性肿瘤细胞生长迅猛,在体内容易发生转移,且存在医治后再次复发的风险,严重危害人体健康,甚至导致死亡.肿瘤发生及发展过程复杂,多方面的影响相互交织,如家族生理遗传、生活环境诱变、机体自身免疫系统失衡等,这些影响导致机体内的个别细胞产生变异,进而逃脱免疫监视快速大量增殖形成肿瘤细胞;与此同时,肿瘤细胞产生不同方向的变异,使同一肿瘤内或不同肿瘤间存在异质性,表现为生长速度、转移能力、耐药程度等各方面出现不同程度差别,大大增加肿瘤治疗的难度[1].基因编辑技术可人为对细胞内基因的表达进行控制,构建体内外肿瘤模型,探究肿瘤发病分子机制,经由修复肿瘤细胞基因突变或沉默特定蛋白表达实现有效的肿瘤治疗.
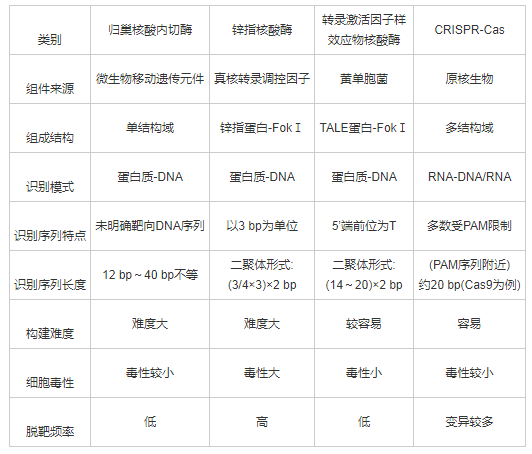
基因编辑技术(site-specific genome editing)指在基因组水平上对特异的基因序列进行增添、敲除及替换等操作的技术,特点为精确、定点、可修饰,产生的双链DNA缺口(double-strand breaks,DSBs)经由体内同源重组(homologous recombination,HR)及非同源重组(non-homologous ending-joining,NHEJ)相关途径完成修复.同源重组途径多发生于同源序列存在的情形[2],损伤的DNA序列修复严格遵守碱基互补配对原则,细胞进行精确的定点修复;而其他情况下细胞更偏向于通过非同源末端连接途径进行DNA双链断裂修复,但这种方式引入的错配较多,易出现损伤位点处碱基的插入或删除,造成基因突变或基因敲除[3].通过探索及开发靶向特定位置DNA缺口的技术方法,可进一步提高基因编辑效率,实现更精准的基因编辑.现有的一些基因编辑技术包括早期的归巢核酸内切酶(homing endonucleases,HEs)、锌指核酸酶(zinc finger nucleases,ZFNs)、转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALENs)、以及CRISPR-Cas系统(clustered regularly interspaced short palindromic repeats and associated protein)等,各类技术的优势与不足之处总结归纳[4],见表1.CRISPR-Cas系统凭借可编辑范围广、编辑效率高、操作简单便捷,同时所需花费时间及成本低等优势,迅速在肿瘤治疗领域得到发展.
表1 4种基因组编辑技术的比较
回顾CRISPR-Cas始源,20世纪80年代末Ishino等[5]进行大肠杆菌K12菌株基因组测序相关研究时,发现一类特殊的重复-间隔-重复序列位于碱性磷酸酶同工转化酶基因(iap gene,iap基因)附近,且该序列不与任何已报道的原核生物序列存在同源关系.随后Lander等[6]报道在其他原核生物(包括细菌、古细菌等)的测序中,皆存在类似结构的串联短重复间隔序列,后将其统称作CRISPR序列.此外Bolotin等[7]发现这一序列和病毒、噬菌体、质粒基因组遗传元件具有高度的同源性,靠近该序列处存在cas相关基因,可编码相关蛋白破坏外源遗传物质功能使之表达沉默[8].由此认识到CRISPR-Cas系统是一类细菌及古细菌清除入侵外源遗传物质的适应性免疫防御系统.
本文将以目前研究较多的第二大类CRISPR-Cas系统作为详例,对其分类、结构、机制及肿瘤研究方面的应用进行介绍.
1、 CRISPR-Cas系统的分类
早期CRISPR-Cas系统基于物种基因组来源进行分类[9],然而,这种分类方式带有进化关系不明且容易混淆等缺点,因而之后统一由Cas蛋白元件的特征及进化关系作为基准,分为第一大类和第二大类[10,11,12],见图1.
图1 CRISPR-Cas系统分类
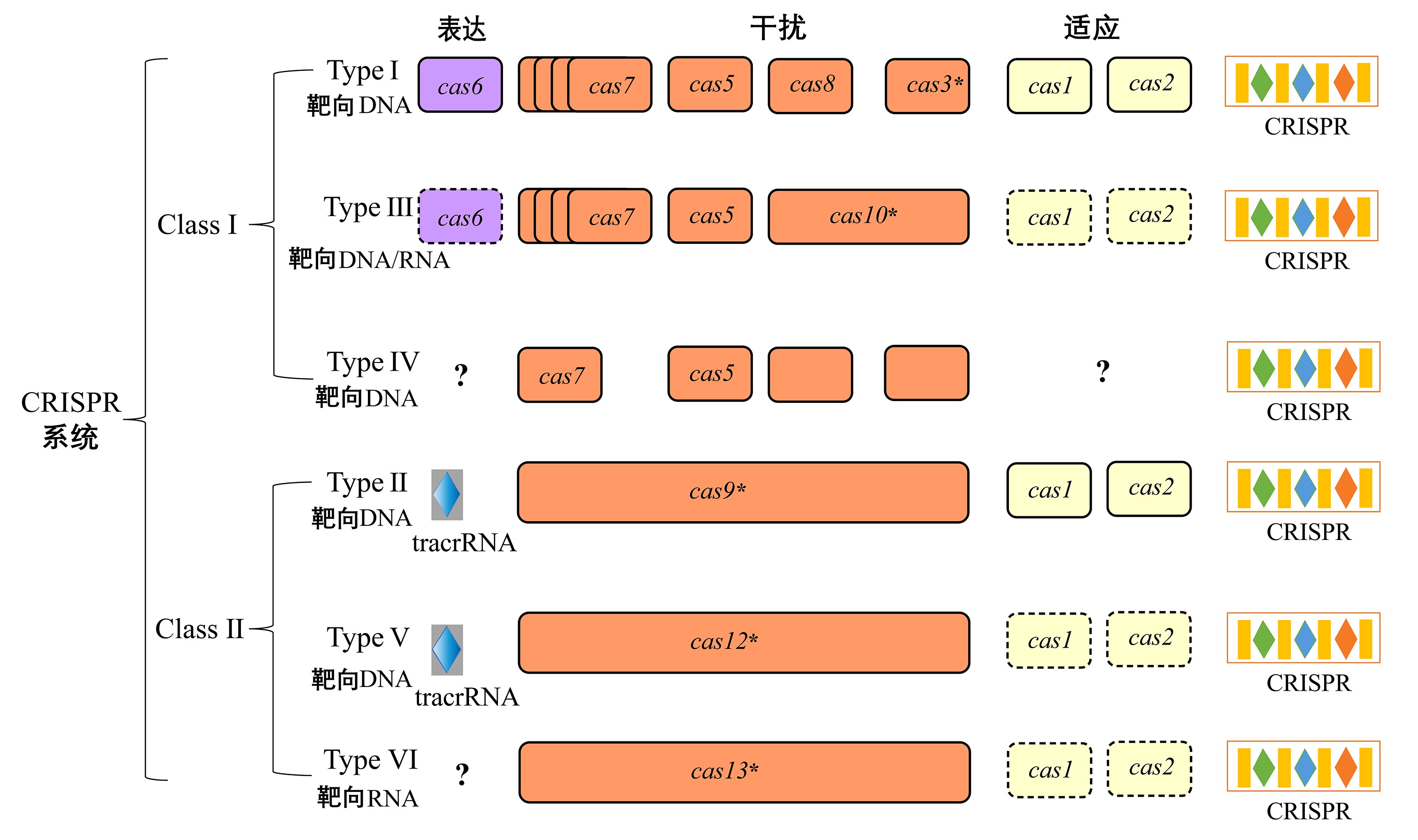
Fig.1 The classification of CRISPR-Cas system
第一大类CRISPR-Cas系统由多个Cas效应蛋白复合物共同作用发挥功能,分成Ⅰ型、Ⅲ型、Ⅳ型.其中Ⅰ型系统的基因座中皆含cas3或cas3突变体特征的基因,编码具有解旋酶及DNA核酸酶活性的蛋白[13],且含共同基因cas1及cas2;Ⅲ型系统以cas10基因作为特征基因,能编码蛋白将ATP向环状腺苷酸产物转化[14];而Ⅳ型系统目前被鉴定出A、B、C等3个亚型,其特别之处在于缺少cas1基因及cas2基因,且与CRISPR序列通常不相邻[15].
第二大类CRISPR-Cas系统的显着优势在于仅由单个效应蛋白即可完成整个干扰过程,分为Ⅱ型、Ⅴ型、Ⅵ型系统.Ⅱ型系统是目前研究最深入、应用最多的一类CRISPR-Cas系统,以cas9基因作为标志基因,且含cas1和cas2共同基因;Ⅴ型系统依靠自身效应蛋白即可对前体CRISPR crRNA(precursor crRNA,pre-crRNA)进行加工,其具备与Ⅱ型系统不同的靶标特异性,进一步拓宽基因编辑的选择范围;Ⅵ型系统是一类最新报道的RNA编辑工具,在成熟的crRNA介导下行使核糖核酸酶(RNase)活性功能[16].不同CRISPR-Cas系统的效应蛋白具备多元化特征,在结构与功能方面表现出明显的差异,见图2、图3.
图2 Cas9、Cas12a和Cas13a的结构域示意图
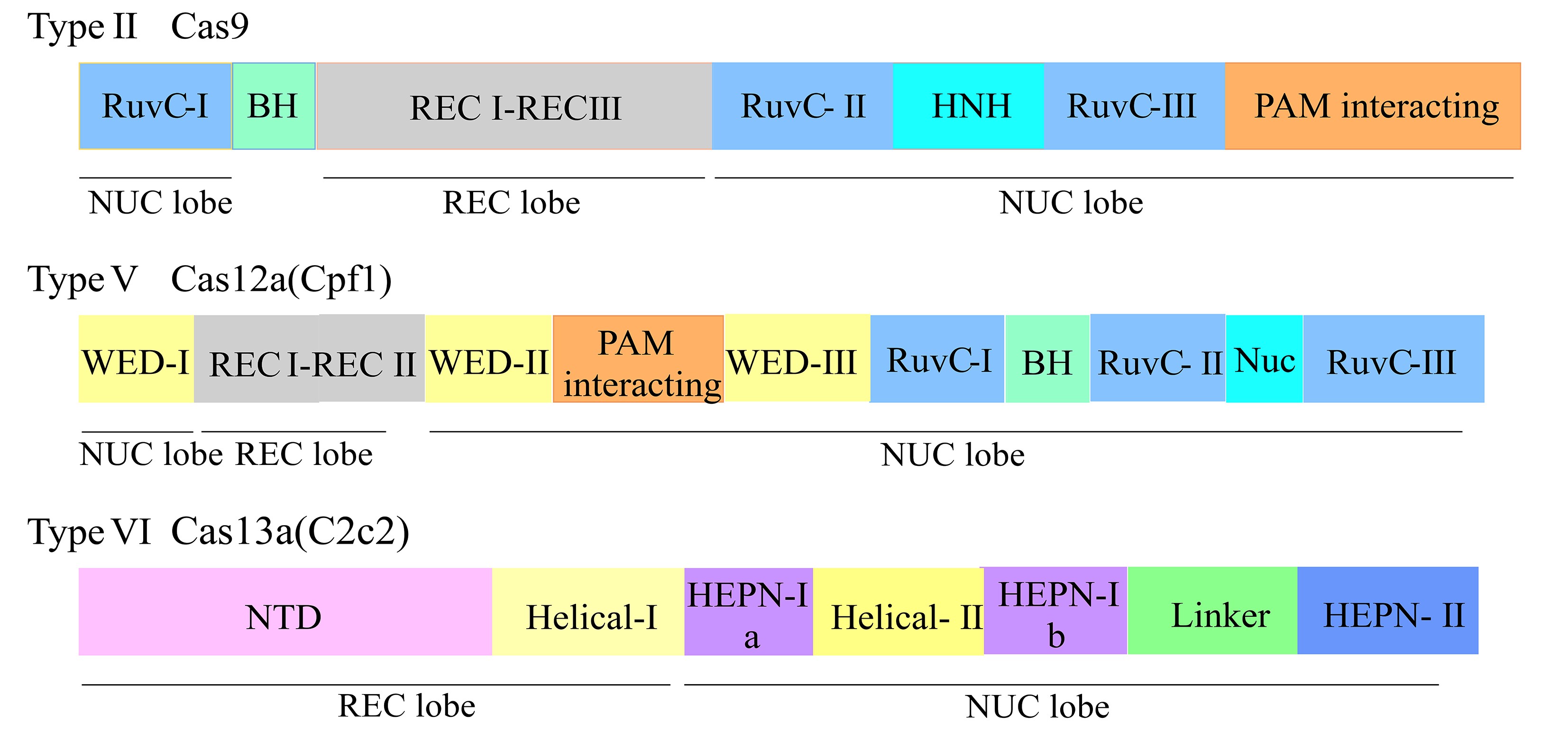
Fig.2 The domain organization of Cas9,Cas12a and Cas13a
BH:富含精氨酸的桥螺旋;REC:识别叶;NUC:核酸酶叶;PAM interacting:PAM作用结构域; WED:楔形结构域;NTD:N端结构域;HEPN:高等真核生物和原核生物的核苷酸结合结构域
1.1 、Ⅱ型CRISPR-Cas9系统效应蛋白的结构与功能
Ⅱ型CRISPR-Cas9系统分为负责识别靶DNA作用的REC叶(recognition lobe),以及负责切割靶DNA功能的NUC叶(nuclease lobe),其中REC叶含REC-1、REC-2以及单个的长α螺旋域,NUC叶含RuvC、HNH和PI结构域(PAM interacting domain)[17].CRISPR-Cas9行使切割靶双链DNA功能时需满足crRNA、tracrRNA(trans-activating crRNA)以及Cas9蛋白3方同时存在的条件,由tracrRNA与crRNA一齐组成特殊的双RNA结构,该结构可与Cas9效应蛋白组合形成复合体,介导Cas9蛋白对靶dsDNA切割,在后续研究中也有报道将该系统进行简并优化,设计为单一的sgRNA(single-guide RNA)以发挥功能[18].识别过程中PAM序列(protospacer adjacent motif,原间隔序列)先被扫描,正确识别后靶DNA同sgRNA发生互补配对;在REC叶与NUC叶间一处带大量正电荷的凹界面,由NUC叶的RuvC域以及HNH域分别靶向切割dsDNA的非互补链及互补链,产生平末端切口.
图3 SpCas9,AsCas12a和LbuCas13a的三元复合物晶体结构

Fig.3 The crystal structures of SpCas9,AsCas12a and LbuCas13a ternary complexes
核酸部分红色表示cRNA,粉色表示靶标核酸链
1.2、 Ⅴ型CRISPR-Cas12系统效应蛋白的结构组成与功能
Ⅴ型CRISPR-Cas系统是一类种类极其丰富的防御系统,其中针对Cas12a的研究及应用更为广泛.Cas12a(也称Cpf1)与Cas9同样呈双叶结构[19],仅含单一的RuvC域,却兼备DNase活性及RNase活性.通过PAM(序列中含丰富的胸腺嘧啶T)对靶DNA目的序列进行识别[20],随后crRNA与靶DNA序列发生碱基配对相互结合.Cas12a发挥酶活的主要部位为RuvC核酸内切酶结构域及特有的Nuc(alkaline nuclease)结构域,2个结构域各自负责切割靶DNA片段的非互补DNA链与互补DNA链,产生具有黏性末端的DSB,同平末端相比更有利于精确整合靶DNA序列.Cas12a发挥其功能只需单个短crRNA(约42nt)介导,不论在长度或结构的复杂程度,皆比Cas9系统所需的tracrRNA-crRNA双RNA结构更有优势,此外Cas12a还具有更低的细胞毒害副作用及脱靶效应,更加适于长序列DNA片段的基因编辑.
1.3、 Ⅵ型CRISPR-Cas13系统效应蛋白的结构与组成与功能
Ⅵ型Cas13系统为第二大类CRISPR系统中唯一一类以ssRNA(single-stranded RNA)作为靶标的基因编辑系统,各效应蛋白除序列中2个HEPN结构域(higher eukaryotes and prokaryotes nucleotide-binding)外无任何同源关系.Cas13的结构与Cas9或Cpf1相似,分为REC叶和NUC叶2个叶片状结构,但其REC叶包含Helical-1结构域以及相对保守性较高的N端结构域(N-terminal domain,NTD).值得注意的是,Cas13a的Helical-1结构域负责加工前体crRNA成熟,Cas13b则由RRI-2(the repeat region interacting domain-2)结构域负责,而Cas13d的HEPN-2结构域在加工前体crRNA过程起关键作用[21,22,23,24],见图4;NUC叶由Helical-2结构域、2个HEPN结构域,以及1个Linker结构域组成.2个HEPN结构域为Cas13a效应蛋白发挥活性的部位,行使切割功能时多受靶RNA序列两侧的PFS(protospacer flanking sequence)序列限制且需二价的金属离子介导[25],一旦酶活被激发,可特异切割靶单链RNA,同时非特异性地切割效应蛋白周边环境任意单链RNA.在Cas13b系统中存在编码小辅助蛋白Csx27及Csx28表达的相关基因[26],对RNA的干扰活性起抑制或加强作用.Cas13d效应蛋白是目前已报道最小的Cas13效应蛋白,其含有1个新的Cas辅助蛋白家族——WYL(consecutive tryptophan-tyrosine-leucine sequence motif)结构域,该结构域负责调控不同种属Cas13d系统对RNA底物切割活性[27].
图4 Ⅵ型Cas13b、Cas13d负责加工前体crRNA的部位

Fig.4 The pre-crRNA processing site of type Ⅵ Cas13b and Cas13d
核酸部分红色表示cRNA;利用红色方框标注加工部位,BzCas13b中负责加工cRNA的结构域为RRI域, a图中以粉色表示;UrCas13b中负责加工cRNA的结构域为HEPN-2域,b图中以淡蓝色表示
2 、CRISPR-Cas系统的作用机理
CRISPR-Cas系统经由3个阶段行使功能清除异己,见图5.第一阶段为外源遗传物质的获取,宿主通过CRISPR-Cas系统获取一部分外源序列整合到CRISPR基因组中形成间隔序列(spacer sequence),同时伴随新的重复序列(repeat sequence)复制,在靠近CRISPR序列的5’端形成重复-间隔-重复单元[28].该过程需要Cas1和Cas2形成Cas1-Cas2复合物参与识别并剪切下合适大小的间隔序列[29],而在部分CRISPR-Cas系统中不存在Cas1和Cas2蛋白,如Cas13b系统,其可能经由同一基因组中其他位点获取适应模块,或是不再积极地获取新的前间隔序列(protospacers),丢失适应模块;第二阶段为crRNA表达及加工成熟,单个的重复序列及间隔序列被一齐转录为前体crRNA,经剪切作用后变为成熟crRNA[30].Ⅱ型通过tracrRNA与前体crRNA相互识别,经由宿主RNaseⅢ以及Cas9对前体crRNA两端进行加工[31],而Ⅴ型、Ⅵ型系统利用自身Cas12、Cas13效应蛋白即可对前体crRNA进行加工,无需tracrRNA参与;第三阶段为外源DNA或RNA的干扰,加工成熟的crRNA与表达的Cas效应蛋白形成复合物,识别再次入侵的外源遗传序列,该过程有时还需要tracrRNA的参与.在外源核酸序列的5’端或者3’端附近存在宿主基因组不具有的几个保守核苷酸序列,即PAM序列或PFS序列,使得宿主CRISPR-Cas系统区分敌我,以防错误的自剪切发生[32].一旦识别到这些序列,Cas效应蛋白在crRNA的指导下对碱基互补配对的靶核酸序列进行切割,清除外源侵入的DNA或RNA.
图5 第二大类CRISPR-Cas系统干扰机制
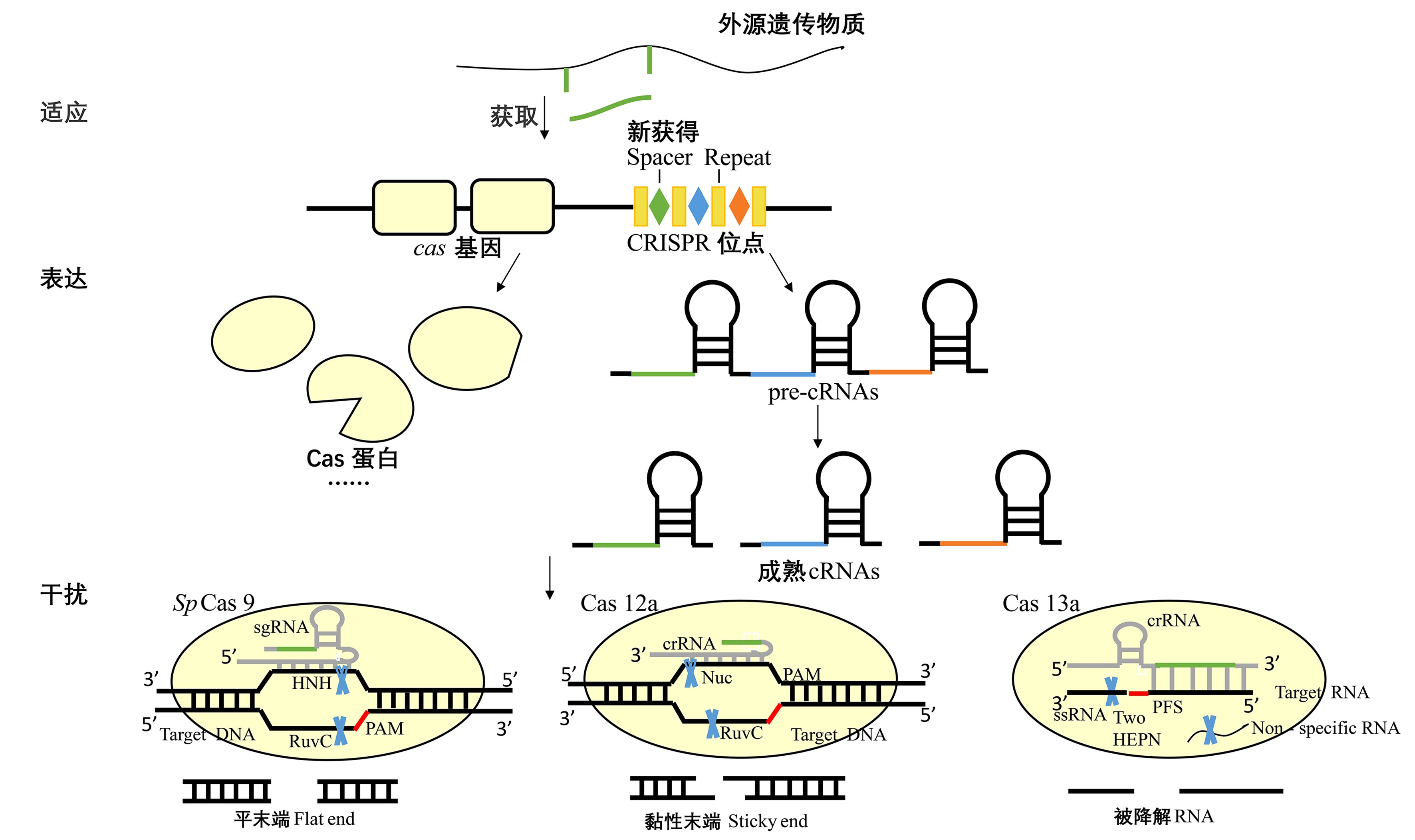
Fig.5 The interference mechanism of Class 2 CRISPR-Cas Systems
3、 CRISPR-Cas系统在肿瘤研究中的应用
3.1 、构建基因敲除肿瘤模型
早期构建肿瘤模型的方法有物理化学手段诱导、手术移植以及基因工程等,但大多耗时久,操作过程复杂,且存在物种限制等问题[33].应用CRISPR-Cas系统模拟体内环境下肿瘤细胞生长环境,获得功能丧失型或功能获得型疾病模型,在此基础上探究肿瘤发病相关肿瘤基因功能,并检验靶向治疗药物效果.2014年Xue等[34]最早报道利用CRISPR-Cas9系统构建肝癌小鼠模型的方法,通过高压水流注射技术从小鼠尾静脉注射表达CRISPR-Cas9系统所需相关的质粒至小鼠肝脏部位,靶向敲除Pten及p53这2个抑癌基因片段,仅短短3个月便形成肝肿瘤,大大节约了肿瘤模型动物培育时间及研究进程.随后,Zhang等[35]向神经元、免疫细胞、内皮细胞等3处导入sgRNA,通过CRISPR-Cas9系统对肺腺癌具最突出影响的KRAS、p53、LKB1这3个基因进行敲除,获得与肺腺癌相同病理特征的小鼠肺癌模型,实现人为对多个特定组织基因的同时敲除,进一步扩展肿瘤模型中基因编辑的范围.乳腺癌在女性患者中较为常见,由于其无确切的治疗靶点且致死率极高,长期临床治疗的效果不理想,三阴性乳腺癌(triple negative breast cancer,TNBC)是其中侵袭能力极强的一类亚型.2019年Guo等[36]首次成功借助CRISPR基因编辑技术破坏并敲除小分子脂质运载蛋白Lipocalin-2(LCN2),构建出三阴性乳腺癌小鼠模型,该技术的特别之处在于能够选择性识别癌细胞阻止体内TNBC肿瘤的扩散,且不会对正常组织产生毒性.更进一步,2020年首次实现在活体动物体内乳腺癌及胶质母细胞瘤的治疗,Rosenbluml等[37]研发出脂质纳米颗粒(LNP)材料的CRISPR-Cas新型递送系统,携带编码Cas9的信使RNA进入体内,由Cas9效应蛋白基因操纵专门对癌细胞DNA产生破坏清除,开辟针对体内癌症治疗与研究的新途径.
3.2、 进行肿瘤免疫治疗
目前临床肿瘤医治方案有手术治疗、放疗、化疗、中医药治疗以及肿瘤免疫治疗等,手术治疗是临床最有效的技术手段,但当患者处于癌症晚期身体机能过差或者肿瘤发病部位不易切除等情况下,患者将无法进行手术失去治疗机会;放疗以及化疗等方法在杀死肿瘤细胞的同时也存在无特异性、无选择性等特点,导致机体出现多种不良反应[38];中医药疗法作为辅助的技术手段,可帮助患者缓解临床症状,减轻病痛,但其无法根治肿瘤;综合上述几类肿瘤免疫治疗手段的优劣势,新的肿瘤治疗方案——肿瘤免疫治疗受到更多人的注目.肿瘤免疫治疗根据肿瘤免疫监视理论,提高效应蛋白杀伤力,阻断或降低免疫细胞的防御,达到控制并清除肿瘤细胞的目的.2017年Eyquem等[39]应用CRISPR-Cas9系统精准定位,向体内插入CAR基因,进一步加强小鼠CAR-T细胞对肿瘤的抑制效用.而这种肿瘤治疗方案对人体是否存在安全隐患,成为大众关心的首要内容.2020年Lu等[40]用CRISPR-Cas9系统介导编辑免疫检查点基因首次在人体内细胞进行试验,初步证实CRISPR-Cas系统编辑T细胞在1个晚期肺癌患者队列中具备较高的安全性与可行性,然而,考虑到该研究中使用技术存在一定局限性,今后试验中仍需进一步优化方案以提高治疗有效性.除此之外新型CRISPR-Cas13a系统也被用于新型高灵敏生物传感器开发,2020年He等[41]报道借助Aptamer-RPA-TMA-Cas13a(ARTCA)的新方法直接对血清外泌体PD-L1含量进行检测,进而对患者肿瘤组织PD-L1蛋白表达水平评估,实现动态监测免疫治疗过程中肿瘤变化情况,以便及时采取有针对性的治疗手段.CRISPR-Cas系统在肿瘤免疫疗法中的应用大大降低肺癌、胃癌、黑色素癌等癌症治疗的技术难度,为肿瘤治疗提供新思路与新手段.
3.3 、研究肿瘤耐药性
肿瘤治疗过程中一旦出现对化疗药物失去敏感性,发生原药耐药及多药耐药反应,临床化疗效果将大打折扣.对此,尝试将CRISPR-Cas9系统应用到肿瘤耐药研究中,根据肿瘤特征有针对性地设计靶向不同组织的向导RNA,从而对肿瘤耐药基因进行定点精确修饰[42].复发难治性、多发性骨髓瘤(multiple myeloma,MM)属于一类临床治疗极具挑战性的恶性肿瘤,该病复发率较高且在化疗中存在多种药物耐性,2016年Song等[43]发现通过CRISPR-Cas9系统敲除MM细胞中高表达的蛋白酶体泛素化受体Rpn13(regulatory particle non-ATPase protein 13),证实Rpn13对多发性骨髓瘤MM细胞存活具有功能性作用,可将其作为潜在治疗靶点,筛选克服MM细胞耐药性的药物.与此同时CRISPR-Cas系统可用于进行致癌基因、抑癌基因的筛选,2018年,Bialk等[44]借由CRISPR-Cas9系统筛选到肺癌细胞阻止化疗药物发挥功能的关键基因NRF2基因(nuclear factor erythroid 2-related factor),通过CRISPR-Cas9敲除该耐药基因与化疗药物联用,阻止肿瘤进一步生长,并取得长足的成效,有效地克服肿瘤耐药.
3.4、 进行核酸检测
第二大类CRISPR系统效应蛋白及其衍生工具可用于核酸检测,进行癌症和抗生素耐药性,以及病毒感染诊断.近年开发出基于CRISPR-Cas12a的DETECTR诊断系统,用于患者血液、唾液、尿液等体液分析.DETECTR原理为DNA样品经扩增后,激活Cas12a的DNase活性,任意地切割携带荧光报告基团FQ的ssDNA,释放荧光信号指示是否存在特异DNA,其优势在于对临床样本DNA需求量较小,实现快速、简便地即时检测.随后2019年进一步研发出基于CRISPR-Cas12a的新型体外检测技术,首次实现在试管或培养皿中对人体内细胞DNA片段多次编辑,开发个性化癌症治疗的新方法[45].DETECTR主要针对DNA进行诊断,而靶向编辑RNA的Cas13a效应蛋白同样作为核酸快速检测手段开发,Zhang等[46]报道了一类SHERLOCK(specific high-sensitivity enzymatic reporter unlocking)检测系统,该系统由Cas13核酸酶和1种会发荧光的RNA报告基团两个部分组成.当Cas13效应蛋白非特异切割RNA的酶活受到激活时,该RNA报告分子将会被酶切,释放出荧光信号从而被检测,通过该监测系统可以鉴定游离于DNA中的低频率癌症突变.当然,对Cas13家族发挥功能时的某些机制仍未完全了解掌握,阻碍其更复杂及标准化的应用发展.
4、 总结展望
自2012年CRISPR-Cas系统作为基因编辑工具应用以来,已报道如Cas9、Cas12以及Cas13系统等多种来自第二大类CRISPR-Cas系统,并进一步开发出众多衍生基因编辑工具,CRISPR-Cas系统推动临床肿瘤疾病诊断及治疗,促进一类针对广泛疾病的全新治疗手段发展.与此同时,CRISPR-Cas系统在基因敲除或插入、精确编辑单碱基或多碱基、植物优良品系改良、功能基因的遗传筛选、细胞活体成像等多方面有新突破,极大的促进生物科研领域向前发展.CRISPR-Cas系统同传统的其它基因编辑技术相比具有更加高效、特异、适于高通量等优点,但也一样存在脱靶现象,可能造成安全隐患,这也成为限制CRISPR-Cas系统应用于体内人体试验的主要原因.通过对造成脱靶的相关因素进行探究,PAM序列、sgRNA的结构及长度、细胞类型、染色质结构等因素皆会对CRISPR-Cas系统精准靶向造成不同程度的影响,对此根据结构研究提出相对应的优化策略,包括对Cas9的PAM序列进行改造;合理设计和修饰sgRNA,提高CRISPR-Cas系统特异性;采用Cas9突变体或Cas9类似物替换等方法,减少非特异性的结合等,推动基于CRISPR-Cas系统的基因治疗更快走向临床应用.基因治疗在基因编辑技术的发展下不断革新,相关的安全意识以及伦理道德教育也应紧步跟上,提高应用CRISPR-Cas系统的安全性,开发出更多更好的CRISPR-Cas工具为人类医疗事业服务.
参考文献
[1]DAVIS A,GAO R,NAVIN N.Tumor evolution:inear,branching,neutral or punctuated?[J]. Biochimica Et Biophysica Acta-Reviews on Cancer,2017, 1867(2):151-161.
[2]URNOV F D,MILLERJ C,LEE Y L,et al.Highly fficient endogenous human gene correction using designed zinc-finger nucleases [J] Nature 2005, 435(7042):646-651.
[3]SALSMAN J,DELLAIRE G Precision genome editing in the CRISPR era [J] Biochemistry and Cell Biology,.2017,95(2):187-201.
[4]吴璐,王磊,任远等基因组编辑技术研究进展[J]生物技术通报,2014(11):184-190.
[5]ISHINO Y,SHINAGAWA H,MAKINO K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion in Escherichia coli,and identification of the gene product [J].Journal of Bacteriology, 1987,169(12):5429-5433.
[6]LANDER E S.The heroes of CRISPR [J]Cell,2016, 164(1/2):18-28.
[7]BOLOTIN A,QUINQUIS B,SOROKIN A,et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin [J]Microbiology,2005, 151(8)-.2551-2561.
[8]JANSEN R,EMBDEN J D,GAASTRA W,et al.ldentification of genes that are associated with DNA repeats in prokaryotes [J].Molecular Microbiology,2002 .43(6): 1565-1575.
[9]HAFT D H,SELENGUT J,MONGODIN E F,et al.A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes [J.PLoS Computational Biology,2005,1(6):60.
[10]MAKAROVA K S,ZHANG F,KOONIN E V.Snapshot:Class 1 CRISPR-Cas systems [J]Cell,2017,168(5):946-946.
[1]MAKAROVA K S,ZHANG F,KOONIN E V.Snapshot:Class 2 CRISPR-Cas systems [J]Cell,2017,168(1/2).328-328.
[12]MOHANRAJU P,MAKAROVA K S,ZETSCHE B,et al.Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems [J] Science. 2016,353(6299):5147.
[13]SINKUNAS T,GASIUNAS G,FREMAUX C,et al.Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system [IJ].EMBO Journal,2011,30(7):1335-1342
[14]KAZLAUSKIENE M,KOSTIUK G,VENCLOVAS,et al.A cyclic oligonucleotide signaling pathway in type皿CRISPR-Cas systems [J].Science,2017,357(6351):605-609.
[15]MAKAROVA K S,WOLF Y I,ALKHNBASHI 0 S,et al.An updated evolutionary classification of CRISPR-Cas systems [J].Nature Reviews:Microbiology,2015, 13(11):722-736.
[16]ABUDAYYEH 0 O,GOOTENBERG J S,KONERMANN S,et al.C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector [J] Science,2016,353(6299):aaf5573.
[17]NISHIMASU H,RAN F A,HSU P D,et al.Crystal structure of Cas9 in complex with guide RNA and target DNA [J]. Cll.2014,156(5):935-949.
[18]JINEK M,CHYLINSKI K,FONFARA I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science,2012,337(6096):816-821.
[19]YAMANO T,NISHIMASU H,ZETSCHE B,et al.Crystal structure of Cpf1 in complex with guide RNA and target DNA [J]Cll,2016,165(4):949-962.
[20]SWARTS D C,VAN DER 00ST J,JINEK M.Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas 12a [J].Molecular Cell,2017.662)-221-233.
[21]LIU L,LIX Y,WANG J Y,et al.Two distant catalytic sites are responsible for C2c2 RNase activities [J]Cell,2017,168(1/2).121-134.
[22]ZHANG B,YE W W,YE Y M,et al.Structural insights into Cas 13b-guided CRISPR RNA maturation and recognition [J].Cell Research,2018,28(12):1198-1201.
[23]KNOTT G J,EAST-SELETSKY A,COFSKY J C,et al.Guide-bound structures of an RNA-targeting A-cleaving CRISPR-Cas13a enzyme [J.Nature Structural & Molecular Biology,2017,24(10)-:825-833.
[24]ZHANG B,YE Y M,YE W W,et al.Two HEPN domains dictate CRISPR RNA maturation and target cleavage in Cas 13d [J.Nature Communication,2019,10(1):2544.
[25]GOOTENBERG J S,ABUDAYYEH 0 O,KELLNER M J,et al.Multiplexed and portable nucleic acid detection platform with Cas13,Cas12a,and Csm6 [J] Science,2018,360(6387):439-444.
[26]SMARGON AA,COX D B T,PYZOCHA N K,et al.Cas13b is a Type VI-B CRISPR-associated RNA-guided RNase difrentially regulated by accessory proteins Csx27 and Csx28 [J]. MolecularCell,2017,65(4):618-630.
[27]YAN W X,CHONG S R,ZHANG H B,et al.Cas13d is a compact RNA-targeting Type VI CRISPR effector positively modulated by a WYL-domain-containing accessory protein [J].Molecular Cel1,.2018,70(2):.327-339.
[28]NUNEZ J K,KRANZUSCH P J,NOESKE J,et al.Cas 1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity [J.Nature Structural & Molecular Biology,2014,21(6):528-534.
[29]YOSEF I,GOREN M G,QIMRON U.Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli [J].Nucleic Acids Research,2012. 40(12):5569-5576.
[30]BROUNS S J,JORE M M,LUNDGREN M,et al.Small CRISPR RNAs guide antiviral defense in prokaryotes [J]. Science,2008,321(5891):960-964.
[31]HATOUM-ASLAN A,MARRAFFINI L A.Mature clustered,regularly interspaced. short palindromic repeats RNA (CrRNA) length is measured by a ruler mechanism anchored at the precursor processing site [J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(52):21218-21222.
[32]DEVEAU H,BARRANGOU R,GARNEAUJ E,et al.Phage response to CRISPR-encoded resistance in Streptococcus thermophilus[J].Journal of Bacteriology,2008,190(4):1390-1400.
[33]DOW L E,LOWE S W.Life in the fast lane:mammalian disease models in the genomics era [J] Cell,2012,148(6):1099- 1109.
[34]XUE W,CHEN S D,YIN H,et al.CRISPR-mediated direct mutation of cancer genes in the mouse liver [J]. Nature,2014,514(7522):380-384.
[35]PLATT R J,CHEN s D,ZHOU Y,et al.CRISPR-Cas9 knockin mice for genome editing and cancer modeling []J]Cell,2014.151(2):440-455.
[36]GUO P,YANG J,HUANG J,et al.Therapeutic genome editing of triple-negative breast tumors using a noncationic and deformable nanolipogel [J].Proceedings of the National Academy of Sciences of the United States of America,2019,116(37):18295-18303.
[37]ROSENBLUM D,GUTKIN A,KEDMI R,et al.CRISPR-Cas9 genome editing using targeted lipid nanoparticles for cancer therapy [J]Science Advances,2020,6(47):eabc9450.
[38]STUPP R,HEGI M E,MASON W P,et al.ffects of radiotherapy with concomitant and adjuvant temozolomide versus radiotherapy alone on survival in glioblastoma in a randomised phase mstudy:5-year analysis of the EORTC-NCIC trial [J].Lancet Oncology,2009, 10(5):459-466.
[39]EYQUEM J,MANSILLA-SOTO J,GIAVRIDIS T,et al.Targeting a CAR to the TRAC locus with CRISPR/Cas9 enhances tumour rejection [J.Nature,2017,543(7643).113-117.
[41]HE Y,WU Y T,WANG Y,et al.Applying CRISPR/Cas13 to construct exosomal PD-L1 ultrasensitive biosensors for dynamic monitoring of tumor progression in immunotherapy [J].Advanced Therapeutics ,2020(3):2000093.
[42]LAWSON K A,SOUSA C M,ZHANG X Y,et al.Functional genomic landscape of cancer-intrinsic evasion of klling by T cells [J]Nature,2020,586(7827): 120-126.
[43]SONG Y,RAY A,LI S,et al. Targeting proteasome ubiquitin receptor Rpn13 in multiple myeloma [J].Leukemia,2016,30(9): 1877-1886.
[44]BIALK P,WANG Y C,BANAS K,et al.Functional gene knockout of NRF2 Increases chemosensitivity of human lung cancer A549 cells in vitro and in a xenograft mouse model [J.Molecular Therapy-Oncolytics,2018.11:75-89.
[45]SANSBURY B M,WAGNER A M,NITZAN E,et al.CRISPR-directed in vitro gene editing of plasmid DNA catalyzed by Cpf1 (Cas12a) nuclease and a mammalian cell-free extract [J]. Crispr Journal,2018,1(2):191-202.
[46]GOOTENBERG J S,ABUDAYYEH 0 O,LEE J W,et al.Nucleic acid detection with CRISPR-Cas13a/C2c2 [J]Science ,2017,356(6336):438-442.






