摘 要: 合成生物学的发展使得人们可以根据需求对微生物进行改造,作为“工厂”高效地合成催化所需物质,并通过添加化学诱导物的方式对生命过程进行调控。然而,化学诱导的潜在毒性以及不可逆性等限制其应用。光遗传学技术利用特定波长的光信号实现对细胞生命过程的调控,具有特异性、可逆性、高时空分辨率等特点。近年来,人们对不同来源的光敏蛋白进行改造,开发出各种不同波长、不同效应的光遗传元件用于基因回路的构建,进而实现对细菌蛋白合成、代谢过程的调控。光遗传技术在人与细菌之间搭起了实时的信号沟通桥梁,实现更为精准的物质生产调控:(1)通过光控治疗因子的合成分泌进行药物递送;(2)通过代谢通路的控制提高目的产物的催化效率;(3)通过光诱导控制生物活材料的形成。随着探索的深入,更小体积、更多波长、更高效率的光遗传元件将被开发出来,实现多输入的细菌生命活动调控。
关键词 : 光遗传学;合成生物学;光敏蛋白;工程菌;生物材料;代谢工程;
Abstract: Advances in synthetic biology made it possible to engineer microorganisms to serve as“factories”to synthesize substances efficiently,and regulate cell activity by adding chemical inducers.However,the toxicity and irreversibility of chemical inducers limited their applications.Optogenetics,which uses light signals of specific wavelengths to regulate the process of cell life,has the characteristics of specificity,reversibility and high spatial and temporal resolution.In recent years,people have modified photosensitive proteins from different sources and developed various optogenetic elements with different wavelengths for the construction of gene circuits,and thus realized the regulation of bacterial protein synthesis and metabolism.Optogenetics technology builds a real-time signal communication between human and bacteria,making the production process more precise and controllable:(1) Drug delivery through bacteria synthesizes therapeutic factors controlled by light;(2) Improve the catalytic efficiency of the target product by controlling the metabolic pathway;(3) Control the formation of living biomaterials under light induction.With further exploration,optogenetic elements with smaller size,more wavelengths and higher efficiency will be developed to realize multi-input regulation of bacterial life activities.
Keyword: Optogenetic; Synthetic biology; Photosensitive protein; Engineered bacteria; Biomaterial; Metabolic engineering;
微生物是自然界最为精妙的“生产车间”,它在基因的指导下有条不紊地同时进行着上千个化学反应,合成所需物质以满足自身的生存需求。近年来合成生物学的迅速发展使得人们可以根据需求对微生物的基因回路进行改造[1,2],合成所需物质,从而应用于药物合成、材料制备、食物生产、环境检测等方面。为了更好地干预微生物的生命过程,目前主要通过添加化学诱导物如IPTG[3]、nisin[4]、阿拉伯糖[5]等对其基因回路进行调控,然而化学诱导方式具有潜在毒性、调控延迟性且不可逆,应用场景大大受限。与之相比,通过光的调控便捷、可逆,具有更高的时空分辨率[-7],可以对微生物生命过程进行精准、快速、实时的调控,具有广阔的应用空间。
自2006年光遗传学被首次提出以来,人们从原核生物中筛选出了一系列光遗传元件如As LOV2[8]、C1V1[9]、ChR2[10]等,通过改造将其应用到哺乳动物细胞中[11],进行脑科学[12]、神经系统[13]以及肿瘤治疗[14]等多方面的研究。随着细菌“工厂化”进程的推进,近年来光遗传元件越来越多地被应用于细菌中,使其可以根据光信号做出即时、准确的响应。2017年,Fernandez-Rodriguez等[15]构建了RGB三色调控基因表达系统,为实现细菌生命活动多模块、多方位的调控提供了思路。通过对光敏蛋白的改造以及基因回路的优化,细菌的光控系统将向着更加特异、更加稳定、更加多元的方向发展。目前,光遗传调控的“细菌工厂”在疾病治疗、材料合成等领域得到广泛应用,极大地促进了细菌生命调控以及合成生物学的进展。本文将概述光遗传学的原理、在细菌生命过程调控中的应用以及潜在价值。
1 、光遗传技术
1.1 、光敏蛋白
双眼赋予了人类视觉感受,是人类获取外界信息的最主要来源之一。自然界中,为了更好地适应环境、获取生存资料,部分植物和微生物进化出了“双眼”———光敏蛋白。光遗传学即是利用基因工程技术对光敏蛋白进行改造并导入特定细胞中,通过光照实现细胞生命活动精准调控的一门技术。
在现阶段研究中,参与构成细菌光遗传调控元件的光敏蛋白主要分为光敏色素与LOV蛋白两大类。1997年,Yeh等[16]在Synechocystis sp.PCC 6803中发现了光敏色素蛋白Cph1,Cph1可在红光与远红光的调控下实现可逆磷酸化调节,进而实现信号的转导。作为光敏色素超家族的成员之一(图1),Cph1具有最为典型的蛋白质结构,即与胆色素发色团结合的感光域和负责信号转导的效应域两部分:位于N端的感光域包括PAS(Per/Arnt/Sim)、GAF(c GMP磷酸二酯酶/腺苷酸环化酶/Fhl A)和PHY三个结构域[17],其中GAF域可与发色团共价结合;C端的效应域为一个组胺酸激酶域(HKD),负责信号转导[18]。除此之外,还有以GAF-PHY结构为感光域的光敏色素蛋白,如Cph2、Rfp A等[19]。蓝细菌光敏色素(cyanobacteriochromes,CBCRs)[20],具有更为简单的感光域,仅由GAF域构成,并可以感受紫外到近红外的所有光谱,因而在感光蛋白的开发中备受关注。1999年,人们通过基因序列分析发现非光和细菌D.radiodurans和P.aeruginosa中也存在着类似光敏色素的感受器[21],并将其命名为细菌光敏色素(bacteriophytochrome photoreceptor,Bph P)。两种细菌分别编码的Dr Bph P和Pa Bph P蛋白通过与发色团BV[22]结合实现失活态(Pr,最大吸收峰约700 nm)与激活态(Pfr,最大吸收峰约750 nm)之间的互相转化[23],是长波长光遗传元件的开发基础。
图1 细菌光敏色素家族成员及其结构域
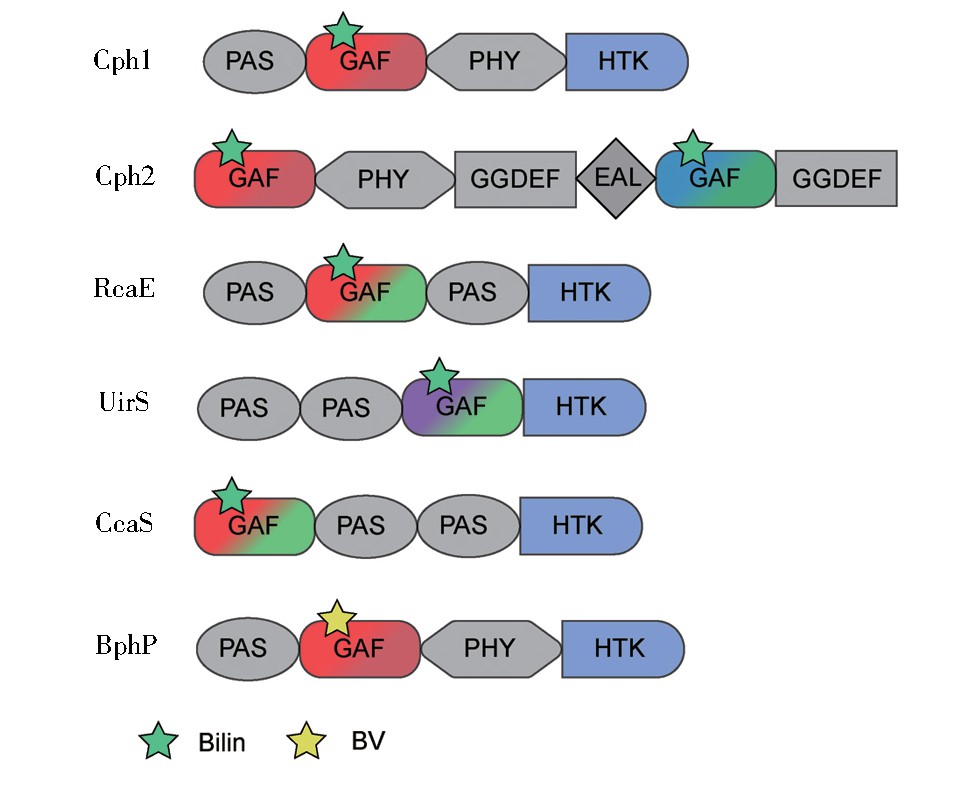
Fig.1 Domains of phytochrome superfamily members
2002年,Losi等[24]在枯草芽孢杆菌中发现了具有光、氧、电压(LOV)结构域的Ytv A蛋白,与高等植物中向光性相关的蓝光感光细胞中向光素的LOV结构域高度同源,均可以与黄素单核苷酸(FMN)结合。LOV属于PAS结构域超家族中的一员,通过与不同的效应域如HK (histidine kinases)、GGDEF-EAL、HTH (helixturn-helix)等结合将光信号进行转导调控生命过程[25]。在蓝光光照条件下,FMN与LOV中的半胱氨酸共价结合,引起结构域的构象发生改变,这是LOV作为光遗传工具发挥光信号转导功能的基础。与光敏色素所需的胆素发色团相比,LOV蛋白所需的辅因子FMN广泛存在于各种细胞中,在设计光遗传元件过程中无须额外合成,因此基于LOV的光遗传工具也有着更广阔的发展前景。
1.2、 光控基因表达系统
与传统的化学诱导方式相比,光具有更低的毒性、可逆性、高时空分辨率等优势,通过合成生物学手段构建光控基因表达系统可以实现光控细菌生命活动,“on”和“off”的即时切换在细菌代谢、蛋白质合成等领域具有广阔的应用前景。
根据系统结构划分,现阶段开发的光控基因表达系统主要分为单组分系统与双组分系统两大类,其中最早出现的双组分系统主要由光敏组件与基因调节组件两部分组成。2005年,Levskaya等[26]在大肠杆菌中首先构建了基于光敏蛋白Cph1的光控基因表达系统Cph8。通过外源引入的藻青素合成酶pcy A和ho1酶将血红素代谢为藻青素(phycocyanobilin,PCB)作为发色团,在黑暗条件下,Cph8磷酸化并将磷酸基团转移至OmpR,启动Omp C启动子下游基因转录翻译;在红光条件下磷酸化被抑制,基因表达终止(图2a)。与之相似的还有基于CBCRs的光控系统,Cca S/CcaR系统[27]在535 nm绿光刺激下启动下游基因表达,在672 nm红光下表达抑制Cca S;Uir S/UirR系统[28]则实现了紫外线启动基因表达,535 nm绿光抑制基因表达。基于Bph Ps的光控转录系统在近红外光照射下,Bph P1被激活并通过结合转录抑制因子PpsR2来激活下游基因的转录(图2b),无须磷酸基团的信号转导过程,因而具有更加快速的反应速率[29]。
多波长、多机制的光控基因表达系统为不同条件、不同需求的细菌控制提供了选择,然而在实际生产中常用的工程细菌底盘均为非光敏细菌,自身无法合成发色团[22],需要引入外源酶基因,而在细菌改造过程中可以接受的外源基因载体总量有限,酶的引入将减少目的基因的插入空间。2006年,M9glich等[30]构建的基于Ytv A-LOV蛋白的光控系统YF1/Fix J则解决了这一问题(图2c)。该系统将慢生根瘤菌中负责蛋白质表达调控的Fix L系统中的氧敏感受器替换为LOV蓝光光敏蛋白,并将新的融合蛋白命名为YF1;在黑暗条件下,YF1磷酸化其同源反应调节因子Fix J,进而驱动Fix K2启动子下游基因的表达,在蓝光条件下这一过程被抑制[31]。
图2 双组分系统构成及其工作原理示意图
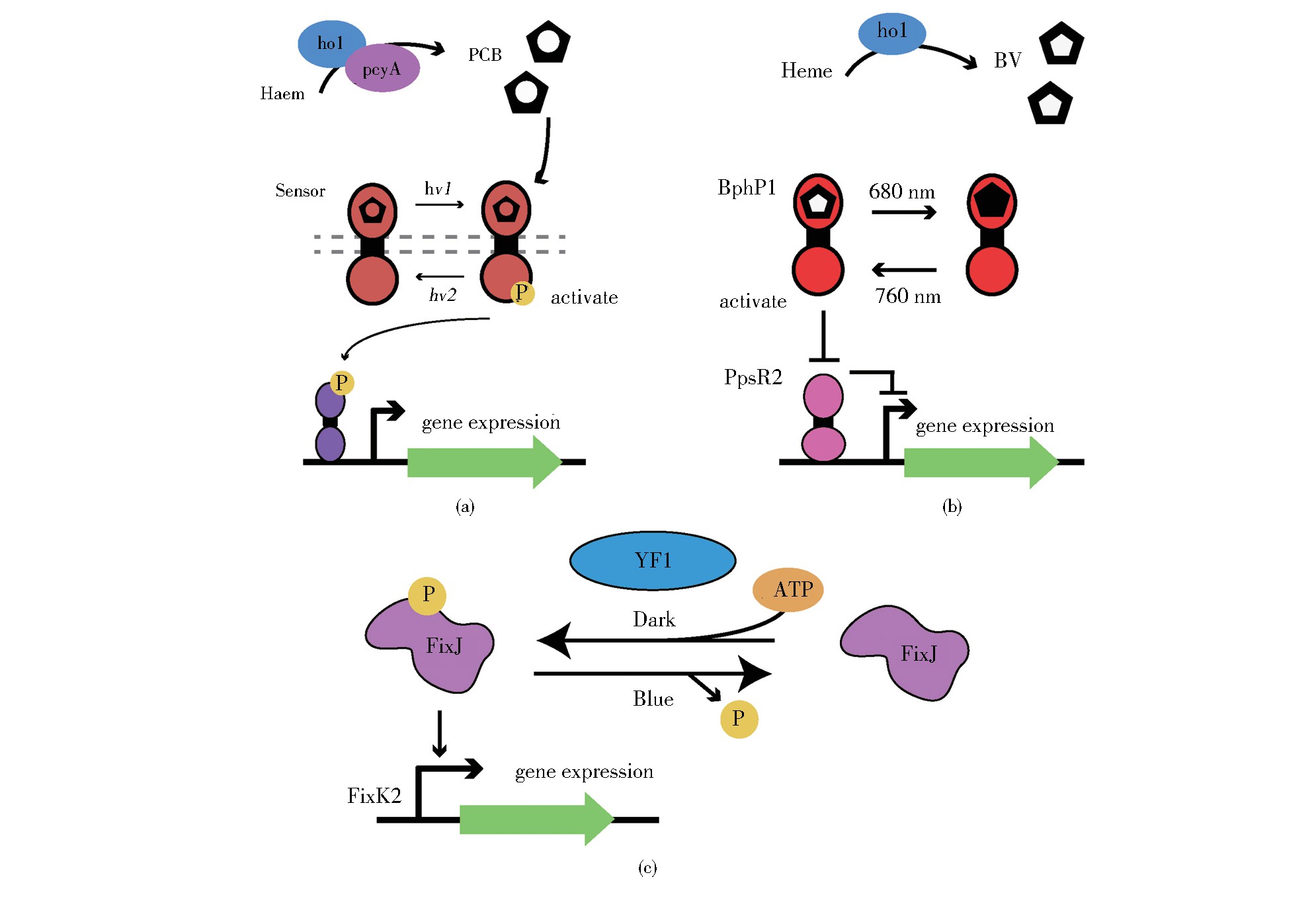
Fig.2 Schematic of two-component systems
(a) Two-component system based on phytochrome needs exogenous enzyme that convert haem into PCB,initiates gene expression in response to light stimulated phosphorylation (b) BphP based system use BV as chromophore,gene expression is activated by interaction between BphP1 and PpsR2(c) YF1 phosphorylates FIXJ under dark conditions and regulates gene expression
与双组分系统相比,单组分光控基因表达系统无须繁琐的信号转导过程,元件更小,更易移植,响应速度更快。到目前为止,开发出的单组分系统均基于LOV家族蛋白,通过将LOV域与不同驱动蛋白相连实现光控基因转录。EL222是源自于海洋细菌Erythrobacter litoralis HTCC2594的光控转录因子,负责感光的LOV结构域与HTH DNA结合域相连[32]。在黑暗条件下,HTH结构域被LOV封闭;经蓝光照射,LOV域构象发生改变并释放HTH域,蛋白质发生二聚化并结合至特定DNA序列,启动基因的转录。哺乳动物细胞研究表明,EL222光控基因表达系统在蓝光光照下可将基因表达水平上调200以上,且具有快速激活(<10s)和快速失活(<50 s)的特点[33]。
与EL222相类似的还有源自Neurospora crassa中基于LOV结构域的感光蛋白VVD(Vivid)[25],可以响应蓝光形成二聚体,进而控制蛋白质活性,调控基因的转录。2012年,Wang等[34]通过将不同的转录激活结构域融合到VVD的羧基端实现了哺乳动物细胞的光控转录调控。由于VVD在蓝光激发状态下存在同源二聚化的现象,当A、B两种蛋白质需要在VVD作用下结合并发挥作用时,常会出现“A+A”与“B+B”的无效反应,大大降低了反应效率。为此,Kawano等[35]开发出了两种分别带有正电荷与负电荷的VVD变体(p Mag/n Mag),并将其命名为Magnets,在二聚化过程中,两种VVD蛋白可以互相识别,大大提高了信号转导效率。2019年,Sheets等[36]利用VVD构建了首个应用于原核生物的光控重组酶,Opto-Cre-Vvd。Cre作为从P1噬菌体中分离的酪氨酸重组酶可以识别DNA中的lox P位点并进行酶切,通过结构解析将Cre分为两部分(n Cre/c Cre)并融合至VVD蛋白,在蓝光照射下,VVD发生二聚化促使n Cre与c Cre结合,对指定DNA序列进行酶切,激活目的基因的表达。到目前为止,应用于细菌的单组分光控基因表达系统均为蓝光激发,与双组分系统相比光谱成分单一,因此需要开发更多波长的单组分光控系统。
2 、光遗传细菌的应用
随着对微观世界的发掘以及微生物研究的深入,人们发现,细胞内一切催化过程、成键与断键均在基因电路的调控下有条不紊地进行,是最为精密的物质合成工厂。通过合成生物学手段对细菌进行改造,可以使其成为合成所需产物的“细菌工厂”(图3)。在工厂的运作过程中,需要与人之间的信号交流以保证物质合成过程的高效可控,光遗传工具因其高时空分辨率、即时可逆等优势在细菌的生产调控中已有了广泛应用。
图3 光遗传细菌的工程应用
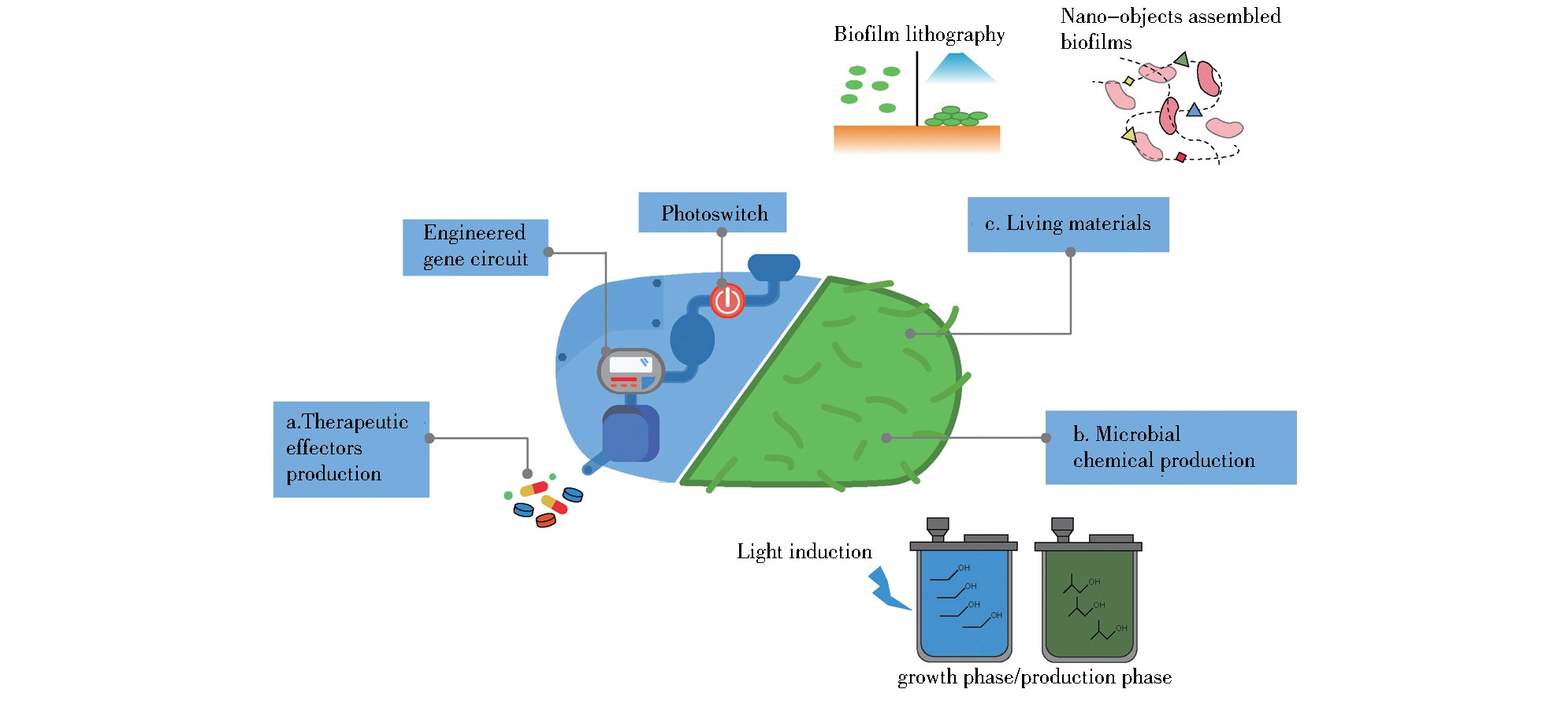
Fig.3 Engineering application of optogenetic bacteria
(a) Light controlled bacterial drug synthesis (b) Light controlled bacterial metabolic process (c) Formation and assembly of light-controlled living biological materials
2.1 、药物合成工厂
通过基因通路的改变和外源基因的引入,人们将细菌改造成为“药物合成工厂”,低成本、高效率地获取治疗药物。光控基因表达系统为细菌合成和代谢途径提供灵活即时的“on/off”开关,从而构建出可控的药物递送系统。2014年,Magaraci等[37]对大肠杆菌进行改造,添加光控原件,分别实现了红光和蓝光调控的细胞毒素Cly A分泌,进而杀伤肿瘤细胞。与传统治疗手段相比,光控细菌给药系统可以更加精确地控制给药剂量,其高时空分辨率的特性也使得治疗过程更加安全可控,有望将“生物活药”推向更多疾病治疗领域。在体外应用领域,Sankaran等[38]将光遗传工程菌与水凝胶材料相结合,实现了药物的长期可控输送。封装于水凝胶中的无内毒素大肠杆菌可以在蓝光调控下分泌抗菌药物紫色杆菌素(deoxyviolacein,d Vio),并可维持至少42天的稳定药物释放,高度契合伤口愈合与皮肤抗菌等应用场景。在后续工作中,这一系统还被应用于蛋白质的可控递送[39],系统中光控元件、分泌物质、细菌底盘与水凝胶基质模块均可根据需求灵活调整,实现个性化治疗。
在体内的原位药物递送研究中,基于合成生物学的下一代微生物疗法通过口服长久生存于肠道中的工程菌被用于肠道炎症治疗[40,41]、炎症检测[42,43,44]、血氨代谢[45]等方面,部分已进入临床研究阶段。在此基础上,光遗传工具的使用将使肠道工程菌的药物递送过程更为可控。为将光信号高效输送至体内发挥调控作用,Yang等[46]构建了上转换光遗传调控系统,利用组织穿透性更好的近红外光,通过体外照射控制肠道内部光响应工程菌的治疗物质分泌,进而抑制肠道炎症或肿瘤的进展。经肠道的工程菌光控药物递送系统避免了药物注射的痛苦以及口服给药的药物损失,且实时可控,随着更加便捷、安全的光照系统的开发,这一给药方式将具有更多的应用场景。
2.2、 催化代谢工厂
微生物发酵被广泛应用于医药工业、食品工业、能源工业、化学工业等多个领域,随着合成生物学的发展,人们通过基因改造将微生物改造成为具有特定功能的“催化代谢工厂”,高效获取目的产物(表1)。在发酵过程中,合理的平衡好菌株生长与物质生产的关系,最优化能量分配是提高生产效率的关键。光遗传技术从工程菌内部基因表达调控出发,为产能的提升提供新思路。在代谢通路的光调控方面,Zhao等[47]改造了EL222光控基因表达系统并应用至酿酒酵母中,利用光照控制代谢酶的表达优化代谢途径,提高目的产物的获取。在蓝光条件下,Opto EXP启动负责丙酮酸脱羧作用的PCD1基因的表达,酿酒酵母进入生长繁殖阶段,代谢产物为乙醇;在黑暗条件下,Opto INVRT启动乙酰乳酸合成酶(Ilv2)的表达,酿酒酵母处于生产阶段,大量合成异丁醇。利用此方法,酿酒酵母可以生产高达(8.49±0.31) g/L的异丁醇和(2.38±0.06) g/L的2-甲基-1-丁醇。与之相似,2019年,Tandar等[48]利用Cca S/CcaR系统通过控制pgi基因的表达来调节大肠杆菌中EMP与ox PP两种糖酵解途径之间的通量分布,在绿光和红光的照射下,二者通量比分别为50∶49和0.5∶99,ox PP代谢途径通量的提高提供了更多的NADPH,进而提高大肠杆菌还原性代谢产物如甲羟戊酸、异丁醇等的产量。除代谢酶的表达调控之外,Ding等[49]通过控制细菌分裂的细胞周期以提高代谢性能。细胞分裂的C期进行DNA复制,D期进行细胞分裂。通过蓝光和近红外分别激活核糖核苷酸还原酶Nrd AB和分裂蛋白Fts ZA的表达,缩短C期和D期,提高细胞的比表面积,并将合成的3-羟基丁酮滴度提高到67.2g/L,与野生型相比提高了39.19%;通过激活核糖核苷酸还原酶Nrd A和分裂蛋白抑制剂Sul A的表达,延长C期和D期,提高细胞的体积,合成的聚乳酸-co-羟基丁酸滴度提高到14.31 g/L,与野生型相比提高了137.71%。随着光遗传代谢通路调控研究的深入,这一技术有望应用于工业生产,通过吸光度监测以及生物传感等手段,实现微生物发酵过程的实时监控及控制。
表1 光遗传用于微生物发酵调控

另外,在传统微生物发酵调控中,人们常采用IPTG诱导物质代谢及重组蛋白生产,与之相比,光诱导具有易调谐、可逆性等优势,是更为理想的诱导手段。2020年,Lalwani等[50]将YF1蓝光感受器与乳糖操纵子结合,在大肠杆菌中构建了一系列光遗传调控的lac操纵子基因电路,并通过正反馈调节显着降低了光泄露效应,在光控发酵中,此系统将甲戊酸酯和异丁醇的产量分别提高了24%和27%。综上所述,在微生物发酵生产中,光遗传技术既可作为“开关”实现产物的实时可控合成,也可作为“调节器”平衡工程菌内部的基因表达关系,大大增强可操控性,将微生物改造成为活体“催化代谢工厂”。
2.3 、生物活材料工厂
作为自然界最为精密的合成工厂,细菌在无机纳米颗粒合成等领域有着广泛应用[51,52],同时由于其本身尺寸较小,约为0.5~5μm,其单体可以作为“像素点”进行生物印刷或3D打印中使用的基质材料[53]。将光遗传工具应用到细菌材料合成的调控中,可以使之成为可编程的智能生物活材料,具有广阔的应用前景。2017年,Chen等[54]利用n Mag与n Mag实现了光控细菌特异性黏附,在蓝光光照下,表达于大肠杆菌表面的p Mag蛋白与基质上的n Mag发生聚合,利用细菌绘制各种图案;Jin等[55]则是利用p Dawn启动子调控表达黏附素Ag43实现“生物膜光刻”技术,在蓝光照射下绘制出分辨率达25μm的图案。
卷曲纤维最早发现于20世纪80年代,并被证明与细菌的黏附及生物膜形成有关[56]。其单体之一Csg A蛋白可在体外自组装成纤维状纳米结构,可以为多种无机材料提供附着位点[57],从而开发出了多种基于Csg A的生物活材料。2018年,Wang等[58]利用蓝光控制大肠杆菌中Csg A的合成,进而在空间上控制生物膜的形成。生物膜可以成功附着在各种形状以及不同材质的材料表面,同时可以为金、量子点等纳米颗粒提供附着位点;Moser等[59]在光控手段上进行研究,构建了可同时由红、绿、蓝三色光调控Csg A生物膜形成的光控基因电路。受贻贝黏着斑蛋白的启发,Wang等[60]在2020年将光控细菌生物膜与仿生羟基磷灰石矿化技术相结合,实现了可控的生物梯度复合材料合成。在蓝光调控下,大肠杆菌表达Cgs A-Mfp3S蛋白并在体外聚集成纤维,随后Mfp3S蛋白沉积溶液中的Ca2+与PO43-并发生矿化,形成的可响应外部环境的生物活材料可用于损伤修复等方面。将光遗传技术应用至细菌生物活材料的合成,可以最大限度地发挥其灵活的空间操控性的优势,随着更多细菌活材料的开发,光遗传技术有望实现不同基质、不同环境、不同形状和大小的材料控制合成。
3 、总结与展望
在合成生物学的迅速发展的推动下,机器逐步生物化,而生物则逐步迈向工程化。实现更加实时精确的细胞生命活动调控一直是合成生物学的研究方向之一。与传统化学诱导方式相比,光控细胞活动有着低毒性、可逆性、高时空分辨率等诸多优势。21世纪以来,人们通过基因工程的手段将植物、光敏/非光敏细菌中发现的感光元件进行优化和改造,构建出各种细菌光遗传工具用于基因表达以及蛋白质活性的控制。双组分光控系统拥有更多的可选择波长,但较大的元件体积和复杂的信号转导过程阻碍了这一生物应用;单组分系统具有响应快、元件体积小的特点,但波长单一,仅可通过蓝光操控。因此,为使细菌光遗传系统能够更好应用到细菌生命活动调控中,开发多波长快速响应的光控系统仍是未来的主要研究方向之一。除此之外,仍需兼顾光毒性以及光泄等问题。作为自然界物质催化合成工厂,光遗传元件的插入可以使工厂更为智能可控。在生物医学领域,光遗传细菌可以实现药物的可控合成与定位定量递送,实现个体化治疗;在生物发酵领域,通过光遗传调控提高有效产物生产效率,实现智能化生产;在生物材料合成领域,光控“生物活材料”概念的引入增加了材料合成的操控性。除此之外,光遗传技术作为外界与细菌进行信号交流的手段,在生物传感以及生物基因电路等领域也有着广阔的应用前景。
参考文献
[1]Way J C.Collins J J,Keasling J D.et al. Integrating biological redesign:where synthetic biology came from and where it needs to go. Cell,2014,157(1):151-161.
[2]Khalil A s,Collins J J. Synthetic biology:applications come of age. Nature Reviews Genetics,2010,11(5):367-379.
[3] Taylor N D,Garruss A S,Moretti R,et al. Engineering an allosteric transcription factor to respond to new ligands. Nature Methods ,2016,13(2)-177-183.
[4]Berlec A , Okrlec K.,Kocjan J,et al. single plasmid systems for inducible dual protein expression and for CRISPR-Cas9/CRISPRi gene regulation in lacticacid bacterium Lactococcus lactis. Scientific Reports ,2018,8:1009.
[5] Vilar J M,Saiz L. DNA looping in gene regulation:from the assembly of macromolecular complexes to the control of transcriptional noise. Current Opinionin Genetics&Development, 2005, 15(2):136-144.
[6]Renicke C,Taxis C. Biophotography:concepts,applications and perspectives. Applied Microbiology and Biotechnology.2016, 100(8):3415-3420.
[7]Shao J W,Wang M Y,Yu G L,et al. Synthetic far-red lightmediated CRISPR-dCas9 device for inducing functional neuronal differentiation. PNAS,2018,115(29):E6722-E6730.
[8] Wu Y I,Frey D,Lungu 0 I,et al. A genetically encoded photoactivatable Rac controls the motility of living cells. Nature,2009,461(7260):104-108.
[9]Kennedy M J,Hughes R M.Peteya L A.et al. Rapid blue-lightmediated induction of protein interactions in living cells. Nature Methods,2010.7(12):973-975.
[10] Ye H F, Fussenegger M. Optogenetic medicine:synthetic therapeutic solutions precision-guided by light. Cold Spring Harbor Perspectives in Medicine,2019,9(9):a034371.
[11] Mansouri M.trittmatter T,Fussenegger M. Light-controlled mammalian cells and their therapeutic applications in synthetic biology. Advanced Science(Weinheim, Baden Wurttemberg,Germany),2019,6(1):1800952.
[12] Gradinaru V, Mogri M, Thompson K R. et al. Optical deconstruction of parkinsonian neural circuitry. Science,2009.324(5925):354-359.
[13]Rost B R,Schneider-Warme F,Schmitz D.et al. Optogenetic tools for subcellular applications in neuroscience. Neuron,2017,96(3):572-603.
[14]Gradinaru V,Zhang F, Ramakrishnan C,et al. Molecular and cellular approaches for diversifying and extending optogenetics Cell.2010,141(1):154-165.
[15] Fernandez-Rodriguez J,Moser F,Song M.et al. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology,2017.13(7):.706-708.
[16] Yeh K C.Wu S H,Murphy J T,et al. A cyanobacterial phytochrome two-component light sensory system. Science, 1997 ,277(5331):.1505-1508.
[17] Wiltbank L B, Kehoe D M. Diverse light responses of cyanobacteria mediated by phytochrome superfamily photoreceptors. Nature Reviews Microbiology.2019,17(1):37-50.
[18]Liu Z D,Zhang J Z,Jin J,et al. Programming bacteria with light-sensors and applications in synthetic biology. Frontiers in Microbiology,2018,9:2692.
[19] Rockwell N C,Lagarias J C. Phytochrome diversification in cyanobacteria and eukaryotic algae. Current Opinion in Plant Biology,2017,37:87-93.
[20] Rockwell N C ,Martin S S,Feoktistova K,et al. Diverse twocysteine photocycles in phytochromes and cyanobacteriochromes PNAS,2011.108(29):11854-11859.
[21] Davis S J. Bacteriophytochromes:phytochrome-like photoreceptors from nonphotosynthetic eubacteria. Science, 1999.286(5449):2517-2520.
[22]Bhoo S H,Davis S J,Walker J,et al. Bacteriophytochromes are photochromic histidine kinases using a biliverdin chromophore. Nature,2001 ,414(6865):776-779.
[23] Giraud E, Zappa S, VuilletL, et al. A new type of bacteriophytochrome Acts in tandem with a classical bacteriophytochrome to control the antennae synthesis in Rhodopseudomonas palustris. Journal of Biological Chemistry,2005,280(37):32389-32397.
[24] Losi A, Polverini E, Quest B, et al. First evidence for phototropin-related blue-light receptors in prokaryotes. Biophysical Journal,2002,82(5):2627-2634.
[25] Herrou J,Crosson s. Function, structure and mechanism of bacterial photosensory LOV proteins. Nature Reviews Microbiology,2011 ,9(10):713-723.
[26] Levskaya A,Chevalier AA,Tabor J J,et al. Engineering Escherichia coli to see light. Nature ,2005.438(7067):441-442.
[27]Tabor J J,Levskaya A,Voigt C A. Multichromatic control of gene expression in Escherichia coli. Journal of Molecular Biology,2011,405(2):315-324.
[28]Ramakrishnan P,Tabor J J. Repurposing Synechocystis PCC6803Uir S-UirR as a UV-violet/green photoreversible transcriptional regulatory toolin E. coli. ACS synthetic Biology,2016,5(7):733-740.
[29]Ong N T,Olson E J,Tabor J J. Engineering an E. coli nearinfrared light sensor. ACS Synthetic Biology,2018,7(1):240-248.
[30] M9gich A, Ayers R A, Moffat K. Design and signaling mechanism of light-regulated histidine kinases. Journal of Molecular Biology,2009,385(5):1433-1444.
[31] Ohlendorf R,Vidavski R R,Eldar A,et al. From dusk till dawn:one-plasmid systems for light-regulated gene expression.Journal of Molecular Biology,2012 ,416(4):534-542.