摘 要: 人类基因组计划(Human Genome Project, HGP)不仅极大地提高了人类对基因组和相关遗传信息的认识水平,而且促进了生命科学研究技术的发展和应用。正是在这样的历史背景下,焦磷酸光化测序技术(pyrosequencing)由瑞典研究人员发明。焦磷酸光化测序技术的基础原理是基于“通过合成测序”原理进行酶促反应的DNA测序方法,通过基于释放焦磷酸盐时的链式反应的可见光检测,即可获得一个特异的检测峰,峰值的高低和相匹配的碱基数成正比。该技术可应用于DNA核苷酸序列和突变的检测、单核苷酸多态性的基因型的鉴定,以及DNA甲基化水平变化的分析等。近年来,随着摄影器材和成像技术的快速发展,这项技术的原理是基于通过酶促反应而实时检测可见光,因此,有望在检测的敏感性方面得到更进一步的发展。该文根据笔者在瑞典近三十年的工作经验和积累的文献,首先阐明焦磷酸光化测序的基本原理,然后介绍该技术的应用,最后讨论其发展前景。
关键词: 人类基因组计划; 焦磷酸光化测序; 基因变异; 基因型; DNA甲基化;
Abstract: The Human Genome Project(HGP) not only greatly improved the understanding of human genome and related genetic information, but also promoted the development of technologies in life science research. It was under this historical context that pyrosequencing was invented by Swedish researchers. Pyrosequencing is a method of DNA sequencing based on the "sequencing by synthesis" principle with enzymatic reactions, and relies on light detection based upon a chain reaction when pyrophosphate is released. The application of this technology involved the detection of DNA nucleotide sequences and mutations, the identification of genotypes of single nucleotide polymorphisms, the analysis of changes in DNA methylation levels etc. With the recent rapid development of photographic equipment and imaging technology, this technology is expected to have an increasing sensitivity in signal detection. Based upon the working experiences in Sweden for nearly 30 years and accumulated literature, this review first clarified the basic principles of pyrosequencing, then introduced the applications of this technology, and finally discussed its development prospects in the near future.
Keyword: Human Genome Project; pyrosequencing; genetic variation; genotype; DNA methylation;
1 、引言
本世纪是生命科学的世纪。生命科学是研究生命现象,揭示生命活动规律和本质的一门学科。在本世纪所有的生命科学研究活动中,规模最大、发展最快和影响最广的科研项目是人类基因组计划(Human Genome Project,HGP)。HGP旨在测定人类染色体(指单倍体)中所包含的60亿(3 000厘摩根,c M,centi-Morgen,参见说明)对核苷酸序列,绘制出载有完整基因及其序列生物学信息的人类基因组图谱,为破译人类遗传信息,深入开展疾病分子病理学研究和提高人类健康水平服务[1]。人类基因图谱早期包括物理图谱和遗传图谱,前者主要将序列标签位点在染色体的绝对位置上进行线性排列,而后者主要根据遗传学连锁和重组的关系确定序列标签位点在染色体的相对位置。HGP于1990年正式启动,作为人类基因组组织(Human Genome Organisation,HUGO)成员之一,作者有幸参加了早期基因图谱制备的科研工作。至1994年9月,物理图谱和遗传图谱已经完成,基因组标签分辨率为1 cM(即1%重组率),为全基因组测序奠定了基础,从此HGP进入了大规模测序阶段。
1953年,英国剑桥大学科研技术工程师Francis Crick和来自美国的James Waston博士共同发现了DNA双螺旋结构以及相关基本特性[2],鉴于他们在生命科学研究中的重大贡献,这两位科学家分享了1962年诺贝尔生理学或医学奖。DNA碱基排列顺序中存在生物的遗传信息,测定其序列尤为重要。1977年,英国的生化学家Frederick Sanger发明了双脱氧链终止法测定DNA核苷酸序列,这项技术的原理是通过使用类似于正常dNTP的2',3'-双脱氧核苷三磷酸dd ATP、dd TTP、dd CTP、dd GTP,将延伸的DNA链特异性地终止。整个反应体系包括单链模板、引物、DNA聚合酶和4种dNTP。待测定样本共分四组,每组按一定比例加入一种dd NTP,在引物与模板互补和延伸的过程中,它能随机掺入合成的DNA链,一旦掺入合成即终止,于是形成各种不同大小的片段,其末端核苷酸必定为该种核苷酸,再通过电泳分离可以读取DNA的核苷酸序列。这项技术以其姓氏Sanger命名而沿用至今,尽管DNA片段的分离方法已改进为毛细管电泳法,标记系统也从放射自显影转化为荧光素标记,人工读序的实验程序已成为全自动测序技术,但基本原理没有改变[3]。Sanger博士在1958年因发现胰岛素的氨基酸序列而获得诺贝尔化学奖,而于1980年因发明双脱氧链终止法测定DNA核苷酸序列的技术而再次获得诺贝尔化学奖。因此,他像居里夫人那样,成为两次获得诺贝尔奖的科学家。

1994年,HGP进入了大规模测序阶段的关键时刻,迫切需要速度更快、原理更先进的测定DNA核苷酸序列的技术。正是在这样的历史背景下,瑞典乌普萨拉大学和斯德哥尔摩皇家理工学院两个科研小组分别开始研发DNA核苷酸序列测定自动化和新型DNA焦磷酸光化测序法,其目的是为人类基因组DNA大规模序列检测服务。然而,科学研究有时会出现“正打歪着”的现象,这两个科研小组最终都没有达到预期的目的。但是,前者相继开发出垫锁式探针(padlock probe)[4]、滚环DNA扩增技术(rolling circle amplification)[5]、邻位接合技术(proximity ligation assay,PLA)[6]和邻位接合延伸(proximity extension assay,PEA)[7]等一系列先进的核苷酸和蛋白质检测技术,而后者发明了焦磷酸光化测序技术(pyrosequencing)。本文首先介绍焦磷酸光化测序技术的基本原理,然后阐明这项技术的应用范畴,最后讨论其发展前景。
2、 焦磷酸光化测序技术的基本原理
1993年,瑞典科研人员Bertil Pettersson、Mathias Uhle和P?l Nyren根据固相测序的基本方法,使用链霉抗生物素蛋白(strepavidin)包被的磁珠黏附待测序的生物素(biontin)标记的单链DNA,然后添加三种酶(DNA聚合酶、ATP硫酸化酶和荧光素酶)和核苷酸(dNTP)的混合物,通过掺入核苷酸产生的焦磷酸来测量发射的光。光的强度决定是否已掺入一个或更多个核苷酸,因此,可以检测模板链上存在多少个互补核苷酸以及哪一种核苷酸。把四种核苷酸逐一加入重复该过程,就可以确定单链模板的DNA序列,这是焦磷酸光化测序法发展的初级阶段[8]。1998年,Mostafa Ronaghi、Mathias Uhle和P?l Nyren通过加入腺苷三磷酸双磷酸酶(Apyrase)去除未被DNA聚合酶掺入的核苷酸和残留的少量ATP,使得整个技术程序更加完善,四种酶即DNA聚合酶、ATP硫酸化酶、荧光素酶和腺苷三磷酸双磷酸酶的混合物在整个过程中保持化学反应,从而适合于仪器自动化检测。他们的科研论文在Science上发表,标志着pyrosequencing正式进入实际应用阶段[9]。次年,德国凯基(Qiagen)公司基于这项技术的原理将焦磷酸光化测序仪推向市场。目前,在中国无论是在百度等网络上,还是科研论文中,关于pyrose-quencing的中文翻译主要是焦磷酸测序,这个专有名词的中文翻译既不全面也不准确,不符合信达雅的要求。因此,根据这项实验技术的基本原理,作者建议pyrosequencing正确的中文译名应当为“焦磷酸光化测序法”,因为焦磷酸发出的可见光才是这项技术的本质和亮点。
焦磷酸光化测序法主要基于四种酶的酶促反应。根据图1所示,pyrosequencing的基本原理是以单链DNA(ssDNA)为模板,首先与以待测DNA核苷酸序列而设计的特异性引物杂交,并与DNA聚合酶、ATP硫酸化酶、荧光素酶和腺苷三磷酸双磷酸酶以及底物腺苷5'-磷酰硫酸盐(adenosine-5'-phosphosulfat,APS)一起孵育。分别添加四种脱氧核苷酸三磷酸(dNTPs)(注意不是双脱氧的ddNTPs,与Frederick Sanger博士发明了双脱氧链终止法有根本的区别)。DNA聚合酶每将正确互补的一个dNTP整合到模板上,掺入互补的新链就释放一个焦磷酸盐(PPi)。未掺入的核苷酸和ATP被腺苷三磷酸双磷酸酶降解,并且反应可以用另一种核苷酸重新开始。ATP硫酸化酶在腺苷5'磷酸硫酸盐存在下将PPi转化为ATP。该ATP充当荧光素酶介导荧光素转化为氧化荧光素的底物,从而产生可见光。这个光的强度与ATP的量成比例,通过照相机检测荧光素酶催化的反应中产生的光测定在程序中分析相对应的DNA核苷酸序列。该技术不需要凝胶电泳,也不需要对DNA样品进行标记和染色,具有大通量、低成本、快速、直观的特点。与Sanger测序法相比,焦磷酸光化测序技术有其特定的优势,已经成为DNA分析研究的重要手段。
图1 焦磷酸光化测序法的基本原理
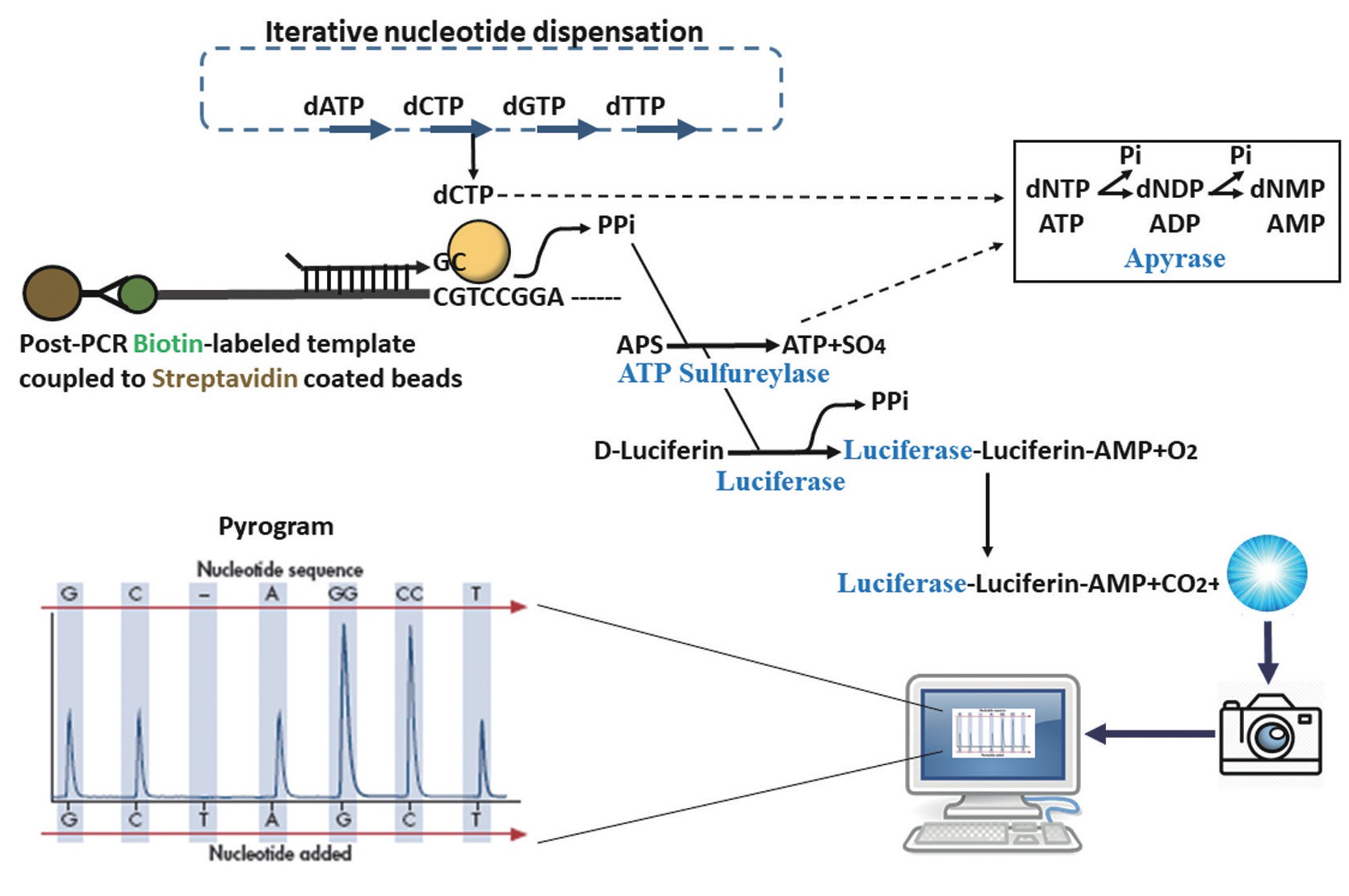
焦磷酸光化测序公司(Pyrosequencing AB)位于瑞典乌普萨拉市,由HealthCap风险投资公司支持而成立,目的是使焦磷酸光化测序技术的仪器和实验试剂盒商业化。1999年,该公司在斯德哥尔摩证券交易所上市,2003年更名为Biotage公司。焦磷酸光化测序业务线于2008年被德国凯基(Qiagen)收购。该公司先后推出PyroQ96、Pyro24h和Pyro48仪器,目前,在中国市场上可购买到Pyro48仪器。焦磷酸光化测序法已经在基因组学、分子遗传学、表观遗传学、临床分子诊断、细菌与病毒分型及法医鉴定等方面得到广泛的应用。近年来,焦磷酸光化测序技术进一步被授权给454生命科学公司,旨在开发基于阵列的焦磷酸测序技术,该技术作为大规模DNA测序的平台出现,包括基因组测序和宏基因组学[10]。
3 、焦磷酸光化测序技术的应用
开发焦磷酸光化测序技术的初衷是为了大规模测定DNA核苷酸序列而服务的,但是,令人尴尬的是由于这项技术的基本原理是通过酶化学反应而产生可见光来检测实时互补的核苷酸,而酶化学反应的代谢产物不可避免地会改变反应条件,因此,当焦磷酸光化测序反应到达150~400个碱基,整个实验由于准确性和敏感性的要求不得不停止。此外,该技术检测三个或者三个以上重复序列有困难,譬如根据光的峰值难以鉴别是GG,还是GGG,或者是AAA,还是AAAA。正当焦磷酸光化测序技术应用处于困惑的艰难时刻,HGP开始实施单核苷酸多态性(single nucleotide polymorphism,SNP)图谱研究,SNP分析并不需要很长的序列,可以采用焦磷酸光化测序法技术来检测基因型。更为重要的是,2006年,Andrew Z.Fire和Craig C.Mello因发现小RNA干扰和基因沉默技术而获得了诺贝尔生理学或医学奖,极大地促进了与小RNA,如RNAi和mi RNA等相关的研究[11]。小RNA片段的序列仅有20多个核苷酸,而焦磷酸光化测序法技术完全可以满足小RNA序列的检测。因此,可以说小RNA的科研工作给了焦磷酸光化测序技术起死回生的发展机会。
焦磷酸光化测序技术的应用主要涉及分子遗传学和表观遗传学领域,下面就焦磷酸光化测序技术的应用问题分三个方面叙述。
3.1、 遗传基因突变的检测
基因突变(gene mutation)是指基因在结构上发生碱基对组成或者排列的改变。基因突变可分为两种主要方式:一种是遗传性突变,是从父母遗传的,并且在人的一生中几乎存在于身体的每个细胞中。另一种是获得性(或体细胞)突变,发生在人的一生中的某个时间,并且仅存在于某些细胞中,而不存在于身体的每个细胞中。这些变化可能是由环境因素引起的,例如来自太阳的紫外线辐射,或者由于DNA在细胞分裂过程中自身复制发生错误而发生的。体细胞获得的突变一般不能传递给下一代。人体具有DNA损伤修复等机制,可通过将突变序列恢复到原始状态来预防或纠正突变。但是,如果没有能够修复,就会导致人体机能发生障碍,产生疾病。所有的癌症都是由于癌细胞基因组DNA序列发生改变而产生的。过去我们已经对基因突变在癌症的病理发生中的作用有所了解。然而,我们现在正进入一个能够获得大量癌症基因组的完整DNA序列的时代。这些研究将为我们提供一个详细和全面的视角,了解个体癌症是如何发展的[12]。譬如,现有的研究资料表明,人群中约有12%的女性在其一生中的某个时候会患上乳腺癌。携带有BRCA1或BRCA2基因突变的女性,患乳腺癌的风险大大增加,可达70%[13,14]。尽管每年进行磁共振成像(MRI)和乳腺X线摄影筛查有助于早期发现乳腺癌,但通过检测基因突变可以早期和准确地诊断乳腺癌,为提高人民的健康水平服务。
检测基因突变的最直接有效的方法就是DNA序列检测。我们知道目前有很多种分析DNA序列的技术,譬如前面所提及的Sanger双脱氧链终止法就是最常用的DNA序列检测技术的基本原理。尽管取得了相当大的进展,DNA测序仍然有些耗时和昂贵,需要三个独立的步骤从模板生成测序产物:扩增目标序列、纯化扩增产物,以及测序反应。常规的方法是将PCR扩增和循环测序结合成一个单一的反应,从而实现基因组DNA的直接测序。以大染料测序试剂(应用生物系统)为载体,加入不同量的正、反向引物、脱氧核苷酸三磷酸(dNTPs)和输入DNA,建立了联合扩增和测序反应。反应被热循环35~45个周期,然后用毛细管电泳对产物进行分析,以检测测序产物。人们利用这项技术进行了多种测序应用,包括鉴定基因的种系突变/多态性,对肿瘤DNA进行测序以鉴定靶基因的体细胞突变等[15]。与常规的Sanger测序技术相比,焦磷酸光化测序法尽管在测序阅读长度具有约400个碱基的上限,但它更具有定量优异的检测限,可以快速准确定量地检测小片段DNA序列变异,如果扬长避短地采用焦磷酸光化测序法技术,可以使得整个实验具有很高的性价比。
3.2 、SNP等位基因分型的签定
单核苷酸多态性(SNP)是在DNA特定位置发生的单个核苷酸置换,是最常见的遗传变异类型。在人类基因组中,每1 000个核苷酸可以出现一次或者多次,这意味着一个人的基因组中大约有400万~500万个SNP。这些遗传变异具有特异性,在世界各地的人群中已发现超过1亿个SNP。更重要的是,SNP可以作为生物标志物,有助于科学家找到与复杂性疾病,如高血压、糖尿病和肿瘤等相关的易感基因或者拮抗基因。当SNP发生在基因内或基因附近的调控区域内时,它们可能通过影响基因的功能在疾病中发挥病理性作用,也可能引起个体对某些药物或者环境的不同反应。因此,鉴定SNP等位基因型是分子遗传学、遗传病理学和遗传药理学等学科最常用的实验技术。
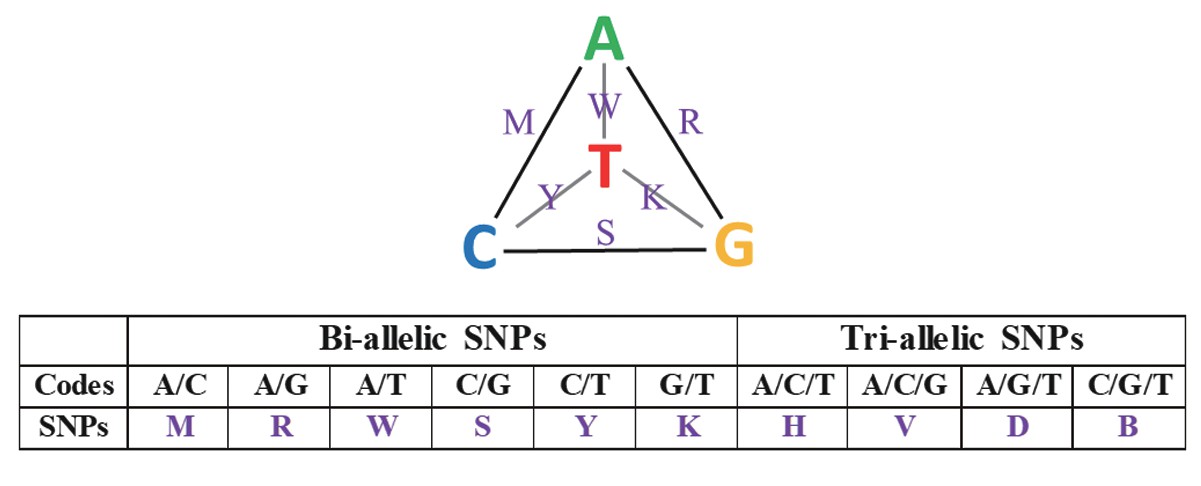
SNP通常由两个等位基因组成,也可能由三个等位基因组成。图2表明在人类基因组中可能出现的各种SNP,图中归纳了所有SNP的代码均是由美国生物化学学会设计和命名的,最常见的SNP是Y=C/T。全世界科学家发现的人类基因组中所有的SNP图谱都汇总记录在SNP数据库,如db SNP(https://www.ncbi.nlm.nih.gov/snp/),这些数据库作为人类的共同信息资料,可公开免费使用。目前,世界上有很多种技术可以用于SNP基因型等位基因分型,这些技术的设计一般是为了分析SNP两个等位基因特有的核苷酸。但是,焦磷酸光化测序法与它们不同,具有独特的技术原理和优势,不仅可以分析由两个等位基因组成的SNP,而且可以鉴定用其他技术无法分析的由三个等位基因组成的SNP,还可以发现已鉴定SNP的周边有没有其他DNA变异。作者所在的实验室曾用动态等位基因特异性杂交技术(dynamic allele specific hybridisation,DASH)研究ICAM-1(intercellular cell adhesion molecule-1,即细胞间黏附分子-1)基因与糖尿病和糖尿病肾病的关联性[16,17]。该基因有一个非同义的单核苷酸多态性,由19号染色体10285007位的E(Glu)密码子GAG和K(Lys)密码子AAG之间的替换引起(Contig ID NT_011295.12,GRCCH38.p7)。结果表明,在瑞典人群体,这个SNP多态性基因型分布中的杂合子频率很高,Kristiansen等[18]和Nishimura等[19]两个科研小组尽管都发表了这个SNP丹麦与和日本人1型糖尿病相关联的文章,但是前者在论文中没有表明其基因型的分布结果。通过与作者交流原始实验结果,这个SNP多态性基因型分布中的杂合子频率在所研究的人群中都很高。一般来说,基因型分布中的高杂合子频率可能是由于基因分型的技术限制或基因组重复(genome duplicon)所造成[20]。为了验证实验的准确性,避免错误结果的发生,我们有必要用另一种基因分型原理不同的技术,即焦磷酸光化测序法重复基因分型实验。最终的实验结果表明,这个SNP高杂合子频率的现象在瑞典、丹麦、日本和马来西亚的人群体中存在[18,19,21,22,23]。这个具体的实验充分说明焦磷酸光化测序法独特的技术原理可以用于基因分型验证等类似的实验研究。
图2 人类基因组中单核苷酸多态性的类型和代码
3.3、 DNA甲基化水平的分析
表观遗传学(epigenetics)主要研究基于DNA序列未发生改变而基因的功能和特性发生异常变化,包括DNA甲基化、组蛋白修饰、染色体重塑和非编码RNA调控等。在人类基因组中,DNA甲基化发生在体细胞中DNA两条链上的胞嘧啶(5mecytosine)环的5-位(CpG),通常DNA甲基化以Cp G岛的形式在基因的启动子或者其他区域出现。现有的资料表明,DNA甲基化的异常变化模式与生长发育、环境影响以及肿瘤等复杂性疾病的发生和发展密切相关。因此,分析DNA甲基化水平的变化已经成为表观遗传学实验研究的主要内容之一。
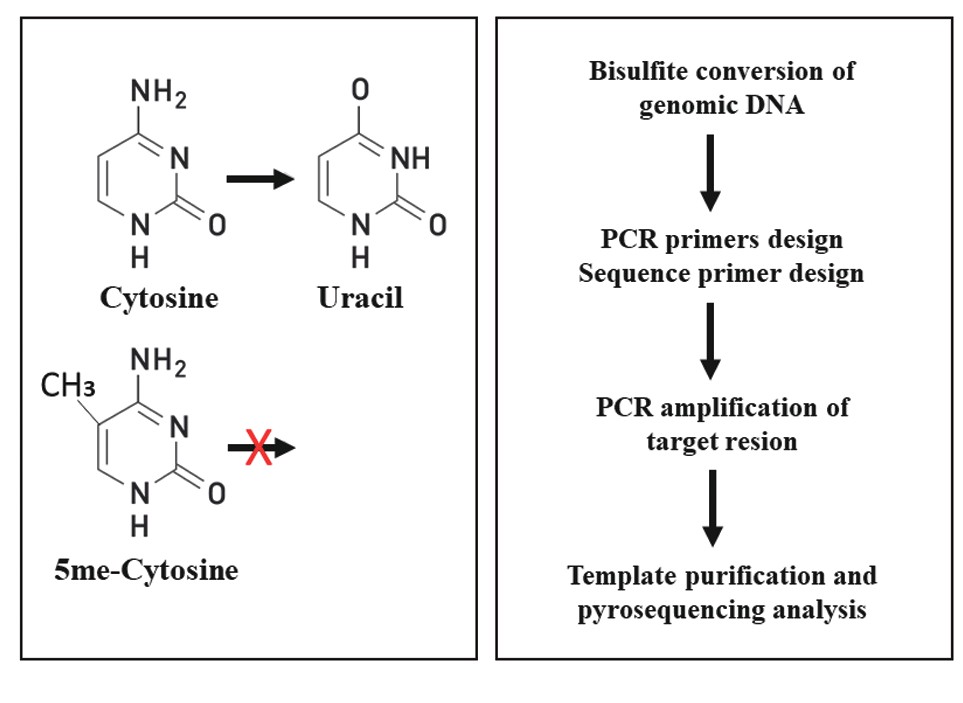
近年来,已经开发了多种用于DNA甲基化研究的技术。但是,相比较而言,焦磷酸光化测序法是一种实时DNA合成测序方法,通过酶促级联反应可以定量将核苷酸掺入时释放的焦磷酸转化为光信号,因此非常适用于亚硫酸氢盐(bisulfite)处理后的DNA甲基化分析。如图3所示,应用焦磷酸光化测序法进行DNA甲基化分析的整个实验程序包括两个部分。第一部分,将待检测的DNA样本用亚硫酸氢盐处理,使得未甲基化的胞嘧啶转化为尿嘧啶(uracil),而待检测甲基化的胞嘧啶(5mecytosine)保持原样不变。第二部分,通过特异性引物和PCR扩增制备DNA模板,在亚硫酸氢盐处理和PCR之后,DNA模板序列中每个CpG位置的每个甲基化程度由T和C的比率确定,此刻用焦磷酸光化测序法就完全可以量化分析发生甲基化的胞嘧啶水平。作者所在实验室的工作实践表明,无论某基因DNA甲基化水平较低还是很高,采用焦磷酸光化测序法分析这些靶基因DNA甲基化水平的变化,都可以取得很好的结果。譬如,最近的研究表明,溶质载体家族30成员8(solute carrier family 30 member8,SLC30A8)是2型糖尿病易感基因[24,25],我们用焦磷酸光化测序法研究马来西亚2型糖尿病患者中该基因DNA甲基化水平的变化,结果表明SLC30A8基因启动子上六个CpG位点平均基因甲基化水平高达约80%,显着高于非糖尿病对照组[26]。胰岛素样生长因子结合蛋白7(insulin-like growth factor-binding protein 7,IGFBP7)基因DNA甲基化水平较低(约15%)。我们分析该基因DNA甲基化水平,发现该基因甲基化水平在男性2型糖尿病患者中明显高于非糖尿病对照组,但在女性2型糖尿病和非糖尿病人群中未发现显着性差异。IGFBP7在胰岛素抵抗的表观遗传学作用方面具有性别差异[27]。在人类基因组中,LINE-1(long interspersed nucleotide element-1)在人类基因组中广泛分布,最近,我们采用Pyro Q96仪和焦磷酸光化测序法分析了英国2型糖尿病患者在14年随访期间基因组DNA中LINE-1 DNA上4个CpG的变化,结果表明整个基因组DNA LINE-1甲基化平均水平为76%,在女性2型糖尿病患者中,LINE-1 DNA甲基化水平的变化与舒张压、肾小球滤过率和胆固醇高密度脂蛋白比值等代谢标记物之间显着负相关[28]。
图3 焦磷酸光化测序法DNA甲基化水平的实验程序
把上述三个研究实例总结起来可以表明,用焦磷酸光化测序法分析DNA甲基化水平具有以下优点:(1)该技术可以直接定量测定甲基化的胞嘧啶;(2)通过DNA样本亚硫酸氢盐的处理,可以使所有检测DNA甲基化统一定量而保证准确性;(3)将与甲基化的胞嘧啶相互补的核苷酸掺入时释放的焦磷酸转化为光信号检测,具有较高的敏感性。因此,焦磷酸光化测序法已经被认为是DNA甲基化分析的金标准实验方法。
4 、焦磷酸光化测序技术的发展前景
生命科学研究技术的发展不是孤立的,而是与工业革命带来的新技术相铺相成共同发展的。从过去的几十年到未来的十年,工业革命在计算机技术、数字化照相和成像技术以及5-G通讯技术等方面,肯定会给现代人类生活和生命科学研究带来很大的改变。正如前面所言,焦磷酸光化测序法技术的本质和亮点是产生焦磷酸而发出的可见光。因此,数字化照相和成像的进步,计算机分析能力的提高,5-G通讯技术的快捷,都有可能促进焦磷酸光化测序技术的改进和新的发展。作者认为主要有两个可能性:第一个是通过检测系统的敏感性的提高,极大地提高对焦磷酸所产生的可见光的捕获和量化分析,这样就有可能只要少量的DNA模板,甚至不需要PCR扩增而直接对于DNA样本进行焦磷酸光化测序法测序。如果能够达到这个水平,那整个实验流程不仅可以节约至少一半的费用,而且可能避免由于PCR扩增出现的误差和人工合成DNA对环境的污染。第二个是通过检测系统敏感性和分辨率的提高,使得焦磷酸光化测序法可以分析模板DNA序列中三个或者三个以上重复序列,譬如GGG或者AAAA等,从而克服了该技术原有的缺陷。总之,在不久的将来,焦磷酸光化测序法很可能从众多的DNA检测技术中脱颖而出,成为一机多用、快捷简便和准确有效的生命科学技术。
说明:厘摩根cM(centi-Morgan)是遗传学上确定基因重组频率的测量单位,是由美国遗传学家Alfred Henry Sturtevant为了纪念他的老师,着名的遗传学家Thomas Hunt Morgan而建立的。1个cM定义为重组频率100次中发生1次。如果两个基因间相距1个cM,那么其后代与父母相比,有1%的个体具有不同的等位基因频率。如果相距50 cM,就意味着这两个基因是不连锁的,很有可能是位于不同的染色体上。对于人类基因组而言,1 cM平均代表7.5×105 bp的距离。
参考文献
[1] Green ED, Watson JD, Collins FS. Human Genome Project:twenty-five years of big biology. Nature, 2015,526:29-31
[2] Watson JD, Crick FH. Molecular structure of nucleic acids:a structure for deoxyribose nucleic acid. Am J Psychiatry, 2003, 160:623-4
[3] Sanger F. Determination of nucleotide sequences in DNA.Biosci Rep, 2004, 24:237-53
[4] Nilsson M, Malmgren H, Samiotaki M, et al. Padlock probes:circularizing oligonucleotides for localized DNA detection. Science, 1994, 265:2085-8
[5] Larsson C, Koch J, Nygren A, et al. In situ genotyping individual DNA molecules by target-primed rolling-circle amplification of padlock probes. Nat Methods, 2004, 1:227-32
[6] Fredriksson S, Gullberg M, Jarvius J, et al. Protein detection using proximity-dependent DNA ligation assays.Nat Biotechnol, 2002, 20:473-7
[7] Assarsson E, Lundberg M, Holmquist G, et al. Homogenous96-plex PEA immunoassay exhibiting high sensitivity,specificity, and excellent scalability. PLoS One, 2014, 9:e95192
[8] Nyrén P, Pettersson B, Uhlén M. Solid phase DNA minisequencing by an enzymatic luminometric inorganic pyrophosphate detection assay. Anal Biochem, 1993, 208:171-5
[9] Ronaghi M, Uhlén M, Nyrén P. A sequencing method based on real-time pyrophosphate. Science, 1998,281:363,365
[10] Margulies M, Egholm M, Altman WE, et al. Genome sequencing in microfabricated high-density picolitre reactors. Nature, 2005, 437:376-80
[11] Fire AZ. Gene silencing by double-stranded RNA. Cell Death Differ, 2007, 14:1998-2012
[12] Stratton MR, Campbell PJ, Futreal PA. The cancer genome. Nature, 2009, 458:719-24
[13] Kotsopoulos J. BRCA mutations and breast cancer prevention. Cancers(Basel), 2018, 10:E524
[14] Narod SA, Salmena L. BRCA1 and BRCA2 mutations and breast cancer. Discov Med, 2011, 12:445-53
[15] Murphy KM, Berg KD, Eshleman JR. Sequencing of genomic DNA by combined amplification and cycle sequencing reaction. Clin Chem, 2005, 51:35-9
[16] Howell WM, Jobs M, Gyllensten U, et al. Dynamic allelespecific hybridization. A new method for scoring single nucleotide polymorphisms. Nat Biotechnol, 1999, 17:87-8
[17] Prince JA, Feuk L, Howell WM, et al. Robust and accurate single nucleotide polymorphism genotyping by dynamic allele-specific hybridization(DASH):design criteria and assay validation. Genome Res, 2001, 11:152-6
[18] Kristiansen OP, Nols?e RL, Holst H, et al. The intercellular adhesion molecule-1 K469E polymorphism in type 1diabetes. Immunogenetics, 2000, 52:107-11
[19] Nishimura M, Obayashi H, Maruya E, et al. Association between type 1 diabetes age-at-onset and intercellular adhesion molecule-1(ICAM-1)gene polymorphism. Hum Immunol, 2000, 61:507-10
[20] Marques-Bonet T, Eichler EE. The evolution of human segmental duplications and the core duplicon hypothesis.Cold Spring Harb Symp Quant Biol, 2009, 74:355-62
[21] Ma J, M?llsten A, Prázny M, et al. Genetic in?uences of the intercellular adhesion molecule 1(ICAM-1)gene polymorphisms in development of type 1 diabetes and diabetic nephropathy. Diabet Med, 2006, 23:1093-9
[22] Abu Seman N, Anderstam B, Wan Mohamud WN, et al.Genetic, epigenetic and protein analyses of intercellular adhesion molecule 1 in Malaysian subjects with type 2diabetes and diabetic nephropathy. J Diabetes Complications,2015, 29:1234-9
[23] Gu HF, Ma J, Gu KT, et al. Association of intercellular adhesion molecule 1(ICAM1)with diabetes and diabetic nephropathy. Front Endocrinol(Lausanne), 2013, 3:179
[24] Flannick J, Thorleifsson G, Beer NL, et al. Loss-offunction mutations in SLC30A8 protect against type 2diabetes. Nat Genet, 2014, 46:357-63
[25] Gu HF. Genetic, epigenetic and biological effects of zinc transporter(SLC30A8)in type 1 and type 2 diabetes. Curr Diabetes Rev, 2017, 13:132-40
[26] Seman NA, Mohamud WN,?stenson CG, et al. Increased DNA methylation of the SLC30A8 gene promoter is associated with type 2 diabetes in a Malay population.Clin Epigenetics, 2015, 7:30
[27] Gu HF, Gu T, Hilding A, et al. Evaluation of IGFBP-7DNA methylation changes and serum protein variation in Swedish subjects with and without type 2 diabetes. Clin Epigenetics, 2013, 5:20
[28] Malipatil N, Lunt M, Narayanan RP, et al. Assessment of global long interspersed nucleotide element-1(LINE-1)DNA methylation in a longitudinal cohort of type 2diabetes mellitus(T2DM)individuals. Int J Clin Pract,2018:e13270