摘 要: 肠道微生物与寄主动物长期共存、协同进化, 在动物的宿主防御、消化吸收、免疫调节、维生素合成等生命活动过程中起着十分重要的作用.肠道微生物与寄主动物共同构成“超级生物”, 被称为动物的“第二基因组”.近年来随着分子生物学技术发展和宏基因组技术应用, 肠道微生物的研究已经成为热点领域.鸟类由于具有飞翔能力, 可以大范围、远距离传播和扩散微生物, 其肠道微生物组成更加复杂多样.目前的研究表明鸟类肠道常见的微生物有五个门, 其中厚壁菌门 (Firmicutes) 和拟杆菌门 (Bacteroidetes) 占据主导地位, 但多样性仍需要进一步深入研究.鸟类肠道微生物多样性及其维持机制和功能解析将成为候鸟病原微生物流行病学、迁徙水鸟肠道微生态学等研究的重要方向和理论基础.
关键词: 肠道微生物; 超级生物体; 鸟类; 宏基因组; 肠道微生态学;
Abstract: Gut microorganisms' co-existing and co-evolving with host animals play a very important role in host defense, digestion and absorption, immune regulation, vitamin synthesis and other life activities, forming “superorganism”with animals.Therefore, gut microbiome is known as the “second genome”of animals.In recent years, with the development of molecular biology technology and the application of metagenomic technology, the study of gut microorganisms has become an important research field.Because of their flying ability, birds can get and spread microbes over vast distances, resulting in complex and diversegut microbiome.Current studies have shown that the common microorganisms in the gut tracts of birds have five phylas, with Firmicutes and Bacteroidetes dominating, but their diversity still needs further study.Gut microbial diversity and its maintenance mechanism function analysis will be an important direction and theoretical basis for the study of pathogenic microbial epidemiology of migratory birds, and gut microecology of migratory water birds in the future.
Keyword: gut microbiota; superorganism; birds; meta-genome; gut microecology;
肠道是动物从外界获得营养的重要器官, 是消化吸收的重要场所.典型的肠道自口至肛门分别由前肠、中肠和后肠3个部分组成, 食物经前端由口摄入, 通过肠道的营养消化吸收后, 残渣成为粪便由后端的肛门排出.低等动物的肠道极其简单, 随着动物由低等向高等进化, 肠道不断分化, 结构趋于复杂, 功能更加多样化.肠道与外界环境紧密关联, 是动物有机体从外界获得营养的重要界面, 因而也成为环境微生物寄生的重要场所.肠道微生物与动物相互作用、协同进化, 关系密切[1].但是人们关于肠道微生物与寄主动物关系的认识仍十分有限[2].
肠道微生物极其丰富, 高等动物肠道的细菌种类约500~1 000种[3], 其鉴定和分类是充分研究这些微生物的前提条件.传统的分类手段是通过微生物的细胞形态、生理生化和遗传学等手段进行.20世纪60年代以后, 人们根据微生物的细胞化学组分、基因型特征, 采用化学和遗传手段, 同时结合数值分类方法等现代手段进行分类.但这些方法在研究如此庞大数量的微生物菌群略显不足, 因为要获得纯培养菌群, 对大量难培养和不可培养的微生物进行研究都是相当困难的.随着分子生物学技术的快速发展, 分子标记技术和高通量测序技术广泛地应用于微生物分类研究, 成功地解决了上述难题, 肠道微生物的研究获得前所未有的机会[4].
鸟类具有较强的飞翔能力, 其生活史复杂, 常常面临更加复杂的环境选择压力, 因此其肠道微生物显得更加复杂, 导致这一领域的研究更具挑战性[5].随着动物肠道微生物研究的不断深入, 鸟类肠道微生物的研究将越来越受到重视[6,7].笔者简要介绍肠道微生物学和微生态学研究进展以及现代分子生物学技术在肠道微生物学研究中的应用, 阐述鸟类肠道微生物基本概况、重点研究内容和研究展望, 为相关研究者提供参考.

1 、肠道微生物学和微生态学
1.1、 肠道微生物学
肠道微生物学 (Gut Microbiology) 是近年来快速发展的一个动物学和微生物学研究新领域.肠道是动物与外界环境联系最为密切的界面, 除了营养物质的消化、吸收和物质交换功能外, 也为细菌、真菌等微生物的定殖、寄生提供良好的环境条件, 成为动物-微生物相互作用的重要场所[2,5,8].肠道微生物随着寄主发育而建立起来, 并随着年龄增加而变化, 逐渐形成稳定的群落[2,9,10].肠道微生物菌群 (gut microbiota) 极其丰富, 包括细菌群、真菌群、原生生物等[3], 其种类和数量受到寄主肠道形态结构、生理、年龄、性别、行为、生活习性和环境因素的影响[2,5,10,11,12], 并在寄主动物生命活动过程中发挥重要作用, 如形成肠道内的菌膜屏障抑制有害菌入侵、调节免疫功能、控制能量同化、促进消化、促进维生素和非必需氨基酸的合成, 参与糖类和蛋白质的代谢, 同时还能促进铁、镁、锌等矿物元素的吸收等[6,7,13], 因而成为维持动物健康的一个重要“器官”[3,14].因此, 动物及其肠道微生物成为不可分割的整体, 构成动物-微生物菌群复合体 (metaorganism) 和超级生物体 (superorganism) [10,15].最早受到关注的肠道微生物 (特别是细菌) 大多与人关系较为密切, 因而针对人、实验动物小鼠、家禽和家畜的肠道微生物研究工作也较为深入[16,17].近年来, 人们关于肠道微生物对于寄主生理和健康意义的认识也在不断深化[17], 并且已经将关注点聚焦到野生动物肠道微生物菌群的形成和维持机制及其对于寄主适合度的潜在意义[3,5,17,18].
1.2 、肠道微生态学
肠道微生物的研究不断深入, 推动了肠道微生态学 (Gut Microecology) 的发展.动物肠道是一个独特的微生态系统, 微生物群落和食物或食物残渣构成微生物生态系统[14].肠道微生物相互之间, 以及微生物和寄主之间存在稳定复杂的关系, 互利共生, 协同进化, 构成寄主-微生物 (host-microbiome) 交互作用的复杂系统[19,20].动物肠道微生物群落结构和功能的稳定受寄主生物学和生态学因素影响[21], 包括地理特征、食物、季节、环境污染、寄生虫感染等环境因素[22,23], 以及寄主种类和性别、免疫、交配模式和行为等寄主生物学因素[6,23,24,25].理解野生动物肠道微生物群落的时空动态和调控机理对于我们理解动物-肠道微生物复合体对环境变化的响应机制, 以及进一步开展疫病生态学、流行病学以及野生动物保护具有十分重要的意义.
2 、分子生物学技术在肠道微生物研究中的应用
传统的微生物学研究手段不能满足动物肠道微生物复合体研究的新形势, 以高通量测序技术为主要手段的宏基因组学 (Metagenomics) 拓展了人们认识环境微生物的能力, 并广泛应用于动物肠道微生物多样性及其结构功能的研究领域, 推动了肠道微生物学和微生物生态学的快速发展.细菌16SrRNA基因是细菌上编码rRNA相对应的DNA序列, 存在于所有细菌的基因组中, 具有高度的保守性和特异性以及该基因序列足够长 (包含约50个功能域) , 提供通用引物的有效靶位点, 含有9个高变区, 成为目前普遍用于肠道微生物分析的分子标记[4].随着PCR技术的发展及核酸研究技术的不断完善, 16s rRNA基因检测技术已成为细菌检测和鉴定的一种强有力工具.同样, 18S和ITS等序列也是真菌和其他真核生物宏基因组学研究的有效途径[26,27,28].第二代、第三代高通量测序和大规模平行测序 (massively parallel sequencing, 简称MPS) 、PCR-DGGE等新技术是目前研究肠道微生物复合体宏基因组十分有效的手段[12,16,20], 通过宏基因组分析实现对肠道微生物群落快速、微量、准确简便的分类鉴定和检测及其功能基因的解析[12].通过生物信息统计分析 (图1) , 可以揭示肠道中微生物的种类以及它们之间的相对丰度和进化关系;此外还可以通过实时荧光定量PCR方法, 定量研究肠道菌群, 探讨微生物多样性, 研究肠道微生物与环境间的相关关系[29].
图1 肠道微生物高通量分析流程
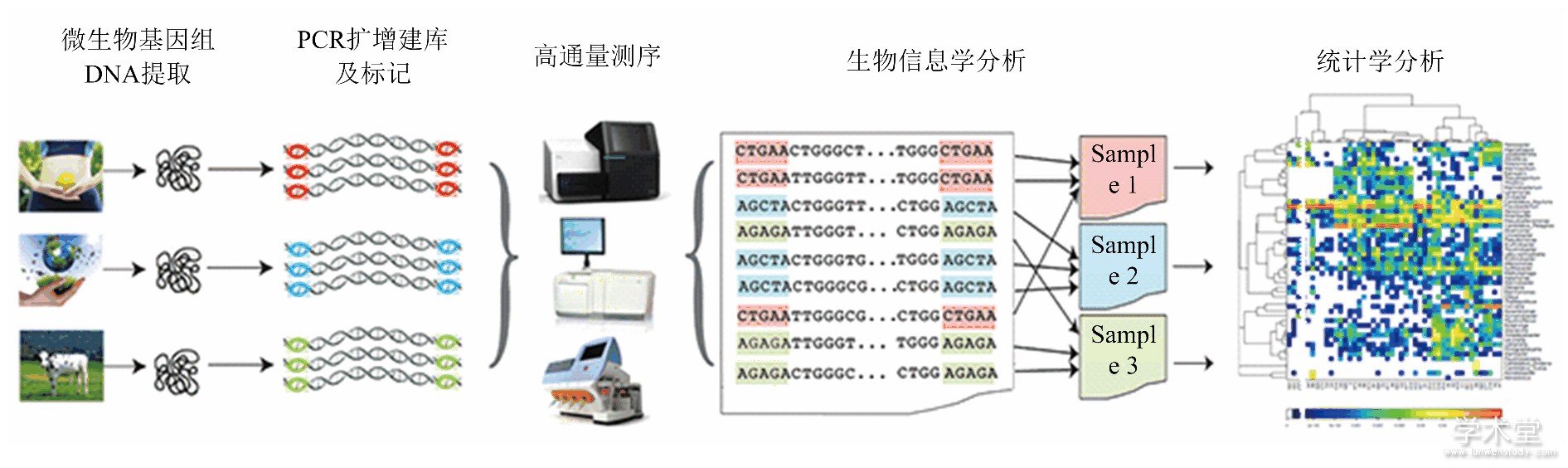
(引自http://www.tinygene.com/microbiota-research-field/gut-fecal-microbiome)
3、 鸟类肠道微生物群落结构
3.1、 鸟类肠道微生物组成特点
鸟类的肠道微生物在高级阶元组成上与哺乳动物十分相似, 在已经认识的75个门中, 厚壁菌门 (Firmicutes) 和拟杆菌门 (Bacteroidetes) 占据了主导地位[5].从已有研究的鸵鸟目 (Struthionformes) 、企鹅目 (Sphenisciformes) 、鸡形目 (Galliformes) 、雁形目 (Anseriformes) 、鹤形目 (Gruiformes) 、鹦形目 (Psittaciformes) 、鹃形目 (Cuculiformes) 、鸥形目 (Lariformes) 等鸟类来看, 其肠道微生物的优势类群集中在厚壁菌门、拟杆菌门、放线菌门 (Actinobacteria) 、变形菌门 (Proteobacteria) 、软壁菌门 (Tenericutes) (表1) [5,30,31].
3.2 、寄主生物学特征对肠道微生物的影响
鸟类的消化道分化为嗉囊、胃、肠 (包括十二指肠、空肠、回肠、盲肠) 和泄殖腔等部分, 胃和肠是消化吸收的重要场所, 消化液的作用和食物在肠道各部位滞留的时间影响肠道微生物分布, 发育过程同样也影响肠道微生物群落发育.家鸡的肠道微生物研究表明, 盲肠中梭菌 (Clostridium) 的水平较高, 而回肠则具更多的乳酸杆菌 (Lactobacillus) [31];家鸡和帽带企鹅 (Pygoscelis antarctica) 的成鸟肠道微生物群落结构有较大差别, 且较雏鸡稳定而复杂[31,32,33].遗传等因素也会影响肠道微生物群落结构.对新热带界59种鸟的肠道微生物研究表明, 相同分类阶元 (目/科/属/种) 的寄主鸟类肠道微生物较相似[34].
表1 若干鸟类肠道微生物群落多样性
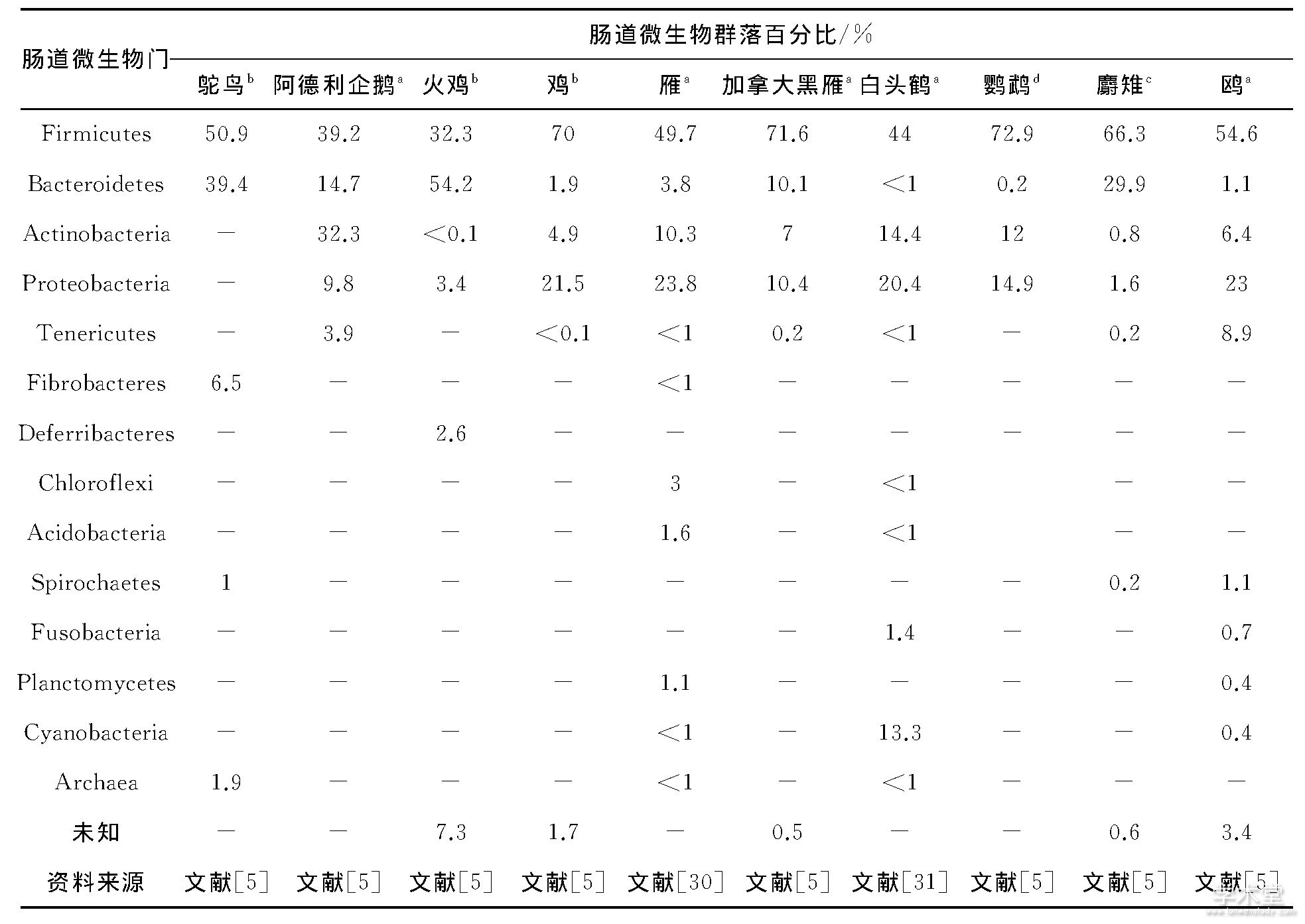
注: (1) 样品:a粪便, b盲肠, c嗉囊, d泄殖腔; (2) 涉及鸟种:阿德利企鹅鸥Pygoscelis adeliae, 加拿大黑雁Branta canadensis, 雁 (Anser fabalis, Anser albifrons, Anser cygnoides) , 鸵鸟Struthio camelus, 火鸡Meleagris gallopavo, 鸡Gallus gallus, 麝雉Opisthocomus hoazin, 鸥Larus spp., 鹦鹉 (数种) , 白头鹤Grus monacha.
3.3 、环境因素对肠道微生物的影响
食物、季节和污染物等环境因素影响鸟类肠道微生物.草食性雁类肠道具有大量与纤维素分解相关的微生物[30];升金湖越冬白头鹤的研究表明, 随着季节的变化肠道微生物的组成发生了改变[31].有机磷杀虫剂处理的雌性日本鹌鹑 (Coturnix japonica) 饲养实验中, 添加低剂量的三氯磷酸酯导致肠道微生物菌群失调[35].
4、 鸟类肠道微生态学的研究热点
鸟类是研究动物-肠道微生物菌群复合体协同进化和适应进化的理想对象.相对于人类和哺乳动物等, 鸟类是脊椎动物中适应辐射能力较强的类群, 种类多、迁徙能力强, 生态位极其复杂多样[7].鸟类的肠道微生物的研究相对较为薄弱, 且以家禽的研究为主.野生鸟类具有复杂的生活史、多样化的食性、生理特征和发育过程, 飞翔生活使它们的生理活动面临较大的选择压力, 从而导致肠道微生物菌群的复杂变化[7,12,14,15,16], 相关的肠道微生物研究也由于受到取样等因素的影响报道则相对较少[5,6].已有的研究表明肠道微生物对寄主的营养、免疫功能和发育都有较大影响[36,37,38], 因此, 开展野生鸟类肠道微生物研究对于全面了解鸟类-微生物菌群复合有机体的结构特点和相互作用过程, 理解自然选择压力下鸟类-微生物的协同进化具有重要意义.在高通量测序技术和宏基因组学研究技术支持下, 鸟类肠道微生物的研究内容不断拓展和深入[39,40,41,42,43], 关注的热点集中在以下方面:
(1) 鸟类肠道微生物菌群复合体结构和功能解析
了解肠道微生物多样性及其对寄主生理功能的影响是阐明鸟类肠道微生物菌群复合体结构功能的重要前提条件.鸟类肠道微生物种类繁多, 目前的研究表明主要包括变形菌、厚壁菌、拟杆菌、放线菌等[41,42,43], 它们在寄主鸟类的营养吸收、免疫、毒素降解和体温调节方面具有重要功能[5,6,7,25].植食性鸟类肠道微生物以拟杆菌和厚壁菌为主[20];一些肠道具有发酵结构的鸟类, 如麝雉 (Opisthocomus hoazin) 和鸵鸟 (Ostrich) , 肠道内纤维素降解微生物较为丰富;在鸡形目的盲肠中存在与糖代谢有关的细菌;一些低蛋白食物的鸟类肠道中出现尿酸代谢菌, 促进氮的回收利用;鸟类肠道中常存在一些与免疫和解毒有关的共生菌, 例如在麝雉的嗉囊中发现降解皂苷菌, 这些共生菌对于取食植物和无脊椎动物的鸟类常摄取的一些次生代谢有毒产物的降解具重要意义[5];雁类肠道微生物宏基因组分析表明, 纤维素降解微生物有利于寄主纤维素的消化[42].由此可见, 了解肠道微生物复合体组成特征对于理解鸟类适应和进化具有重要意义.
(2) 鸟类肠道微生物复合体结构和功能多样性的维持机制
鸟类肠道的核心微生物通常保持相对稳定, 以维持正常的生命活动.鸟类肠道菌群结构既受寄主鸟类本身因素的影响, 又有环境因素影响, 其中寄主的系统地位以及宿主的食性被认为是影响动物肠道菌群组成的两大重要因素, 特别是食性对动物的肠道菌群的形成有极其重要的作用[36,41].从食叶南美麝雉和牛肠道微生物的比较发现, 肠道前段与后段的菌群差别较大, 具有发酵功能的前段, 其菌群甚至与牛的瘤胃类似[44].Roggenbuck等[45]的研究发现秃鹫 (Coragyps atratus, Cathartes aura) 的肠道尽管存在许多致病菌, 但是他们肠道中的微生物的组成和结构却异常稳定, 由于适应腐食生活, 肠道微生物以梭状芽胞杆菌 (Clostridia) 和梭菌 (Fusobacteria) 为优势.松鸡 (Tetrao urogallus) 肠道微生物随着食物资源的变化而呈现季节性变化[46].阿德利企鹅 (Pygoscelis adeliae) 的肠道微生物受地理分布的影响较小, 与其遗传距离表现较强的负相关[20].几种雀形目鸟类肠道微生物的研究显示明显的趋同适应现象, 相反喜鹊巢寄生的大斑凤头鹃 (Clamator glandarius) 幼鸟和寄主喜鹊 (Pica pica) 幼鸟的肠道微生物群落组成有较大差别[39,47].此外, 野生鸟类发育过程中育雏行为对肠道微生物群落发育的影响等都有待于进一步研究[48].随着有机体内外环境因素对于肠道微生物影响研究的不断深入, 鸟类肠道微生物复合体多样性的决定机制仍将是研究热点.
(3) 鸟类肠道微生物病原体的流行病学
野生鸟类与家禽和家畜往往利用同一生境, 同域动物存在交互感染的风险[49,50], 如在禽流感病毒的传播中发挥重要作用, 并且对人类的安全构成威胁.野生鸟类中候鸟具有迁徙习性, 许多鸟类是跨区域迁徙鸟类, 它们最容易携带病原微生物广泛传播[49,51].迁徙水鸟具有集群特征, 常跨区域迁飞, 导致病原体传播[20].此外, 鸟类病原微生物和寄生虫感染导致免疫能力下降, 适合度降低[52].因此, 野生鸟类和家禽的交互传播是值得关注的现实问题.
(4) 迁徙和越冬水鸟类肠道微生态学
迁徙水鸟具有复杂的周年生活史, 它们每年在繁殖地、迁徙停歇地和越冬地之间长距离往返迁飞, 利用多种生境, 面临复杂多样的环境选择压力, 因此成为动物肠道微生物学的理想研究对象[40,42,50].迁徙水鸟肠道微生物菌群复合体的时空变化受众多因素影响.在繁殖地, 迁徙水鸟的繁殖种群与其他鸟类相似[20,42], 通过交配、生长和发育等一系列生活史过程, 新生个体建立起肠道微生物群落, 同时成体之间也进行肠道微生物交流.在迁徙停歇地和越冬地, 水鸟往往集结成群, 聚集在有限的生境中[40,50], 一方面, 同域集群为种内及种间的肠道微生物交换创造条件[40,49,50,51], 另一方面, 生境退化和丧失、种间竞争和人为干扰、严酷的气候条件等环境胁迫使水鸟及其肠道微生物复合体面临复杂的选择压力.因此, 迁徙水鸟迁徙和越冬期肠道微生物复合体结构的时空变化研究, 对于进一步理解水鸟种群生存状况和开展种群保护, 特别是濒危物种种群保护具有重要意义, 同时也为病原生物的流行病学研究提供有益信息.
参考文献
[1] MOELLER A H, CARO-QUINTERO A, MJUNGU D, et al.Cospeciation of gut microbiota with hominids[J].Science, 2016, 353 (6297) :380-382.
[2] CRESCI G A, BAWDEN E.The gut microbiome:what we do and don’t know[J].Nutrition in Clinical Practice, 2015, 30 (6) :734-746.
[3] SOMMER F, BCKHED F.The gut microbiota-masters of host development and physiology[J].Nature Reviews Microbiology, 2013, 11 (4) :227-238.
[4]吴森, 张莺莺, 昝林森.基于高通量测序的宏基因组学技术在动物胃肠道微生物方面的研究进展[J].生物技术进展, 2015, 29 (12) :1721-1735.
[5] KOHL K D.Diversity and function of the avian gut microbiota[J].Journal of Comparative Physiology B, 2012, 182:591-602.
[6] WAITE D W, TAYLOR M W.Characterizing the avian gut microbiota:membership, driving influences, and potential function[J].Frontiers in Microbiology, 2014, 5:223.
[7] WAITE D W, TAYLOR M W.Exploring the avian gut microbiota:current trends and future directions[J].Frontiers in Microbiology, 2015, 6:673.
[8] SOMMER F, BCKHED F.The gut microbiota-masters of host development and physiology[J].Nature Reviews Microbiology, 2013, 11 (4) :227-238.
[9] BARBOSA A, BALAGUV, VALERA F, et al.Age-related differences in the gastrointestinal microbiota of Chinstrap Penguins (Pygoscelis antarctica) [J].PLoS ONE, 2016, 11 (4) :e0153215.
[10] OTTAVIANI E, VENTURA N, MANDRIOLI M, et al.Gut microbiota as a candidate for lifespan extension:an ecological/evolutionary perspective targeted on living organisms as metaorganisms[J].Biogerontology, 2011, 12:599-609.
[11] WAITE D W, TAYLOR M W.Characterizing the avian gut microbiota:membership, driving influences, and potential function[J].Frontiers in Microbiology, 2014, 5:223.
[12] GILL S R, POP M, DEBOY R T, et al.Metagenomic analysis of the human distal gut microbiome[J].Science, 2006, 312:1355-1359.
[13] CANI P D, EVERARD A, DUPARC T.Gut microbiota, enteroendocrine functions and metabolism[J].Current Opinion in Pharmacology, 2013, 13:935-940.
[14] AZIZ Q, DORE J, EMMANUEL A, et al.Gut microbiota and gastrointestinal health:current concepts and future directions[J].Neurogastroenterology&Motility, 2013, 25:4-15.
[15] THOMAS C, BOSCH1G, MCFALL-NGAI M J.Metaorganisms as the new frontier[J].Zoology (Jena) , 2011, 114 (4) :185-190.
[16] YOON S S, KIM E K, LEE W J.Functional genomic and metagenomic approaches to understanding gut microbiota-animal mutualism[J].Current Opinion in Microbiology, 2015, 24:38-46.
[17] ROSENGAUS R B, ZECHER C N, SCHULTHEIS K F, et al.Disruption of the termite gut microbiota and its prolonged consequences for fitness[J].Applied and Environmental Microbiology, 2011, 77 (13) :4303-4312.
[18] MENKE S, WASIMUDDIN, MEIER M, et al.Oligotyping reveals differences between gut microbiomes of free-ranging sympatric Namibian carnivores (Acinonyx jubatus, Canis mesomelas) on a bacterial species-like level[J].Frontiers in Microbiology, 2014, 5:526.
[19] YEOMAN C J, CHIA N, YILDIRIM S, et al.Towards an evolutionary model of animal-associated microbiomes[J].Entropy, 2011, 13:570-594.
[20] VO A T.Determinants and implications of variation in the avian gut microbiota[D].Berkeley:Integrative Biology UC Berkeley, 2014.
[21] BENSON A K, KELLY S A, LEGGE R, et al.Individuality in gut microbiota composition is a complex polygenic trait shaped by multiple environmental and host genetic factors[J].Proceedings of the National Academy of Sciences of the United States of America, 2010, 107:18933-18938.
[22] SPOR A, KOREN O, LEY R.Unravelling the effects of the environment and host genotype on the gut microbiome[J].Nature Reviews Microbiology, 2011, 9:279-290.
[23] KOCH H, SCHMID-HEMPEL P.Gut microbiota instead of host genotype drive the specificity in the interaction of a natural host-parasite system[J].Ecology Letters, 2012, 15 (10) :1095-1103.
[24] BOLNICK D I, SNOWBERG L K, HIRSCH P E, et al.Individual diet has sex-dependent effects on vertebrate gut microbiota[J].Nature Communications, 2014, 5:4500.
[25] LEE W Y.Avian gut microbiota and behavioral studies[J].The Korean Journal of Ornithology, 2015, 22 (1) :1-11.
[26] MANTER D K, VIVANCO J M.Use of the ITS primers, ITS1Fand ITS4, to characterize fungal abundance and diversity in mixed-template samples by qPCR and length heterogeneity analysis[J].Journal of Microbiological Methods, 2007, 71:7-14.
[27] HUGERTH L W, MULLER E E L, HU Y O O, et al.Systematic design of 18SrRNA gene primers for determining eukaryotic diversity in microbial consortia[J].PLoS ONE, 2014, 9 (4) :e95567.
[28] HADZIAVDIC K, LEKANG K, LANZEN A, et al.Characterization of the 18SrRNA gene for designing universal eukaryote specific primers[J].PLoS ONE, 2014, 9 (2) :e87624.
[29] CLAUS S P, GUILLOU H, ELLERO-SIMATOS S.The gut microbiota:a major player in the toxicity of environmental pollutants?[J].npj Biofilms and Microbiomes, 2016, 2:16003.
[30] YANG Y, DENG Y, CAO L.Characterising the interspecific variations and convergence of gut microbiota in Anseriformes herbivores at wintering areas[J].Scientific Reports, 2016, 6:32655.
[31] ZHAO G H, ZHOU L Z, DONG Y Q, et al.The gut microbiome of hooded cranes (Grus monacha) wintering at Shengjin Lake, China[J].MicrobiologyOpen, 2017, 6 (3) :e447.
[32] ROTO S M, RUBINELLI P M, RICKE S C.An introduction to the avian gut microbiota and the effects of yeast-based prebiotic-type compounds as potential feed additives[J].Frontiers in Veterinary Science, 2015, 2:28.
[33] BARBOSA A, BALAGUV, VALERA F, et al.Age-related differences in the gastrointestinal microbiota of Chinstrap Penguins (Pygoscelis antarctica) [J].PLoS ONE, 2016, 11 (4) :e0153215.
[34] HIRD S M, SNCHEZ C, CARSTENS B C, et al.Comparative gut microbiota of 59neotropical bird species[J].Frontiers in Microbiology, 2015, 6:1403.
[35] CRISOL-MARTíNEZ E, MORENO-MOYANO L T, WILKINSON N, et al.A low dose of an organophosphate insecticide causes dysbiosis and sex-dependent responses in the intestinal microbiota of the Japanese quail (Coturnix japonica) [J].PeerJ, 2016, 4:e2002.
[36]刘蓓一.扬州鹅肠道微生物多样性及其受饲粮纤维水平的调控研究[D].扬州:扬州大学动物科学与技术学院, 2012.
[37] KNARREBORG A, SIMON M A, ENGBERG R M, et al.Effects of dietary fat source and subtherapeutic levels of antibiotic on the bacterial community in the ileum of broiler chickens at various ages[J].Applied and Environmental Microbiology, 2002, 68 (12) :5918-5924.
[38] SCUPHAM A J.Succession in the intestinal microbiota of preadolescent turkeys[J].FEMS Microbiology Ecology, 2007, 60:136-147.
[39] HIRD S M, SNCHEZ C, CARSTENS B C, et al.Comparative gut microbiota of 59neotropical bird species[J].Frontiers in Microbiology, 2015, 6:1403.
[40] RYU H, GROND K, VERHEIJEN B, et al.Intestinal microbiota and species diversity of Campylobacter and Helicobacter spp.in migrating shorebirds in Delaware Bay[J].Applied and Environmental Microbiology, 2014, 80 (6) :1838-1847.
[41]沈佳斌, 张雪敬, 吴蔚, 等.雉科鸟类肠道微生物的多样性与功能及其对食性的适应[J].南京师范大学学报 (自然科学版) , 2016, 39 (2) :90-95.
[42] YANG Y, DENG Y, CAO L.Characterising the interspecific variations and convergence of gut microbiota in Anseriformes herbivores at wintering areas[J].Scientific Reports, 2016, 6:32655.
[43] GAO G, ZHAO X, LI Q, et al.Genome and metagenome analyses reveal adaptive evolution of the host and interaction with the gut microbiota in the goose[J].Scientific Reports, 2016, 6:32961.
[44] GODOY-VITORINO F, GOLDFARB K C, KARAOZ U, et al.Comparative analyses of foregut and hindgut bacterial communities in hoatzins and cows[J].The ISME Journal, 2012, 6 (3) :531-541.
[45] ROGGENBUCK M, SCHNELL I B, BLOM N, et al.The microbiome of New World vultures[J].Nature Communications, 2014, 5:5498.
[46] WIENEMANN T, SCHMITT-WAGNER D, MEUSER K, et al.The bacterial microbiota in the ceca of Capercaillie (Tetrao urogallus) differs between wild and captive birds[J].Systematic and Applied Microbiology, 2011, 34 (7) :542-551.
[47] RUIZ-RODRGUEZ M, LUCAS F S, HEEB P, et al.Differences in intestinal microbiota between avian brood parasites and their hosts[J].Biological Journal of the Linnean Society, 2009, 96:406-414.
[48] CARON A, DE GARINE-WICHATITSKY M, GAIDET N, et al.Estimating dynamic risk factors for pathogen transmission using community-level bird census data at the wildlife/domestic interface[J].Ecology and Society, 2010, 15 (3) :25.
[49] GROND K, RYU H, BAKER A J, et al.Gastro-intestinal microbiota of two migratory shorebird species during spring migration staging in Delaware Bay, USA[J].Journal of Ornithology, 2014, 155:969-977.
[50] LOYE J E, ZUK M.Bird-parasite interactions:ecology, evolution and behaviour[M].New York:Oxford University Press/OUP USA, 1991.
[51] JOURDAIN E, GAUTHIER-CLERC M, BICOUT D J, et al.Bird migration routes and risk for pathogen dispersion into western Mediterranean wetlands[J].Emerging Infectious Diseases, 2007, 13 (3) :365-372.
[52] LEWIS W B, MOORE F R, WANG S A.Characterization of the gut microbiota of migratory passerines during stopover along the northern coast of the Gulf of Mexico[J].Journal of Avian Biology, 2016, 47 (5) :659-668.
鸟类对环境的变化非常敏感,环境的变化常常引起鸟类的变化,而鸟类的变化也可反映环境的变化.鸟类对湿地生态系统的结构和功能中起着重要的调控作用,因此湿地鸟类多样性及动态变化是评价湿地生态系统的重要内容之一.哈素海以特殊的地理环境和复杂多样的生...
讷河市位于黑龙江省西北部, 地处松嫩平原最北端, 与大、小兴安岭南缘相接, 在嫩江中游东岸, 124°18′50″~125°59′30″ E, 47°51′30″~48°56′16″ N。北靠嫩江县, 东邻五大连池市、克山县, 南接依安、富裕县, 西与内蒙古自治区莫力达瓦达斡尔族自治旗...