摘 要: 翻译延伸是核糖体将信使RNA (mRNA)蕴含的遗传信息解码为蛋白质的有序过程,是细胞维持基本代谢活动的核心步骤。多种人类疾病(如神经退行性疾病、癌症等)都与翻译延伸的异常有关。翻译延伸作为中心法则的关键步骤曾是现代分子生物学研究的重点内容,然而方法学上的限制却阻碍了对其动态过程以及调控规律的进一步研究。近年来,对翻译延伸调控相关方法的突破让与其相关的生命科学研究获得了长足的发展,尤其是近10年来的研究揭示了翻译延伸的复杂调控机理和多种生物学效应,为理解蛋白表达调控和疾病发生的关联提供了新的理论视角。本文在总结翻译延伸研究方法的基础上,重点探讨了顺式调控元件(mRNA与新生肽链序列)对局部翻译延伸速率的调控作用,同时列举了翻译延伸调控对模板mRNA和蛋白质产物功能的影响,包括mRNA稳定性、蛋白质的合成与降解、蛋白质亚细胞定位以及蛋白质共翻译折叠等,以期吸引生命科学各领域的学者共同参与翻译延伸领域的研究。
关键词: 翻译延伸; 核糖体; 核糖体印迹测序; 蛋白质共翻译折叠; 密码子使用偏好; RNA二级结构; 新生肽链;
Abstract: Proteins are biological macromolecules essential for cells to maintain their metabolic activities. Proteins are synthesized during translation elongation, a synergistic process in which ribosomes decode the genetic information transmitted in mRNA, using tRNA. Numerous human diseases, such as neurodegenerative diseases and cancers, are known to be related to abnormal translation elongation. Translation elongation, as one of the two critical steps for the central dogma, used to be the focus of research in molecular biology. However, limitations in methodology had hindered further investigations on the dynamic process of translation elongation and its regulation. Recently, breakthroughs in methodology have revived this research field. Studies in the past decade or so have revealed that, beyond simple decoding of genetic information in mRNA, translation elongation entails sophisticated regulatory mechanisms and multifaceted biological consequences; such insights have provided a novel theoretical framework for understanding the maintenance of protein homeostasis and the development of diseases. In this review, we summarize the most updated methods that can be used to investigate the processes of translation elongation and highlight the mechanisms by which mRNA and protein sequences modulate the local rate of translation elongation. We further enumerate the consequences of dysregulation in translation elongation, from various aspects such as mRNA stability, protein synthesis and degradation, protein subcellular localization,and co-translational protein folding. We anticipate that this review will serve to draw the attention of scholars in various research fields to participate in the study of translation elongation.
Keyword: translation elongation; ribosome; ribo-seq; co-translational protein folding; codon usage bias; RNA secondary structure; nascent polypeptide;
蛋白质是生命代谢最重要的有机大分子,是细胞功能的主要执行者。生物体内所有的蛋白质都是以信使RNA(mRNA)作为遗传信息的载体通过核糖体合成的,这一过程被称为翻译(translation)。翻译过程是分子生物学的重要研究对象,3位美国科学家Holley、Khorana和Nirenberg凭借对三联体核苷酸密码子(codon)的破译工作于1968年获得了诺贝尔生理学或医学奖。
翻译过程通常包括4个步骤:起始(initiation)、延伸(elongation)、终止(termination)和核糖体循环利用(recycling)。其中,翻译起始长久以来被认为是一条mRNA单位时间合成蛋白质数量的主要调节因素[1],受到翻译起始因子、Shine-Dalgarno(SD)序列等多方面的调控。然而近年来的研究表明,翻译延伸——核糖体从mRNA的5′端到3′端定向移动的同时将三联体核苷酸密码子的信息解码为氨基酸序列的过程——同样受到精细且严格的调控。翻译延伸的异常将引发mRNA的降解、错误的蛋白质亚细胞定位和折叠以及蛋白质的非生理性聚集[2],进而阻碍胚胎发育与神经系统的功能维持,研究显示这些影响与包括脆性X染色体综合征(fragile X syndrome)、神经退行性疾病(neurodegenerative diseases)和癌症在内的多种人类疾病的发生有关[3]。
翻译延伸的过程可以分解为3个过程:(1)解码过程——在核糖体中mRNA上的三联密码子被特定转运RNA(tRNA)上的反义密码子(anticodon)所识别;(2)肽键合成过程——将该tRNA上携带的氨基酸连接到肽链羧基端,同时核糖体构象发生改变;(3)移位过程——核糖体向mRNA的3′端移动一个密码子,并恢复至延伸初始构象[4]。具体而言,核糖体内部有3个可容纳tRNA的位点,从mRNA的5′端至3′端依次被称为核糖体E(Exit)位点、P(Peptidyl)位点和A(Acceptor)位点。其中,A位点用于接纳携带单个氨基酸的氨酰tRNA(aminoacyl-tRNA或aa-tRNA)并完成密码子识别;P位点用于装载携带多肽链的肽酰tRNA(peptidyl-tRNA);而脱酰基后的tRNA(deacylated tRNA)则通过E位点被核糖体释放。翻译延伸需要延伸因子(elongation factor,EF)的辅助。结合有高能分子GTP的延伸因子eEF-1(真核生物)、aEF-1(古细菌)或EF-TU(真细菌)携带氨酰tRNA进入核糖体的A位点;如果该氨酰tRNA的反义密码子可以与A位点处mRNA的密码子配对识别,则引发GTP水解,促使EF-1/EF-TU离开核糖体A位点;GTP水解提供的能量引起核糖体发生构象变化,使位于A位点的氨酰tRNA的3′端与位于P位点的肽酰基tRNA紧密接触;当两个tRNA同时完成A-P位点转移(A位点的tRNA部分移动到P位点)和P-E位点转移后,肽键合成发生,此时由刚移动到P位点的tRNA携带多肽链,并且在羧基端增加了一个氨基酸,而刚移动到E位点的tRNA则成为脱氨酰tRNA;接下来,另一个延伸因子eEF-2(真核生物)、aEF-2(古细菌)或EF-G(真细菌)进入A位点,并通过水解其携带的GTP分子将核糖体恢复至延伸初始构象(图1)。在上述过程的不断重复中,多肽链持续延伸,直到mRNA上的终止密码子进入核糖体的A位点为止。

虽然翻译延伸过程是上述核心步骤的循环往复,且在真核和原核生物间高度保守,但众多研究结果表明mRNA不同区域的解码速度并不恒定,在翻译过程中会存在核糖体在mRNA特定位置的短期暂停(pause)甚至中止(stall)等事件,暗示着翻译延伸过程受到了严格的调控[5]。本文将主要从研究方法、顺式调控机理和生物学效应3个角度介绍翻译延伸领域的主要研究进展。
图1 翻译延伸过程示意图
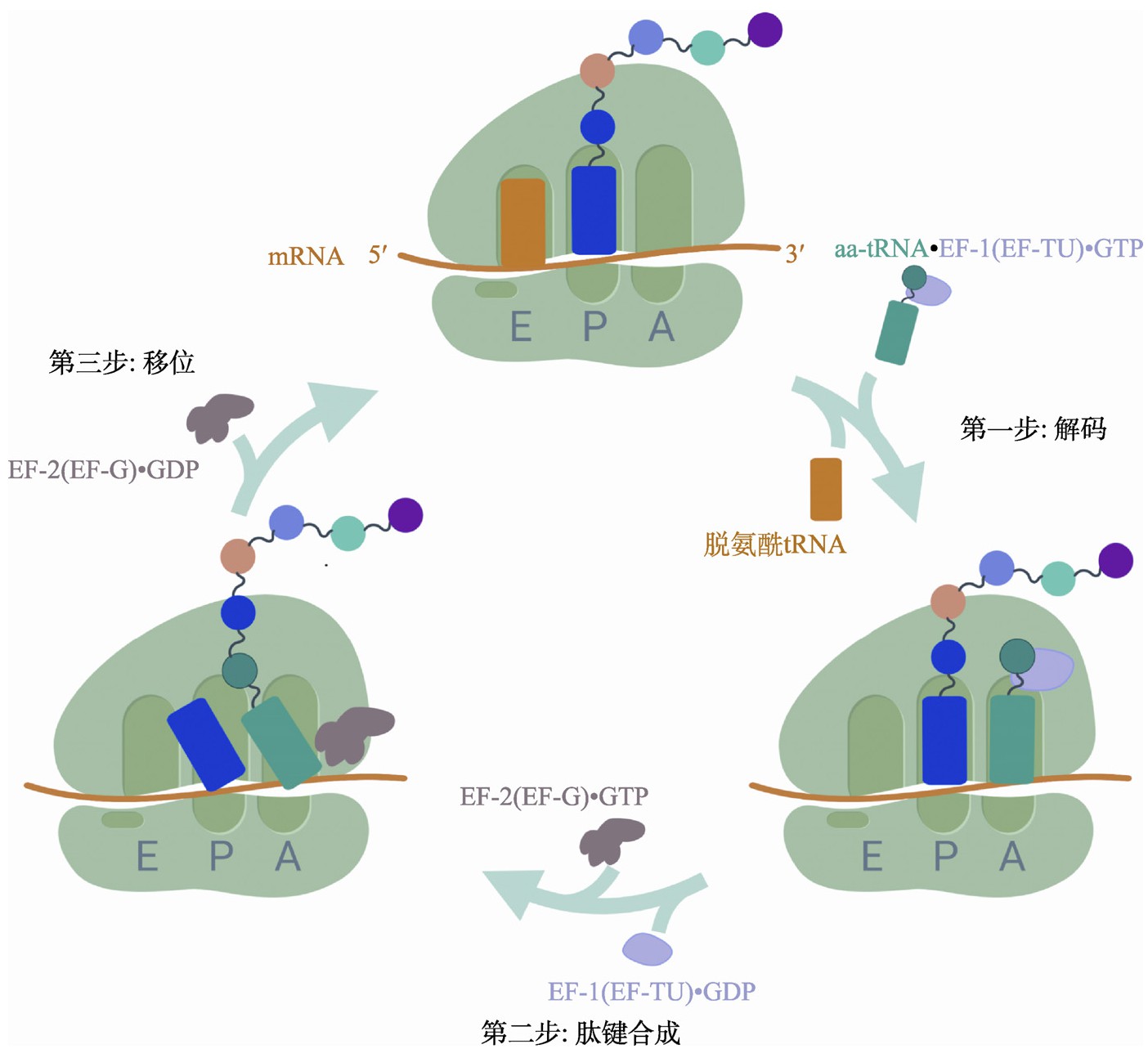
Fig.1 Schematic of the translation elongation cycle
第一步:解码过程。结合高能分子GTP的翻译延伸因子EF-1(EF-TU)携带氨酰tRNA(aa-tRNA)进入核糖体A位点进行密码子配对,E位点的脱氨酰tRNA离开核糖体。第二步:肽键合成过程。GTP水解引发核糖体的构象变化,使其内部两个tRNA紧密接触,肽键合成发生。第三步:移位过程。核糖体向mRNA的3′端移动一个密码子,并通过水解翻译延伸因子EF-2(EF-G)所携带的GTP分子提供能量,使核糖体恢复至初始构象。图制于Biorender.com。
1、 翻译延伸的研究方法
1.1、 核糖体结构解析
核糖体结构的解析是研究翻译过程的重要手段,主要包括X射线晶体解析法(X-ray crystallography)、核磁共振波谱测定法(nuclear magnetic resonance[NMR]spectroscopy)以及冷冻电镜技术(cryogenic electron microscopy,cryo-EM)3大类。X射线晶体解析法获得的核糖体结构分辨率最高(2.0~3.5?)[6],并且常常用于翻译延伸相关复合物的结构解析[7,8]。2009年,美国科学家Ramakrishnan、Steitz和以色列科学家Yonath以X射线晶体解析法为基础的核糖体结构研究获得了诺贝尔化学奖。X射线晶体解析法需要首先对核糖体进行结晶。然而,核糖体是大量蛋白质与多条RNA组成的大分子复合物,且在翻译延伸过程中存在一些能量不稳定的中间态构象,这些因素使得核糖体很难形成结晶。即使成功结晶,也会破坏构象的异质性。上述问题阻碍了生理状态下对核糖体结构的全面与快速解析。核磁共振波谱测定法也可对核糖体结构域[9]或者新生肽链的结构动力学[10]进行测定。尽管Nygaard等[11]通过固态NMR方法解析了大肠杆菌(Escherichia coli)完整核糖体的化学组成和分子间相互作用,但在大多数情况下NMR测定对象的大小局限于500 kDa范围内,不足以解析完整的原核核糖体(2000 kDa)或真核核糖体(3200 kDa)结构。最近10年来,伴随技术的突破,冷冻电镜对核糖体结构的分辨率已经可以与X射线晶体解析法相媲美[12,13](图2A)。冷冻电镜技术无需结晶,核糖体可保持其生理状态下的异质性特征与翻译延伸过程的中间状态,因此,在研究核糖体及其与翻译延伸因子等形成的大型复合物的结构时,发挥了越来越重要的作用[14,15,16,17]。
1.2、 生化动力学研究法
翻译延伸速率(特别是肽键形成的速率)可以通过生化动力学方法研究。这通常需要人工配制的体外翻译体系,该体系包含翻译过程所需的所有组分,并且可以通过放射性同位素或荧光对新生蛋白质产物进行标记和浓度测定。通过检测多个时间点的蛋白质合成量,可以绘制模型曲线,进而推算翻译延伸速率[18,19]。例如,Wohlgemuth等[20]为了研究不同氨基酸提供羧基时形成肽键的速率,在翻译体系中加入了嘌呤霉素(puromycin)(图2B)。嘌呤霉素可以作为氨酰tRNA类似物进入核糖体A位点与位于P位点的肽链发生类似肽键形成的生化反应,在反应结束后终止翻译延伸并释放翻译产物。该研究在嘌呤霉素浓度饱和时测定特定氨基酸与嘌呤霉素形成肽键的速率常数,发现氨基酸在提供羧基时的肽键形成速率不尽相同:(赖氨酸=精氨酸)>丙氨酸>丝氨酸>(苯丙氨酸=缬氨酸)>天冬氨酸>>脯氨酸。
1.3 、报告基因检测法
使用人工构建的报告基因载体,将待检测序列插入报告基因特定位置,则可以通过分析报告基因的表达水平估算插入序列对于翻译延伸的影响。例如,Chu等[21]采用荧光素酶(luciferase)报告系统,将待检测的序列插入到萤火虫荧光素酶(firefly-luciferase)的起始密码子下游,并将位于同一载体上且具有独立启动子的海肾荧光素酶(renilla-luciferase)作为内参,通过比较两种荧光信号的强度推测该待检测序列对翻译延伸速率的影响。
伴随着单分子荧光技术的发展,在活细胞中观察单条mRNA翻译动态的方法日趋成熟[22]。单分子翻译检测技术可将人为设计的报告mRNA锚定在细胞膜结构上,并采用不同的荧光分子分别标记mRNA和新生肽链,在较长的时间范围内持续记录翻译延伸的进程[23,24,25]。例如,Yan等[23]通过抑制剂阻断翻译起始,并统计单位时间内从mRNA上释放的带有荧光标记的核糖体的数目,该数目越大则意味着翻译延伸越快。针对mRNA序列进行修改,则可以进一步探究翻译延伸速率的顺式调控因素(图2C)。该方法目前的局限性在于只适用于对单一基因翻译过图2翻译延伸的研究方法程的逐个研究,尚无法推广到高通量研究之中。
图2 翻译延伸的研究方法
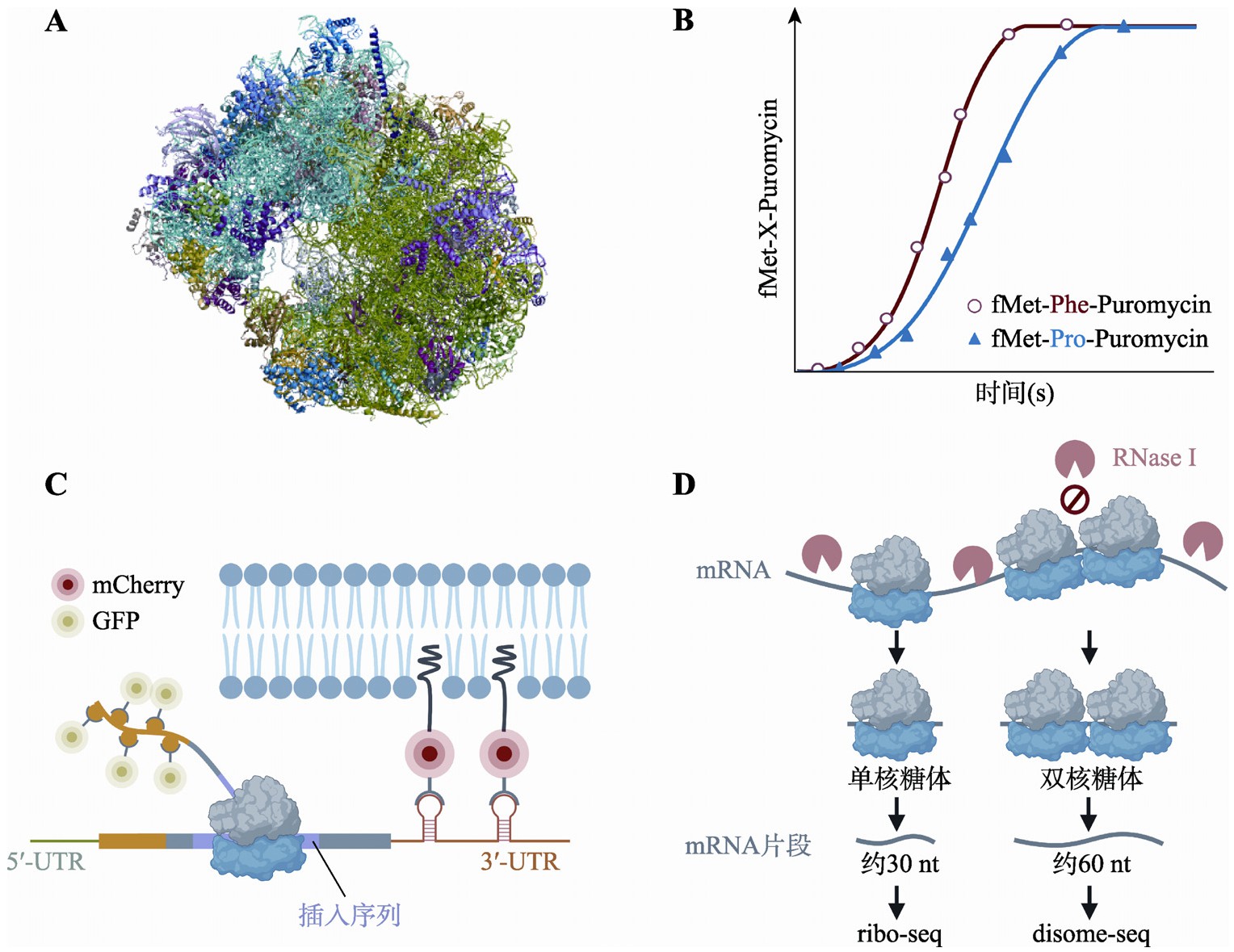
Fig.2 Methods for understanding translation elongation
A:酿酒酵母80S核糖体冷冻电镜解析模型(http://www.rcsb.org/structure/6SNT)。B:肽键形成生化动力曲线示意图。fMet:甲酰甲硫氨酸,Puromycin:嘌呤霉素,X:待检测氨基酸。C:单分子mRNA动态翻译检测示意图。D:ribo-seq与disome-seq实验流程示意图。图制于Biorender.com。
1.4 、核糖体印迹测序(ribo-seq)法
在全基因组范围研究翻译调控可以通过ribo-seq实现。早在20世纪60年代,Steitz[26]就研发出通过密度梯度离心按照核糖体结合数目分离核糖体-mRNA复合物的实验技术(polysome profiling),用于检测mRNA的翻译状态。伴随着高通量测序技术的迅猛发展,polysome profiling技术与大规模测序技术的有力结合诞生出了ribo-seq技术。此技术采用RNA酶处理核糖体与mRNA的复合物,将因被核糖体保护而无法被RNA酶降解的mRNA片段用于高通量测序的文库构建和序列信息读取(图2D)。该技术可以实现对某一时刻细胞内所有转录本上核糖体的位置信息与状态信息的高通量捕获,并可达到单碱基的分辨率[27],因此,在内源基因翻译延伸调控的研究领域具有得天独厚的优势。除ribo-seq技术外,Pelechano等[28]建立的5PSeq技术也可以在单碱基分辨率下高通量捕获核糖体的位置信息。这一技术的开发基于mRNA共翻译降解的原理——mRNA在5?脱帽后由5?外切酶自5?端至3?端逐个碱基降解,由于外切速率大于翻译延伸速率,5?外切酶紧跟正在进行最后一轮翻译的核糖体行使其降解功能。因此,对无帽mRNA的5?端碱基进行测序即可获得这些核糖体的位置信息。上述高通量技术已成为测定翻译延伸速率和检测翻译延伸迟滞事件的重要手段[29,30]。
如果一个基因内部的翻译延伸速率无差异,则核糖体应该大致均匀分布于转录本编码区的各个区域。反之,如果检测到核糖体在某段编码区上严重堆积,则提示该区段存在翻译延伸的迟滞。根据这一原理,通过ribo-seq实验以及后续数据分析可以推断核糖体停滞的位置[31]。进一步的工作针对riboseq数据创立了算法,可以系统性地检测翻译延伸迟滞的顺式调控因素,并将其应用于估算61个编码氨基酸的密码子各自的解码时间[32]。
需要特别注意的是,为了防止在样品制备过程中核糖体在mRNA上的进一步延伸,往往需要在核糖体提取的试剂中加入翻译延伸抑制剂——放线菌酮(cycloheximide)。早期酿酒酵母(Saccharomyces cerevisiae)的ribo-seq实验通常还包含细胞与放线菌酮在室温下共孵育的步骤[27]。后续研究发现,室温下的放线菌酮处理并不能完全阻止翻译延伸过程,而且会对不同密码子产生不同的抑制作用,从而影响延伸速率的测量[33,34]。因此,在使用ribo-seq研究翻译延伸迟滞事件时应尽量避免细胞与放线菌酮在室温下的共孵育。
当一个核糖体发生翻译延伸迟滞时,如果其上游正常延伸的核糖体距离不远,则可能与其发生碰撞(ribosome collision)并在mRNA上形成串联的双核糖体结构。因此,该结构可以更为特异地反映翻译延伸迟滞事件的发生。基于这一想法,一些研究工作通过改造ribo-seq实验体系,建立了串联双核糖体(disome)的检测技术disome-seq(图2D),并成功地在细胞内捕获到了翻译延伸迟滞的信号。例如在缺失亮氨酸和丝氨酸的培养基中生长的大肠杆菌内,可在编码亮氨酸与丝氨酸的密码子处观察到大量串联双核糖体的富集[35]。同样,当酿酒酵母的组氨酸合成被抑制时,可在编码组氨酸的密码子处观察到串联双核糖体信号[36]。
ribo-seq可以用于研究同一基因不同区域的翻译延伸速率,然而这一方法在研究不同基因的翻译延伸速率时会失效。其原因是,不同基因上核糖体的分布除了由其延伸速率决定,也受到翻译起始速率的影响。特别值得注意的是,在计算中通常使用的对mRNA水平的均一化并不能解决这个问题。从ribo-seq数据中拆分翻译起始速率和延伸速率在计算上仍然存在一定困难,因此仅有少量研究工作通过实验方法系统地比较了不同基因的翻译延伸速率。例如,Ingolia等[31]在小鼠(Mus musculus)的胚胎干细胞培养液中添加了翻译起始抑制剂用以阻断核糖体在mRNA上的持续起始,在之后的多个时间点分别进行ribo-seq分析,进而通过测定核糖体单位时间内从mRNA上的释放数目来估算翻译延伸速率。此外,将ribo-seq与蛋白质组联合分析可估算翻译起始速率,进而间接推算翻译延伸速率[37]。
2 、翻译延伸的调控机理
顺式和反式因子均对翻译延伸有重要的调控作用。由于反式因子如翻译延伸因子[38,39,40]、small RNA[41,42]等对翻译延伸的调控作用已有较详细的总结和讨论,下文主要从顺式元件角度出发,着重介绍mRNA与氨基酸序列对翻译延伸速率的调控机理。
2.1、 mRNA序列对翻译延伸的调控作用
2.1.1、 同义密码子使用对翻译延伸的调控作用
在大多数生物中,20种氨基酸由61种密码子编码。其中,18种氨基酸由不止一种密码子编码。人们将编码同一氨基酸的不同密码子称为同义密码子(synonymous codon)。研究发现,不同物种之间、同一个物种的不同基因之间以及同一个基因的不同区域之间,同义密码子使用频率均存在差异[43,44,45,46]。一个基因组内部基因之间同义密码子差异使用的现象被称为密码子使用偏好性(codon usage bias)[46,47]。高表达基因倾向于使用的同义密码子被称为偏好密码子(preferred codon),其他的被称为稀有密码子(rare codon)。
密码子的使用偏好参与了翻译调控。一些学者认为偏好密码子具有更高的解码准确性[48,49]。而以Ikemura为代表的另一派学者则认为偏好密码子具有更高的翻译延伸速率(图3A),因此获得更高的核糖体使用效率[44,50,51]。该“效率”假说指出,在细胞中偏好密码子通常对应含量充足的同工tRNA(cognate tRNA),而识别稀有密码子的同工tRNA则拷贝数较低且含量稀少[3,52]。当稀有密码子出现在核糖体A位点时,由于其对应的氨酰tRNA在细胞内浓度低,核糖体需要花费更长的时间才能完成tRNA的识别与装载。因此,稀有密码子具有更低的翻译延伸速率[53]。
Curran和Yarus[54]巧妙地通过lacZ和RF-2的非同框融合基因报告系统检测了29个YNN(N代表A、C、G、T四个碱基之一,Y代表C或T)密码子的识别效率。RF-2的基因内部存在滑动序列(slippery sequence),可以促进核糖体以一定概率在特定位点发生单碱基移位读码。Curran和Yarus将该滑动序列(23个碱基)下游连接YNN密码子,并整体插入lacZ基因的上游。只有当在该滑动序列上发生向3′端的单碱基核糖体移码时,lacZ基因才能在正确的开放阅读框中翻译。两位作者假定核糖体的移位读码在单位时间内以一定的概率发生,且与正常的翻译延伸构成竞争。因此,若密码子YNN被快速识别,则移位读码不会发生,报告基因lacZ无法产生功能蛋白;当密码子YNN识别较慢时,则发生核糖体移码的概率上升,从而合成出正确的lacZ蛋白。他们发现偏好密码子的识别速率显着快于稀有密码子,且密码子之间的识别速率差异可高达25倍。S?rensen等[55]也发现在lacZ基因内插入偏好密码子检测到的翻译延伸效率比插入稀有密码子的版本快6倍。需要注意的是,上述研究均使用了少数报告基因,因此统计学上无法完全排除mRNA二级结构、碱基GC含量以及特定基序等干扰因素。通过对ribo-seq数据进行分析则可以从海量内源基因中归纳密码子使用偏好对翻译延伸的调控作用。Qian等[32]开创了依据ribo-seq数据演算每个密码子翻译延伸速率的先河,发现了同义密码子与同工tRNA的平衡使用可以避免核糖体的闲置并促进细胞整体的蛋白质合成效率。在认识到放线菌酮共孵育所带来的影响后,采用速冻法(flash frozen)产生的ribo-seq数据定性地支持了前人报告基因的研究结果——即偏好密码子的使用可提高翻译延伸速率。然而,同义突变对翻译延伸速率的影响在数值上仍然存在分歧意见[33,34,56]。
图3 翻译延伸的调控机理与生物学效应
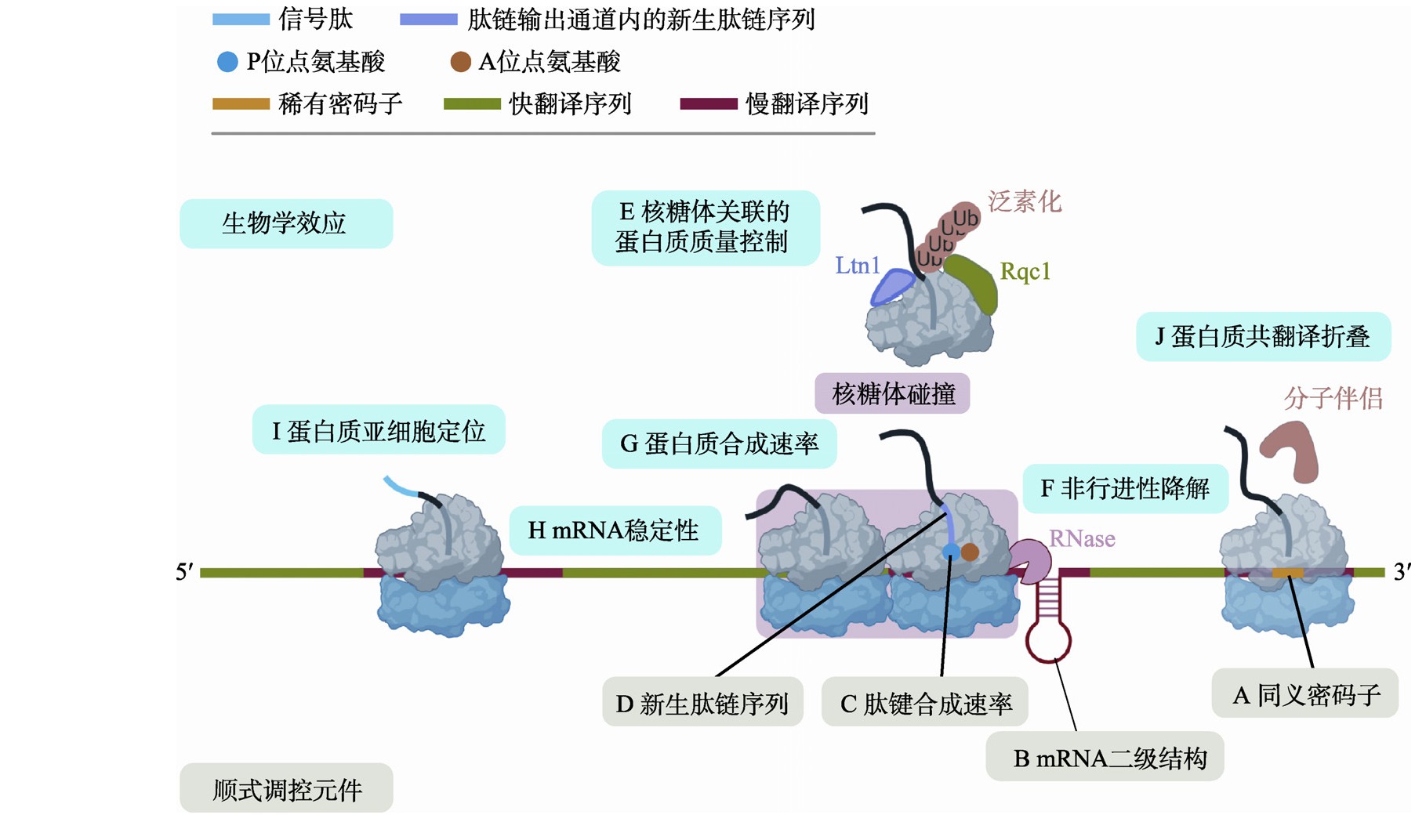
Fig.3 Regulatory mechanisms and biological effects of translation elongation
A:同义密码子使用对翻译延伸的调控作用。当位于核糖体A site的是稀有密码子(黄色线段)时,由于其对应的同工tRNA浓度较低,因此在该区域翻译延伸速率变慢(红色线段)。B:mRNA二级结构对翻译延伸的调控。位于编码区的mRNA二级结构抑制翻译延伸。C:肽键合成速率对翻译延伸的调控。氨基酸可以通过与PTC的相互作用影响肽键合成速率。D:新生肽链序列对翻译延伸的调控。新生肽链序列可通过与核糖体肽链输出通道相互作用调控翻译延伸速率。E:核糖体关联的蛋白质质量控制(RQC)。由于翻译延伸中止导致的串联双核糖体可被E3连接酶识别并进行泛素化修饰,从而引发核糖体大小亚基解离和RQC过程。在RQC过程中,已解离的核糖体大亚基中的新生肽链在E3连接酶Ltn1和Rqc1蛋白的协同作用下被泛素化修饰。F:非行进性降解(NGD)。内切酶切割串联双核糖体结合处的mRNA序列,残余的mRNA片段则被降解。G:调控蛋白质合成速率。翻译延伸的调控(如同义密码子的使用)对蛋白质合成速率具有重要影响。H:调控mRNA稳定性。含有稀有密码子的mRNA倾向于具有更低的稳定性。I:调控蛋白质亚细胞定位。分泌蛋白氨基端的信号序列(蓝色线段)被翻译完成后的翻译暂停会促进其与信号肽识别因子的结合而实现正确的亚细胞定位。J:调控蛋白质共翻译折叠。同义密码子的使用和mRNA二级结构可通过影响翻译延伸速率调控蛋白质折叠。另外,核糖体关联分子伴侣与新生肽链的结合也可以辅助蛋白质的共翻译折叠。图制于Biorender.com。
2.1.2、 mRNA二级结构对翻译延伸的调控作用
RNA二级结构是指RNA分子通过碱基互补配对而形成的茎环(stem loop)或假结(pseudoknot)等结构。位于mRNA 5′-UTR区的二级结构可以阻碍核糖体小亚基对起始密码子的扫描,而位于3′-UTR区的二级结构则可能削弱小RNA介导的翻译抑制[57,58]。由于翻译延伸过程消耗GTP中高能磷酸键的能量,部分学者认为核糖体及其结合蛋白可以有效打开位于编码区的mRNA二级结构[59]。
然而,一些报道又显示位于编码区的mRNA二级结构可能具有翻译延伸抑制效应(图3B)。例如,酿酒酵母ASH1基因编码调控交配型转换的转录因子,该蛋白质在出芽繁殖时主要定位于子细胞而非母细胞中,研究发现这种蛋白质的极性定位是通过ASH1编码区的mRNA二级结构抑制该基因位于母细胞中的转录本的翻译实现的[60]。另外,单分子光镊技术检测单核糖体在mRNA上移动速率的结果也显示,稳定的mRNA二级结构可以导致核糖体的延伸暂停[61]。基于ribo-seq的高通量数据分析结果则显示,位于核糖体入口处的mRNA二级结构对延伸速率的影响最为明显[37];且高表达基因倾向于具有更高的mRNA二级结构,可以通过降低翻译延伸速率的方式确保翻译的准确性[37]。还有研究指出,mRNA二级结构倾向存在于编码蛋白质结构域的连接区(protein domain junction)或无序区(disordered region)[62,63,64]。由此Tang等[63]提出假说,当核糖体行进至这些区域时,其延伸速率会被mRNA二级结构下调,确保新生肽链有充分的时间实现蛋白质结构域的正确折叠。
在病毒中,mRNA二级结构常常与滑动序列成对出现。当核糖体的延伸被mRNA茎环或假结等二级结构阻拦后会滞留在上游紧邻的滑动序列区,并因此有一定概率在此区域发生核糖体的移位读码,进而将新的读码框延伸到下游序列[65]。这种发生在固定位置的核糖体移位读码是程序性的,借此,病毒可使用有限的基因组编码更多种类的蛋白质[66]。导致新冠肺炎(COVID-19)疫情的新冠病毒(SARS-CoV-2),在其第一个开放阅读框(ORF1ab)中就存在一个程序性核糖体移位读码位点[67],以此维持该基因编码的pp1a和pp1b两段多肽的剂量平衡。此外,也有报道显示在原核与真核生物的基因组中存在程序性核糖体移位读码事件[38,68]。上述发现从另一角度表明了一些mRNA二级结构对翻译延伸的抑制作用。
但也有研究结果指出大多数mRNA二级结构对翻译延伸的影响力可能较弱。例如,大肠杆菌的体内mRNA二级结构谱图分析显示,mRNA二级结构对翻译的调控作用似乎仅局限于翻译起始过程,对翻译延伸效率几乎没有影响[69]。对热激处理水稻(Oryza sativa)的mRNA二级结构与ribo-seq谱图的比较分析结果也表明,mRNA二级结构的改变与热诱导的翻译水平变化无显着相关性[70]。Beaudoin等[58]甚至提出了“因果互换”的新假说:翻译过程中的核糖体塑造了mRNA的二级结构,而非mRNA二级结构指导了核糖体的翻译延伸。上述各类研究结果的矛盾之处使得mRNA二级结构在翻译延伸过程中的调控功能显得扑朔迷离,其相关机理亟待进一步的分析与挖掘。
2.2、 氨基酸序列对翻译延伸的调控作用
除mRNA序列外,氨基酸序列——位于核糖体A位点和P位点的氨基酸之间的肽键形成以及位于核糖体肽链输出通道(exit tunnel)内的新生肽链序列——也是调控翻译延伸速率的重要因素。
2.2.1、 肽键形成速率对翻译延伸的调控作用
由于氨基酸侧链的空间结构和化学性质存在差异,其形成肽键的速率各不相同(图3C)。脯氨酸是20种生物体主要氨基酸中最为特殊的一种,其氨基(-NH2)与侧链成环后仅存余亚氨基(-NH-)结构,因此准确地说是亚氨基酸。这使得其在位于核糖体A位点处时成为肽键形成的弱受体[20,71,72]。近期,A位点含有脯氨酰-tRNA类似物的核糖体结构进一步显示,脯氨酸作为底物在肽基转移酶中心(peptidyl transferase center,PTC)内处于不利的空间位置[73]。此外,不利的空间位置也使得位于核糖体P位点处的脯氨酸成为肽键形成的弱供体[74]。由于脯氨酸既是肽键形成的弱供体也是弱受体,当其串联出现时,对翻译延伸的抑制作用尤为明显[74,75,76,77,78,79]。
除脯氨酸外,另一个推测可以降低肽键形成速率的氨基酸是甘氨酸[80]。大肠杆菌的ribo-seq结果显示富含甘氨酸的三肽在三肽组合中具有最强的翻译延伸抑制作用[77]。此外,对小鼠ribo-seq数据的分析也发现,在核糖体P位点富集的氨基酸中,甘氨酸紧随脯氨酸之后位列第二,暗示了其对翻译延伸的抑制作用[31]。
2.2.2 、新生肽链序列对翻译延伸的调控作用
阻碍肽键形成的氨基酸通过与核糖体肽基转移酶中心区域的相互作用来抑制翻译延伸,而某些新生肽链序列则可以通过与核糖体肽链输出通道的相互作用降低翻译延伸速率(图3D)[81,82]。
肽链输出通道是核糖体内部紧邻P位点的一段狭长的管道结构,此管道不同位置的内径并不一致。其中内径最小的区段被称作狭窄段(constriction site),研究者认为其可通过阻碍特定新生肽链的通过而抑制翻译延伸。例如在一个被广泛研究的细菌案例中,SecM蛋白包含一段长度为17个氨基酸的F150XXXXWIXXXXGIRAGP166(X代表可变的氨基酸)序列[83]。此序列作为新生肽链通过核糖体的肽链输出通道会导致核糖体的暂停,此时G165占据在P位点的位置[84]。此暂停可引发mRNA二级结构的调整,其下游secA基因本来位于茎环内的翻译起始位点因此暴露出来,从而促进SecA蛋白的合成[82]。在之后的研究中,越来越多的细菌多肽序列被发现对翻译延伸具有抑制作用。例如,通过遗传筛选的方法,FXXYXIWPP等肽段被鉴定为翻译延伸的抑制信号[77,85];通过生物信息学分析,一系列在蛋白质组中低频出现的短肽段也被发现具有抑制翻译延伸的作用[86]。
也有观点认为核糖体的肽链输出通道是通过电势作用来调控翻译延伸的。有报道显示,肽链输出通道内壁的静电势为负值[87],当富含正电荷氨基酸(例如精氨酸和赖氨酸)的新生肽链通过带负电的肽链输出通道时,因电荷的异性相吸作用,抑制了核糖体的延伸[88]。例如,基于分子卷尺(molecular tape measure)的体外实验表明,含连续精氨酸或赖氨酸的肽链在完全通过肽链输出通道之前会引发核糖体暂停[88]。大肠杆菌和酿酒酵母的体内实验结果也显示,在荧光素酶报告基因的5′编码区插入连续的赖氨酸密码子(AAG或AAA)可显着下调报告基因的蛋白质合成效率[89]。在基因组水平上,Charneski和Hurst[90]对酿酒酵母ribo-seq数据的分析显示,新生肽链中的正电荷氨基酸是核糖体翻译延伸速率的主要抑制因素;且他们发现正电荷氨基酸对翻译延伸的抑制作用具有累加效应,即正电荷氨基酸密度越高则核糖体停滞现象越明显。然而,也有实验室对该结果提出了不同的看法。Artieri和Fraser[91]指出,Charneski和Hurst的报道可能是数据的低覆盖度导致的假阳性结果。Sabi和Tuller[92]对9个物种的ribo-seq数据进行了联合分析,却仅在3个物种中检测到了正电荷氨基酸对翻译延伸的抑制作用,而他们还在5个物种中发现负电荷氨基酸也可抑制翻译延伸。因此,肽链输出通道中新生肽链是否可通过电荷相互作用调控翻译延伸尚存在争议,相关机理有待进一步的分析与探讨。
3、 翻译延伸的生物学效应
诸多情况都可能导致核糖体无法继续行进,例如细胞处于应激状态下,在转录加工错误或化学损伤的mRNA上,或当翻译终止错误造成核糖体行进至3′-UTR甚至poly(A)尾时。此时,滞留的核糖体将激活细胞内的质量监控系统,降解模板mRNA和翻译中间产物[93]。然而,相对于这些不可逆的翻译延伸中止事件,细胞内更普遍存在的可能是那些核糖体可以恢复行进的翻译延伸暂停事件。这些翻译暂停事件通常被认为是程序性的且具有生物适应性意义[5],参与调控多种分子生物学过程,如蛋白质表达水平、mRNA稳定性、蛋白质亚细胞定位以及蛋白质折叠[3,94]。
3.1、 翻译延伸中止的生物学效应
当核糖体翻译延伸中止时,细胞会启动核糖体关联的蛋白质质量控制(ribosome-associated protein quality control,RQC),通过特定调控通路降解潜在有害的新生肽链;并同时启动非行进性降解(no-go decay,NGD),特异性地降解发生翻译延伸中止的mRNA[93]。
3.1.1、 核糖体关联的蛋白质质量控制RQC
延伸中止的核糖体可能会与位于其同一mRNA上游的核糖体发生“碰撞”或“追尾”,形成串联双核糖体结构。近期的研究结果表明,串联双核糖体可能是引发RQC的关键结构单元(图3E)。冷冻电镜结果显示,组成串联双核糖体的两个核糖体40S小亚基之间紧密接触,为E3泛素连接酶Hel2提供了良好的底物识别平台。在酿酒酵母中,Hel2蛋白可在核糖体40S小亚基上添加赖氨酸连接的多聚泛素化修饰[95,96]。泛素化修饰的核糖体可被RQC触发复合体(RQC-triggering complex)识别,并导致大小亚基的解离与RQC过程[95,97]。在RQC过程中,残留于核糖体60S大亚基中的新生肽链在E3泛素连接酶Ltn1和Rqc1蛋白的协同作用下被泛素化修饰(图3E)[97]。同时,Rqc2蛋白会在该多肽链的羧基端添加多聚丙氨酰和三酰残基尾(CAT-tailing)[98]。最终泛素化的多肽链被Vms1从核糖体大亚基中释放[99,100],并由Cdc48蛋白携带进入蛋白酶体(proteasome)降解[97,101,102]。
3.1.2、 非行进性降解NGD
串联双核糖体在引发RQC的同时还会引发NGD。在NGD过程中,被泛素化修饰的核糖体40S小亚基可诱发细胞内某尚未报道的内切酶在串联双核糖体结合的mRNA序列附近进行切割(图3F)[103]。之后,位于切割断点5′一侧的核糖体将被Dom34-Hbs1蛋白质复合体释放[104,105,106],残余的5′-NGD mRNA片段则被Ski复合体和外切酶体(exosome)降解;而位于切割断点3′一侧的停滞核糖体可能被释放,也可能重新启动完成完整的翻译过程。之后,残余的3′-NGD mRNA片段由Xrn1外切酶降解[5]。
值得注意的是,迄今为止被观察到的NGD和RQC事件大部分来自于含有人为设计的翻译延伸中止序列的报告系统或处于恶劣环境条件下的细胞中。例如,Doma等[103]在酿酒酵母中首次发现NGD途径时使用的是含有稳定茎环结构的PGK1-SL报告载体。此后,含有串联多聚腺苷酸[107,108,109]、串联精氨酸稀有密码子CGA[110]、多聚正电氨基酸[97]以及被氧化的mRNA[111]等可严重阻碍翻译延伸的特定序列也被广泛应用于人工报告系统研究NGD和RQC。此外,在氨基酸饥饿[36]、tRNA缺乏[112]和氧化胁迫[111]等条件下也可检测到NGD或RQC现象。在正常生长条件下的细胞内源基因上检测到的NGD或RQC事件较为少见。造成这一结果的原因可能是:在正常生长的细胞中,发生翻译延伸中止的mRNA比例较低,且其蛋白质中间产物和mRNA又会被NGD和RQC途径迅速识别降解,以目前检测手段的灵敏度尚无法及时捕获这些中间产物。通过实验方法鉴定在正常生长条件下NGD或RQC的目标内源基因,需要人为提高细胞内发生翻译延伸中止但尚未完全被NGD途径降解的mRNA比例。敲除或者敲降这两条调控通路关键的调控蛋白(如Hel2、Dom34和Xrn1等)从而减缓相关过程可能是实现这一目标的有效手段[111]。
3.2、 翻译延伸暂停的生物学效应
在正常生长的细胞中,ribo-seq实验观察到了大量翻译过程中核糖体的堆积位点,暗示了翻译延伸迟滞事件的广泛存在性[31]。如果这些核糖体以及被其翻译的mRNA均进入降解途径,将会是极大的细胞资源浪费并会引发严重的细胞代谢紊乱。例如,有约10%的真核生物蛋白质含有多聚脯氨酸序列[5],如果这10%的蛋白质均因多聚脯氨酸序列对翻译延伸的抑制作用而发生翻译中止并且被降解,对细胞将是巨大的伤害,在这样的情况下这些多聚脯氨酸序列理应在进化中被自然选择淘汰。事实上,多聚脯氨酸序列对翻译延伸的抑制作用可被翻译延伸因子eIF5A(尽管从名字看是翻译起始因子)所缓解[76,113]:在暂停后,核糖体可沿mRNA继续行进,翻译出完整的蛋白质。我们将这种现象与翻译延伸中止相区分,称之为翻译延伸暂停。翻译延伸暂停事件在细胞内的普遍存在暗示其具有功能性调控作用,下文将分别从蛋白质表达水平、mRNA稳定性、蛋白质亚细胞定位和蛋白质折叠等研究较多的4个方面详细探讨其生物学效应。
3.2.1 、调控蛋白质表达水平
翻译起始一般被认为是翻译的限速步骤,其速率直接影响蛋白质合成速率[23,114]。然而越来越多的证据表明,翻译延伸速率对蛋白质产量也具有重要的调控作用(图3G)。例如Carlini等[115]改造了果蝇(Drosophila melanogaster)乙醇脱氢酶基因(ADH),将它的1个、6个或10个偏好密码子替换为稀有密码子,体内实验显示稀有密码子的使用降低了ADH蛋白的水平。反之亦然,将人类(Homo sapiens)HEK-HT细胞中的原癌基因KRAS编码区的稀有密码子替换为偏好密码子之后,突变体中的KRas蛋白的表达水平上调,移植瘤生长实验显示肿瘤增大[116]。Chu等[21]的研究工作结合报告基因和计算模拟的手段,系统性地探讨了翻译起始与延伸对蛋白质合成速率的协同调控作用。该研究将报告基因分别设计为全部使用偏好密码子、使用正常配比的同义密码子、以及全部使用稀有密码子3个版本。实验结果显示,当基因具有高翻译起始速率时,偏好密码子版本的蛋白质产量明显高于稀有密码子版本;而当基因具有低翻译起始速率时,3个版本的蛋白质产量则无显着差异。这一研究结果表明,只有当核糖体可以在起始密码子下游快速延伸的时候,高的翻译起始速率才可能获得高丰度蛋白质产物;否则,与高起始速率不相匹配的缓慢的翻译延伸将成为限速步骤。更为有趣的是,还有研究表明,酿酒酵母在胁迫条件下可以通过改变特定tRNA的浓度,调控同义密码子的翻译延伸速率,选择性地加速合成响应该胁迫条件的蛋白质,促进环境适应[117]。
另外,Zhao等[118]发现拟南芥(Arabidopsis thaliana)mRNA的poly(A)尾中含有非腺嘌呤核苷酸(C、G和T),而且腺嘌呤纯度(即一条mRNA poly(A)尾中腺嘌呤所占poly(A)尾全长的比例)的下降可以抑制蛋白质的合成。这可能是因为腺嘌呤纯度较高的poly(A)尾可以通过5′端帽子(5′-cap)结合蛋白(eIF4E)、脚手架蛋白(eIF4G)和poly(A)结合蛋白(PABP)构成的蛋白质复合体促进mRNA的首尾成环(cap-to-tail looping),提升翻译结束后被释放的核糖体重启翻译的回收效率,进而提高翻译起始速率和蛋白质合成速率。
3.2.2、 调控mRNA稳定性
翻译延伸速率不仅可影响蛋白质表达量,还有报道显示其对mRNA水平的调控:同义密码子的使用可直接调控mRNA的稳定性(图3H)。例如,Chen等[119]通过构建报告基因的上千个同义突变体发现,含有偏好密码子的基因倾向于具有更高的mRNA稳定性和表达水平;在排除了mRNA二级结构与GC含量等干扰因素的影响后依然如此。Schikora-Tamarit和Carey[120]认为这是一种细胞识别“移码”转录本并将其降解的机制,因为阅读框移码后偏好密码子的使用可能会减少。Espinar等[121]进一步发现组成型表达的基因的mRNA水平受密码子偏好性的影响更大,其机制与RNA解旋酶Dbp2有关。其他研究组的工作将密码子使用偏好性、mRNA稳定性和翻译调控3方面进行联系:在斑马鱼(Danio rerio)和爪蟾(Xenopus)的卵母细胞向合子的转变过程中,富含偏好密码子的基因具有更高的mRNA稳定性、更长的poly(A)尾和更高的翻译效率[122];酿酒酵母中的研究结果则表明,偏好密码子在具有更快的翻译延伸速率的同时,也具有更高的mRNA稳定性[123],而这一偶联是由Dhh1蛋白介导的[124]。另一个斑马鱼中的研究工作更是直接指出,稀有密码子介导的mRNA降解过程是依赖于mRNA翻译的——当人为阻断翻译起始时,富含稀有密码子的mRNA不再被快速降解[125]。
3.2.3 、调控蛋白质亚细胞定位
翻译延伸还可以调控蛋白质的亚细胞定位(图3I)。定位于内质网的分泌蛋白通常在其氨基端具有5~30个氨基酸的信号肽序列(signal peptide),当此序列被翻译完成后会与信号肽识别因子(signal recognition particle,SRP)结合,与此同时信号肽识别因子会将自身插入核糖体A位点,通过阻止氨酰tRNA进入的方式暂停翻译延伸,直至翻译暂停的mRNA-核糖体复合物正确定位至内质网后,翻译延伸才重新恢复[126,127]。一些学者认为神经退行性疾病亨廷顿综合征(Huntington’s disease,HD)是由翻译延伸调控紊乱引发的蛋白质错误定位导致的。亨廷顿综合征的致病蛋白Htt的第一个外显子从氨基端起依次编码17个氨基酸的定位信号肽(N17)、19个谷氨酰胺的串联序列以及长度为38个氨基酸的富脯氨酸序列[128],其中谷氨酰胺串联序列的加长突变(≥35个氨基酸)可引发疾病[129,130,131]。在正常的Htt中,脯氨酸序列位于信号肽N17序列下游的30~57个氨基酸的最佳位置来暂停翻译延伸,此时N17刚刚从核糖体肽链输出通道中暴露出来,为信号肽识别因子提供了最优结合肽链长度和充足的识别时间,使得Htt蛋白可以在信号肽识别因子的帮助下正确定位于高尔基体、内质网或线粒体[132];而在突变的Htt中,随着N17序列从核糖体肽链输出通道出现,其后方的加长谷氨酰胺区域将被快速翻译(速率比脯氨酸快2~6倍)[71,91],因此信号肽识别因子将没有足够的时间与N17结合,进而导致突变的Htt错误地聚集于细胞质并引发疾病[133]。
3.2.4 、调控蛋白质折叠
新生肽链的正确折叠对蛋白质正常执行其分子功能至关重要,而蛋白质的错误折叠和聚集则可导致多种严重的人类疾病,如阿尔茨海默病(Alzheimer’s disease,AD)、朊病毒(prion)相关疾病和帕金森病(Parkinson’s disease,PD)等[134,135,136]。模拟分析结果显示,近1/3的胞质蛋白质存在共翻译折叠现象(co-translational protein folding)[137],暗示了翻译延伸速率可能对蛋白质折叠发挥重要的调控作用(图3J)。
核糖体在同一条mRNA上延伸速率的异质性可能直接影响新合成蛋白质的构象。迄今为止,大多数相关例证均与密码子使用偏好性有关[138]。例如,稀有密码子被发现在蛋白质结构域边界区成簇出现,由其导致的翻译暂停一般被认为可为其上游蛋白质结构域的正确折叠提供充足时间[119,139,140,141,142]。通过改变同义密码子使用的方式改变翻译延伸速率可生成构象和功能异常的蛋白质[143,144,145,146]。除同义密码子的使用之外,一些基因组证据显示mRNA二级结构也可通过调控翻译延伸速率影响蛋白质折叠[147]。例如,拟南芥的mRNA二级结构倾向于在蛋白质结构域连接区或无序区增强,这可能会通过减缓翻译延伸保证新生肽链有充足的时间折叠出正确的蛋白质结构域[63];美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)Koonin课题组通过分析两个真核生物和3个原核生物的蛋白质结构数据也得到了类似的结论[148]。
蛋白质的共翻译折叠在新生肽链进入核糖体肽链输出通道的一刻即可开始[10]。核糖体肽链输出通道可容纳30~40个氨基酸长度的新生肽链,其宽度(10~20?)一般认为足够容纳α-螺旋(α-helix)等小型蛋白质二级结构在其内部折叠[149,150,151,152,153]。肽链输出通道出口外的新生肽链则可以与核糖体表面相互作用,这也可能调控蛋白质的折叠[154,155,156]。位于肽链输出通道外部的新生肽链还会接触到大量细胞质中的核糖体关联蛋白因子(ribosome-associated protein factor)与分子伴侣(chaperone),这些蛋白因子与核糖体及新生肽链的动态结合也被认为对蛋白质的共翻译折叠起到重要的调控作用[157,158,159]。例如,Liu等[160]研究显示,细菌中唯一的核糖体相关分子伴侣——触发因子(trigger factor)可通过结合EF-G的新生肽链来抑制结构域之间的错误相互作用,并防止未折叠区域的多肽对已折叠结构域的构象破坏。
4 、结语与展望
翻译延伸并非mRNA上密码子解码过程的简单重复,mRNA的编码序列携带的信息也并非只有氨基酸序列。相反,mRNA序列里蕴含着丰富的遗传信息,可调控蛋白质的合成、折叠、亚细胞定位以及mRNA的稳定性。伴随着研究手段的不断突破与创新,翻译延伸的调控机理和生物学效应开始逐步被解析与认知。考虑到生物体在不同的外界环境下、不同发育时间节点、不同器官组织细胞类型之间乃至同一细胞内的不同mRNA中,翻译延伸过程均可能受到特异的调控,翻译延伸过程的动态性与复杂性可能远远超过人们目前的认知。
关于翻译延伸当下仍有许多问题有待进一步探索。例如,细胞内核糖体组分的异质性是否参与调控翻译延伸速率?细胞如何区分翻译中止与暂停产生的串联双核糖体并引导不同的下游响应?细胞是通过哪些反式作用因子调整翻译延伸响应外界环境变化的?翻译延伸调控机器是否参与应激颗粒(stress granule)的形成从而迅速响应外界环境变化?翻译延伸调控的紊乱究竟是如何参与神经退行性疾病的发生与发展的?大量已有的以核糖体为靶点的药物是否可以用于改善翻译延伸速率并促进病理蛋白质的正确折叠?这一系列问题的解答必将为蛋白折叠相关疾病的诊断与治疗提供新的见解。
另外,设计新的生物系统、赋予细胞崭新的生物学功能的合成生物学方兴未艾。还有一系列翻译调控相关问题有待回答。例如,如何优化mRNA序列以避免翻译延伸迟滞对核糖体的占用和对细胞整体翻译效率的影响?如何优化mRNA序列以促进特定蛋白质的正确折叠与亚细胞定位?因此,对于翻译延伸调控背后错综复杂的分子机理的解析还将帮助合成生物学建立未来人工设计的新规则。
参考文献
[1]Zheng CX,Ma XF,Zhang YH,Li HJ,Zhang GF.Research progress in the mechanism of translation initiation of eukaryotic m RNAs.Hereditas(Beijing),2018,40(8):607-619.郑超星,马小凤,张永华,李洪杰,张根发.真核生物m RNA翻译起始机制研究进展.遗传,2018,40(8):607-619.
[2]Stein KC,Frydman J.The stop-and-go traffic regulating protein biogenesis:how translation kinetics controls proteostasis.J Biol Chem,2019,294(6):2076-2084.
[3]Richter JD,Coller J.Pausing on polyribosomes:make way for elongation in translational control.Cell,2015,163(2):292-300.
[4]Choi J,Grosely R,Prabhakar A,Lapointe CP,Wang Jf,Puglisi JD.How messenger RNA and nascent chain sequences regulate translation elongation.Annu Rev Biochem,2018,87:421-449.
[5]Buskirk AR,Green R.Ribosome pausing,arrest and rescue in bacteria and eukaryotes.Philos Trans R Soc Lond B Biol Sci,2017,372(1716):20160183.
[6]Voorhees RM,Ramakrishnan V.Structural basis of the translational elongation cycle.Annu Rev Biochem,2013,82:203-236.
[7]Lin JZ,Gagnon MG,Bulkley D,Steitz TA.Conformational changes of elongation factor G on the ribosome during t RNA translocation.Cell,2015,160(0):219-227.
[8]Gagnon MG,Lin JZ,Bulkley D,Steitz TA.Crystal structure of elongation factor 4 bound to a clockwise ratcheted ribosome.Science,2014,345(6197):684-687.
[9]Mustoe AM,Brooks CL,Al-Hashimi HM.Hierarchy of RNA functional dynamics.Annu Rev Biochem,2014,83:441-466.
[10]Javed A,Christodoulou J,Cabrita LD,Orlova EV.The ribosome and its role in protein folding:looking through a magnifying glass.Acta Crystallogr D Struct Biol,2017,73(pt 6):509-521.
[11]Nygaard R,Romaniuk JAH,Rice DM,Cegelski L.Whole ribosome NMR:dipolar couplings and contributions to whole cells.J Phys Chem B,2017,121(40):9331-9335.
[12]Bai XC,Fernandez IS,Mc Mullan G,Scheres SH.Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles.e Life,2013,2:e00461.
[13]Amunts A,Brown A,Bai XC,Llacer JL,Hussain T,Emsley P,Long F,Murshudov G,Scheres SHW,Ramakrishnan V.Structure of the yeast mitochondrial large ribosomal subunit.Science,2014,343(6178):1485-1489.
[14]Liu Z,Gutierrez-Vargas C,Wei J,Grassucci RA,Sun M,Espina N,Madison-Antenucci S,Tong L,Frank J.Determination of the ribosome structure to a resolution of 2.5?by single-particle cryo-EM.Protein Sci,2017,26(1):82-92.
[15]Loveland AB,Demo G,Grigorieff N,Korostelev AA.Ensemble cryo-EM elucidates the mechanism of translation fidelity.Nature,2017,546(7656):113-117.
[16]Fischer N,Neumann P,Konevega AL,Bock LV,Ficner R,Rodnina MV,Stark H.Structure of the E.coli ribosome-EF-Tu complex at<3?resolution by Cs-corrected cryo-EM.Nature,2015,520(7548):567-570.
[17]Zhang YQ,Mandava CS,Cao W,Li XJ,Zhang DJ,Li NN,Zhang YX,Zhang XX,Qin Y,Mi KX,Lei JL,Sanya S,Gao N.Hfl X is a ribosome-splitting factor rescuing stalled ribosomes under stress conditions.Nat Struct Mol Biol,2015,22(11):906-913.
[18]Johansson M,Bouakaz E,Lovmar M,Ehrenberg M.The kinetics of ribosomal peptidyl transfer revisited.Mol Cell,2008,30(5):589-598.
[19]Wang JF,Kwiatkowski M,Forster AC.Kinetics of ribosome-catalyzed polymerization using artificial aminoacyl-t RNA substrates clarifies inefficiencies and improvements.ACS Chem Biol,2015,10(10):2187-2192.
[20]Wohlgemuth I,Brenner S,Beringer M,Rodnina MV.Modulation of the rate of peptidyl transfer on the ribosome by the nature of substrates.J Biol Chem,2008,283(47):32229-32235.
[21]Chu D,Kazana E,Bellanger N,Singh T,Tuite MF,von der Haar T.Translation elongation can control translation initiation on eukaryotic m RNAs.EMBO J,2014,33(1):21-34.
[22]Morisaki T,Stasevich TJ.Quantifying single m RNAtranslation kinetics in living cells.Cold Spring Harb Perspect Biol,2018,10(11):a032078.
[23]Yan XW,Hoek TA,Vale RD,Tanenbaum ME.Dynamics of translation of single m RNA molecules in vivo.Cell,2016,165(4):976-989.
[24]Wu B,Eliscovich C,Yoon YJ,Singer RH.Translation dynamics of single m RNAs in live cells and neurons.Science,2016,352(6292):1430-1435.
[25]Morisaki T,Lyon K,De Luca KF,De Luca JG,English BP,Zhang ZJ,Lavis LD,Grimm JB,Viswanathan S,Looger LL,Lionnet T,Stasevich TJ.Real-time quantification of single RNA translation dynamics in living cells.Science,2016,352(6292):1425-1429.
[26]Steitz JA.Polypeptide chain initiation:nucleotide sequences of the three ribosomal binding sites in bacteriophage R17 RNA.Nature,1969,224(5223):957-964.
[27]Ingolia NT,Ghaemmaghami S,Newman JR,Weissman JS.Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling.Science,2009,324(5924):218-223.
[28]Pelechano V,Wei W,Steinmetz LM.Widespread co-translational RNA decay reveals ribosome dynamics.Cell,2015,161(6):1400-1412.
[29]Michel AM,Baranov PV.Ribosome profiling:a Hi-Def monitor for protein synthesis at the genome-wide scale.Wiley Interdiscip Rev RNA,2013,4(5):473-490.
[30]Zhao J,Zhang G.Translatomics:methods and applications.Chem Life,2017,37(1):70-79.赵晶,张弓.翻译组学:方法及应用.生命的化学,2017,37(1):70-79.
[31]Ingolia NT,Lareau LF,Weissman JS.Ribosome profiling of mouse embryonic stem cells reveals the complexity and dynamics of mammalian proteomes.Cell,2011,147(4):789-802.
[32]Qian WF,Yang JR,Pearson NM,Maclean C,Zhang JZ.Balanced codon usage optimizes eukaryotic translational efficiency.PLo S Genet,2012,8(3):e1002603.
[33]Weinberg DE,Shah P,Eichhorn SW,Hussmann JA,Plotkin JB,Bartel DP.Improved ribosome-footprint and m RNA measurements provide insights into dynamics and regulation of yeast translation.Cell Rep,2016,14(7):1787-1799.
[34]Hussmann JA,Patchett S,Johnson A,Sawyer S,Press WH.Understanding biases in ribosome profiling experiments reveals signatures of translation dynamics in yeast.PLo S Genet,2015,11(12):e1005732.
[35]Subramaniam AR,Zid BM,O'Shea EK.An integrated approach reveals regulatory controls on bacterial translation elongation.Cell,2014,159(5):1200-1211.
[36]Guydosh NR,Green R.Dom34 rescues ribosomes in 3untranslated regions.Cell,2014,156(5):950-962.
[37]Yang JR,Chen XS,Zhang JZ.Codon-by-codon modulation of translational speed and accuracy via m RNA folding.PLo S Biol,2014,12(7):e1001910.
[38]Dever TE,Dinman JD,Green R.Translation elongation and recoding in eukaryotes.Cold Spring Harb Perspect Biol,2018,10(8):a032649.
[39]Chen PJ,Huang YS.CPEB2-e EF2 interaction impedes HIF-1αRNA translation.EMBO J,2012,31(4):959-971.
[40]Grelet S,Howe PH.hn RNP E1 at the crossroads of translational regulation of epithelial-mesenchymal transition.J Cancer Metastasis Treat,2019,5:16.
[41]Desnoyers G,Bouchard MP,Masse E.New insights into small RNA-dependent translational regulation in prokaryotes.Trends Genet,2013,29(2):92-98.
[42]Ambros V,Chen XM.The regulation of genes and genomes by small RNAs.Development,2007,134(9):1635-1641.
[43]Grantham R,Gautier C,Gouy M,Mercier R,PavéA.Codon catalog usage and the genome hypothesis.Nucleic Acids Res,1980,8(1):r49-r62.
[44]Ikemura T.Correlation between the abundance of Escherichia coli transfer RNAs and the occurrence of the respective codons in its protein genes:A proposal for a synonymous codon choice that is optimal for the E.coli translational system.J Mol Biol,1981,151(3):389-409.
[45]Hershberg R,Petrov DA.Selection on codon bias.Annu Rev Genet,2008,42:287-299.
[46]Plotkin JB,Kudla G.Synonymous but not the same:the causes and consequences of codon bias.Nat Rev Genet,2011,12(1):32-42.
[47]Gingold H,Pilpel Y.Determinants of translation efficiency and accuracy.Mol Syst Biol,2011,7:481.
[48]Akashi H.Synonymous codon usage in Drosophila melanogaster:natural selection and translational accuracy.Genetics,1994,136(3):927-935.
[49]Stoletzki N,Eyre-Walker A.Synonymous codon usage in Escherichia coli:selection for translational accuracy.Mol Biol Evol,2007,24(2):374-381.
[50]Ikemura T.Correlation between the abundance of yeast transfer RNAs and the occurrence of the respective codons in protein genes.Differences in synonymous codon choice patterns of yeast and Escherichia coli with reference to the abundance of isoaccepting transfer RNAs.J Mol Biol,1982,158(4):573-597.
[51]Ikemura T.Codon usage and t RNA content in unicellular and multicellular organisms.Mol Biol Evol,1985,2(1):13-34.
[52]Novoa EM,Ribas de Pouplana L.Speeding with control:codon usage,t RNAs,and ribosomes.Trends Genet,2012,28(11):574-581.
[53]Buhr F,Jha S,Thommen M,Mittelstaet J,Kutz F,Schwalbe H,Rodnina MV,Komar AA.Synonymous codons direct cotranslational folding toward different protein conformations.Mol Cell,2016,61(3):341-351.
[54]Curran JF,Yarus M.Rates of aminoacyl-t RNA selection at 29 sense codons in vivo.J Mol Biol,1989,209(1):65-77.
[55]S?rensen MA,Kurland CG,Pedersen S.Codon usage determines translation rate in Escherichia coli.J Mol Biol,1989,207(2):365-377.
[56]Gardin J,Yeasmin R,Yurovsky A,Cai Y,Skiena S,Futcher B.Measurement of average decoding rates of the 61 sense codons in vivo.e Life,2014,3:e03735.
[57]Kertesz M,Iovino N,Unnerstall U,Gaul U,Segal E.The role of site accessibility in micro RNA target recognition.Nat Genet,2007,39(10):1278-1284.
[58]Beaudoin JD,Novoa EM,Vejnar CE,Yartseva V,Takacs CM,Kellis M,Giraldez AJ.Analyses of m RNAstructure dynamics identify embryonic gene regulatory programs.Nat Struct Mol Biol,2018,25(8):677-686.
[59]Rouskin S,Zubradt M,Washietl S,Kellis M,Weissman JS.Genome-wide probing of RNA structure reveals active unfolding of m RNA structures in vivo.Nature,2014,505(7485):701-705.
[60]Chartrand P,Meng XH,Huttelmaier S,Donato D,Singer RH.Asymmetric sorting of ash1p in yeast results from inhibition of translation by localization elements in the m RNA.Mol Cell,2002,10(6):1319-1330.
[61]Qu XH,Wen JD,Lancaster L,Noller HF,Bustamante C,Tinoco I.The ribosome uses two active mechanisms to unwind messenger RNA during translation.Nature,2011,475(7354):118-121.
[62]Kertesz M,Wan Y,Mazor E,Rinn JL,Nutter RC,Chang HY,Segal E.Genome-wide measurement of RNAsecondary structure in yeast.Nature,2010,467(7311):103-107.
[63]Tang Y,Assmann SM,Bevilacqua PC.Protein structure is related to RNA structural reactivity in vivo.J Mol Biol,2016,428(5):758-766.
[64]Watts JM,Dang KK,Gorelick RJ,Leonard CW,Bess JW,Swanstrom R,Burch CL,Weeks KM.Architecture and secondary structure of an entire HIV-1 RNAgenome.Nature,2009,460(7256):711-716.
[65]Dinman JD.Mechanisms and implications of programmed translational frameshifting.Wiley Interdiscip Rev RNA,2012,3(5):661-673.
[66]Biswas P,Jiang X,Pacchia AL,Dougherty JP,Peltz SW.The human immunodeficiency virus type 1 ribosomal frameshifting site is an invariant sequence determinant and an important target for antiviral therapy.J Virol,2004,78(4):2082-2087.
[67]Wu F,Zhao S,Yu B,Chen YM,Wang W,Song ZG,Hu Y,Tao ZW,Tian JH,Pei YY,Yuan ML,Zhang YL,Dai FH,Liu Y,Wang QM,Zheng JJ,Xu L,Holmes EC,Zhang YZ.A new coronavirus associated with human respiratory disease in China.Nature,2020,579(7798):265-269.
[68]Caliskan N,Peske F,Rodnina MV.Changed in translation:m RNA recoding by-1 programmed ribosomal frameshifting.Trends Biochem Sci,2015,40(5):265-274.
[69]Mustoe AM,Busan S,Rice GM,Hajdin CE,Peterson BK,Ruda VM,Kubica N,Nutiu R,Baryza JL,Weeks KM.Pervasive regulatory functions of m RNA structure revealed by high-resolution SHAPE probing.Cell,2018,173(1):181-195.e118.
[70]Su Z,Tang Y,Ritchey LE,Tack DC,Zhu M,Bevilacqua PC,Assmann SM.Genome-wide RNA structurome reprogramming by acute heat shock globally regulates m RNA abundance.Proc Natl Acad Sci USA,2018,115(48):12170-12175.
[71]Pavlov MY,Watts RE,Tan Z,Cornish VW,Ehrenberg M,Forster AC.Slow peptide bond formation by proline and other N-alkylamino acids in translation.Proc Natl Acad Sci USA,2009,106(1):50-54.
[72]Muto H,Ito K.Peptidyl-prolyl-t RNA at the ribosomal P-site reacts poorly with puromycin.Biochem Biophys Res Commun,2008,366(4):1043-1047.
[73]Melnikov S,Mailliot J,Rigger L,Neuner S,Shin BS,Yusupova G,Dever TE,Micura R,Yusupov M.Molecular insights into protein synthesis with proline residues.EMBO Rep,2016,17(12):1776-1784.
[74]Doerfel LK,Wohlgemuth I,Kothe C,Peske F,Urlaub H,Rodnina MV.EF-P is essential for rapid synthesis of proteins containing consecutive proline residues.Science,2013,339(6115):85-88.
[75]Ude S,Lassak J,Starosta AL,Kraxenberger T,Wilson DN,Jung K.Translation elongation factor EF-P alleviates ribosome stalling at polyproline stretches.Science,2013,339(6115):82-85.
[76]Gutierrez E,Shin BS,Woolstenhulme CJ,Kim JR,Saini P,Buskirk AR,Dever TE.e IF5A promotes translation of polyproline motifs.Mol Cell,2013,51(1):35-45.
[77]Woolstenhulme CJ,Parajuli S,Healey DW,Valverde DP,Petersen EN,Starosta AL,Guydosh NR,Johnson WE,Wilson DN,Buskirk AR.Nascent peptides that block protein synthesis in bacteria.Proc Natl Acad Sci USA,2013,110(10):E878-E887.
[78]Starosta AL,Lassak J,Peil L,Atkinson GC,Virum?e K,Tenson T,Remme J,Jung K,Wilson DN.Translational stalling at polyproline stretches is modulated by the sequence context upstream of the stall site.Nucleic Acids Res,2014,42(16):10711-10719.
[79]Peil L,Starosta AL,Lassak J,Atkinson GC,Virum?e K,Spitzer M,Tenson T,Jung K,Remme J,Wilson DN.Distinct XPPX sequence motifs induce ribosome stalling,which is rescued by the translation elongation factor EF-P.Proc Natl Acad Sci USA,2013,110(38):15265-15270.
[80]Rodnina MV.The ribosome in action:tuning of translational efficiency and protein folding.Protein Sci,2016,25(8):1390-1406.
[81]Ito K,Chiba S.Arrest peptides:cis-acting modulators of translation.Annu Rev Biochem,2013,82:171-202.
[82]Tenson T,Ehrenberg M.Regulatory nascent peptides in the ribosomal tunnel.Cell,2002,108(5):591-594.
[83]Nakatogawa H,Ito K.The ribosomal exit tunnel functions as a discriminating gate.Cell,2002,108(5):629-636.
[84]Muto H,Nakatogawa H,Ito K.Genetically encoded but nonpolypeptide prolyl-t RNA functions in the A site for Sec M-mediated ribosomal stall.Mol Cell,2006,22(4):545-552.
[85]Tanner DR,Cariello DA,Woolstenhulme CJ,Broadbent MA,Buskirk AR.Genetic identification of nascent peptides that induce ribosome stalling.J Biol Chem,2009,284(50):34809-34818.
[86]Navon SP,Kornberg G,Chen J,Schwartzman T,Tsai A,Puglisi EV,Puglisi JD,Adir N.Amino acid sequence repertoire of the bacterial proteome and the occurrence of untranslatable sequences.Proc Natl Acad Sci USA,2016,113(26):7166-7170.
[87]Lu JL,Kobertz WR,Deutsch C.Mapping the electrostatic potential within the ribosomal exit tunnel.JMol Biol,2007,371(5):1378-1391.
[88]Lu JL,Deutsch C.Electrostatics in the ribosomal tunnel modulate chain elongation rates.J Mol Biol,2008,384(1):73-86.
[89]Koutmou KS,Schuller AP,Brunelle JL,Radhakrishnan A,Djuranovic S,Green R.Ribosomes slide on lysine-encoding homopolymeric A stretches.e Life,2015,4:e05534.
[90]Charneski CA,Hurst LD.Positively charged residues are the major determinants of ribosomal velocity.PLo SBiol,2013,11(3):e1001508.
[91]Artieri CG,Fraser HB.Accounting for biases in riboprofiling data indicates a major role for proline in stalling translation.Genome Res,2014,24(12):2011-2021.
[92]Sabi R,Tuller T.A comparative genomics study on the effect of individual amino acids on ribosome stalling.BMC Genomics,2015,16(Suppl.10):S5.
[93]Ikeuchi K,Izawa T,Inada T.Recent progress on the molecular mechanism of quality controls induced by ribosome stalling.Front Genet,2018,9:743.
[94]Sharma AK,O'Brien EP.Non-equilibrium coupling of protein structure and function to translation-elongation kinetics.Curr Opin Struct Biol,2018,49:94-103.
[95]Matsuo Y,Ikeuchi K,Saeki Y,Iwasaki S,Schmidt C,Udagawa T,Sato F,Tsuchiya H,Becker T,Tanaka K,Ingolia NT,Beckmann R,Inada T.Ubiquitination of stalled ribosome triggers ribosome-associated quality control.Nat Commun,2017,8(1):159.
[96]Ikeuchi K,Tesina P,Matsuo Y,Sugiyama T,Cheng J,Saeki Y,Tanaka K,Becker T,Beckmann R,Inada T.Collided ribosomes form a unique structural interface to induce Hel2-driven quality control pathways.EMBO J,2019,38(5):e100276.
[97]Brandman O,Stewart-Ornstein J,Wong D,Larson A,Williams CC,Li GW,Zhou S,King D,Shen PS,Weibezahn J,Dunn JG,Rouskin S,Inada T,Frost A,Weissman JS.A ribosome-bound quality control complex triggers degradation of nascent peptides and signals translation stress.Cell,2012,151(5):1042-1054.
[98]Shen PS,Park J,Qin YD,Li XM,Parsawar K,Larson MH,Cox J,Cheng YF,Lambowitz AM,Weissman JS,Brandman O,Frost A.Protein synthesis.Rqc2p and 60Sribosomal subunits mediate m RNA-independent elongation of nascent chains.Science,2015,347(6217):75-78.
[99]Verma R,Reichermeier KM,Burroughs AM,Oania RS,Reitsma JM,Aravind L,Deshaies RJ.Vms1 and ANKZF1 peptidyl-t RNA hydrolases release nascent chains from stalled ribosomes.Nature,2018,557(7705):446-451.
[100]Zurita Rendon O,Fredrickson EK,Howard CJ,Van Vranken J,Fogarty S,Tolley ND,Kalia R,Osuna BA,Shen PS,Hill CP,Frost A,Rutter J.Vms1p is a release factor for the ribosome-associated quality control complex.Nat Commun,2018,9(1):2197.
[101]Defenouillère Q,Yao YH,Mouaikel J,Namane A,Galopier A,Decourty L,Doyen A,Malabat C,Saveanu C,Jacquier A,Fromont-Racine M.Cdc48-associated complex bound to 60S particles is required for the clearance of aberrant translation products.Proc Natl Acad Sci USA,2013,110(13):5046-5051.
[102]Verma R,Oania RS,Kolawa NJ,Deshaies RJ.Cdc48/p97 promotes degradation of aberrant nascent polypeptides bound to the ribosome.e Life,2013,2:e00308.
[103]Doma MK,Parker R.Endonucleolytic cleavage of eukaryotic m RNAs with stalls in translation elongation.Nature,2006,440(7083):561-564.
[104]Shoemaker CJ,Eyler DE,Green R.Dom34:Hbs1promotes subunit dissociation and peptidyl-t RNAdrop-off to initiate no-go decay.Science,2010,330(6002):369-372.
[105]Shoemaker CJ,Green R.Kinetic analysis reveals the ordered coupling of translation termination and ribosome recycling in yeast.Proc Natl Acad Sci USA,2011,108(51):E1392-1398.
[106]Pisareva VP,Skabkin MA,Hellen CU,Pestova TV,Pisarev AV.Dissociation by Pelota,Hbs1 and ABCE1 of mammalian vacant 80S ribosomes and stalled elongation complexes.EMBO J,2011,30(9):1804-1817.
[107]Dimitrova LN,Kuroha K,Tatematsu T,Inada T.Nascent peptide-dependent translation arrest leads to Not4p-mediated protein degradation by the proteasome.J Biol Chem,2009,284(16):10343-10352.
[108]Juszkiewicz S,Hegde RS.Initiation of quality control during poly(A)translation requires site-specific ribosome ubiquitination.Mol Cell,2017,65(4):743-750.e744.
[109]Bengtson MH,Joazeiro CAP.Role of a ribosomeassociated E3 ubiquitin ligase in protein quality control.Nature,2010,467(7314):470-473.
[110]Letzring DP,Wolf AS,Brule CE,Grayhack EJ.Translation of CGA codon repeats in yeast involves quality control components and ribosomal protein L1.RNA,2013,19(9):1208-1217.
[111]Simms CL,Hudson BH,Mosior JW,Rangwala AS,Zaher HS.An active role for the ribosome in determining the fate of oxidized Mrna.Cell Rep,2014,9(4):1256-1264.
[112]Ishimura R,Nagy G,Dotu I,Zhou HH,Yang XL,Schimmel P,Senju S,Nishimura Y,Chuang JH,Ackerman SL.RNA function.Ribosome stalling induced by mutation of a CNS-specific t RNA causes neurodegeneration.Science,2014,345(6195):455-459.
[113]Saini P,Eyler DE,Green R,Dever TE.Hypusinecontaining protein e IF5A promotes translation elongation.Nature,2009,459(7243):118-121.
[114]Shah P,Ding Y,Niemczyk M,Kudla G,Plotkin JB.Rate-limiting steps in yeast protein translation.Cell,2013,153(7):1589-1601.
[115]Carlini DB,Stephan W.In vivo introduction of unpreferred synonymous codons into the Drosophila Adh gene results in reduced levels of ADH protein.Genetics,2003,163(1):239-243.
[116]Lampson BL,Pershing NL,Prinz JA,Lacsina JR,Marzluff WF,Nicchitta CV,Mac Alpine DM,Counter CM.Rare codons regulate KRas oncogenesis.Curr Biol,2013,23(1):70-75.
[117]Torrent M,Chalancon G,de Groot NS,Wuster A,Madan Babu M.Cells alter their t RNA abundance to selectively regulate protein synthesis during stress conditions.Sci Signal,2018,11(546):eaat6409.
[118]Zhao TL,Huan Q,Sun J,Liu CY,Hou XL,Yu X,Silverman IM,Zhang Y,Gregory BD,Liu CM,Qian WF,Cao XF.Impact of poly(A)-tail G-content on Arabidopsis PAB binding and their role in enhancing translational efficiency.Genome Biol,2019,20:189.
[119]Chen SY,Li K,Cao WQ,Wang J,Zhao T,Huan Q,Yang YF,Wu SH,Qian WF.Codon-resolution analysis reveals a direct and context-dependent impact of individual synonymous mutations on m RNA level.Mol Biol Evol,2017,34(11):2944-2958.
[120]Schikora-Tamarit MA,Carey LB.Poor codon optimality as a signal to degrade transcripts with frameshifts.Transcription,2018,9(5):327-333.
[121]Espinar L,Schikora Tamarit M?,Domingo J,Carey LB.Promoter architecture determines cotranslational regulation of m RNA.Genome Res,2018,28(4):509-518.
[122]Bazzini AA,Del Viso F,Moreno-Mateos MA,Johnstone TG,Vejnar CE,Qin YD,Yao J,Khokha MK,Giraldez AJ.Codon identity regulates m RNA stability and translation efficiency during the maternal-to-zygotic transition.EMBO J,2016,35(19):2087-2103.
[123]Presnyak V,Alhusaini N,Chen YH,Martin S,Morris N,Kline N,Olson S,Weinberg D,Baker KE,Graveley BR,Coller J.Codon optimality is a major determinant of m RNA stability.Cell,2015,160(6):1111-1124.
[124]Radhakrishnan A,Chen YH,Martin S,Alhusain N,Green R,Coller J.The DEAD-box protein Dhh1p couples m RNA decay and translation by monitoring codon optimality.Cell,2016,167(1):122-132.e129.
[125]Mishima Y,Tomari Y.Codon usage and 3'UTR length determine maternal m RNA stability in zebrafish.Mol Cell,2016,61(6):874-885.
[126]Siegel V,Walter P.Each of the activities of signal recognition particle(SRP)is contained within a distinct domain:analysis of biochemical mutants of SRP.Cell,1988,52(1):39-49.
[127]Halic M,Becke T,Pool MR,Spahn CM,Grassucci RA,Frank J,Beckmann R.Structure of the signal recognition particle interacting with the elongation-arrested ribosome.Nature,2004,42(6977):808-814.
[128]Crick SL,Ruff KM,Garai K,Frieden C,Pappu RV.Unmasking the roles of N-and C-terminal flanking sequences from exon 1 of huntingtin as modulators of polyglutamine aggregation.Proc Natl Acad Sci USA,2013,110(50):20075-20080.
[129]Brinkman RR,Mezei MM,Theilmann J,Almqvist E,Hayden MR.The likelihood of being affected with Huntington disease by a particular age,for a specific CAG size.Am J Hum Genet,1997,60(5):1202-1210.
[130]Li SH,Li XJ.Aggregation of N-terminal huntingtin is dependent on the length of its glutamine repeats.Hum Mol Genet,1998,7(5):777-782.
[131]Lee JM,Ramos EM,Lee JH,Gillis T,Mysore JS,Hayden MR,Warby SC,Morrison P,Nance M,Ross CA,Margolis RL,Squitieri F,Orobello S,Di Donato S,Gomez-Tortosa E,Ayuso C,Suchowersky O,Trent RJ,Mc Cusker E,Novelletto A,Frontali M,Jones R,Ashizawa T,Frank S,Saint-Hilaire MH,Hersch SM,Rosas HD,Lucente D,Harrison MB,Zanko A,Abramson RK,Marder K,Sequeiros J,Paulsen JS,Landwehrmeyer GB,Myers RH,Mac Donald ME,Gusella JF.CAG repeat expansion in Huntington disease determines age at onset in a fully dominant fashion.Neurology,2012,78(9):690-695.
[132]Rockabrand E,Slepko N,Pantalone A,Nukala VN,Kazantsev A,Marsh JL,Sullivan PG,Steffan JS,Sensi SL,Thompson LM.The first 17 amino acids of Huntingtin modulate its sub-cellular localization,aggregation and effects on calcium homeostasis.Hum Mol Genet,2007,16(1):61-77.
[133]Nissley DA,O'Brien EP.Altered co-translational processing plays a role in Huntington's pathogenesis-a hypothesis.Front Mol Neurosci,2016,9:54.
[134]Hartl FU.Protein misfolding diseases.Annu Rev Biochem,2017,86(1):21-26.
[135]Chiti F,Dobson CM.Protein misfolding,amyloid formation,and human disease:a summary of progress over the last decade.Annu Rev Biochem,2017,86:27-68.
[136]Soto C,Pritzkow S.Protein misfolding,aggregation,and conformational strains in neurodegenerative diseases.Nat Neurosci,2018,21(10):1332-1340.
[137]Ciryam P,Morimoto RI,Vendruscolo M,Dobson CM,O'Brien EP.In vivo translation rates can substantially delay the cotranslational folding of the Escherichia coli cytosolic proteome.Proc Natl Acad Sci USA,2013,110(2):E132-140.
[138]Komar AA.Synonymous codon usage-a guide for co-translational protein folding in the cell.Mol Biol(Mosk),2019,53(6):883-898.
[139]Clarke TF,Clark PL.Rare codons cluster.PLo S One,2008,3(10):e3412.
[140]Komar AA,Lesnik T,Reiss C.Synonymous codon substitutions affect ribosome traffic and protein folding during in vitro translation.FEBS Lett,1999,462(3):387-391.
[141]Zhang G,Ignatova Z.Folding at the birth of the nascent chain:coordinating translation with co-translational folding.Curr Opin Struct Biol,2011,21(1):25-31.
[142]Rodnina MV,Wintermeyer W.Protein elongation,co-translational folding and targeting.J Mol Biol,2016,428(10):2165-2185.
[143]Cortazzo P,Cerve?ansky C,Marín M,Reiss C,Ehrlich R,Deana A.Silent mutations affect in vivo protein folding in Escherchia coli.Biochem Biophys Res Commun,2002,293(1):537-541.
[144]Kimchi-Sarfaty C,Oh JM,Kim IW,Sauna ZE,Calcagno AM,Ambudkar SV,Gottesman MM.A“silent”polymorphism in the MDR1 gene changes substrate specificity.Science,2007,315(5811):525-528.
[145]Walsh IM,Bowman MA,Soto Santarriaga IF,Rodriguez A,Clark PL.Synonymous codon substitutions perturb cotranslational protein folding in vivo and impair cell fitness.Proc Natl Acad Sci USA,2020,117(7):3528-3534.
[146]Agashe D,Sane M,Phalnikar K,Diwan GD,Habibullah A,Martinez-Gomez NC,Sahasrabuddhe V,Polachek W,Wang J,Chubiz LM,Marx CJ.Large-effect beneficial synonymous mutations mediate rapid and parallel adaptation in a bacterium.Mol Biol Evol,2016,33(6):1542-1553.
[147]Yang JR.Does m RNA structure contain genetic information for regulating co-translational protein folding?Zool Res,2017,38(1):36-43.
[148]Faure G,Ogurtsov AY,Shabalina SA,Koonin EV.Role of m RNA structure in the control of protein folding.Nucleic Acids Res,2016,44(22):10898-10911.
[149]Voss NR,Gerstein M,Steitz TA,Moore PB.The geometry of the ribosomal polypeptide exit tunnel.JMol Biol,2006,360(4):893-906.
[150]Bhushan S,Gartmann M,Halic M,Armache JP,Jarasch A,Mielke T,Berninghausen O,Wilson DN,Beckmann R.Alpha-Helical nascent polypeptide chains visualized within distinct regions of the ribosomal exit tunnel.Nat Struct Mol Biol,2010,17(3):313-317.
[151]Nilsson OB,Hedman R,Marino J,Wickles S,Bischoff L,Johansson M,Müller-Lucks A,Trovato F,Puglisi JD,O'Brien EP,Beckmann R,von Heijne G.Cotranslational protein folding inside the ribosome exit tunnel.Cell Rep,2015,12(10):1533-1540
[152]Lu JL,Deutsch C.Folding zones inside the ribosomal exit tunnel.Nat Struct Mol Biol,2005,12(12):1123-1129.
[153]Woolhead CA,Mc Cormick PJ,Johnson AE.Nascent membrane and secretory proteins differ in FRET-detected folding far inside the ribosome and in their exposure to ribosomal proteins.Cell,2004,116(5):725-736.
[154]Kaiser CM,Goldman DH,Chodera JD,Tinoco I,Bustamante C.The ribosome modulates nascent protein folding.Science,2011,334(6063):1723-1727.
[155]Cabrita LD,Cassaignau AME,Launay HMM,Waudby CA,Wlodarski T,Camilloni C,Karyadi ME,Robertson AL,Wang XL,Wentink AS,Goodsell L,Woolhead CA,Vendruscolo M,Dobson CM,Christodoulou J.Astructural ensemble of a ribosome-nascent chain complex during cotranslational protein folding.Nat Struct Mol Biol,2016,23(4):278-285.
[156]Cabrita LD,Hsu ST,Launay H,Dobson CM,Christodoulou J.Probing ribosome-nascent chain complexes produced in vivo by NMR spectroscopy.Proc Natl Acad Sci USA,2009,106(2):22239-22244.
[157]Sandikci A,Gloge F,Martinez M,Mayer MP,Wade R,Bukau B,Kramer G.Dynamic enzyme docking to the ribosome coordinates N-terminal processing with polypeptide folding.Nat Struct Mol Biol,2013,20(7):843-850.
[158]D?ring K,Ahmed N,Riemer T,Suresh HG,Vainshtein Y,Habich M,Riemer J,Mayer MP,O'Brien EP,Kramer G,Bukau B.Profiling Ssb-nascent chain interactions reveals principles of Hsp70-assisted folding.Cell,2017,170(2):298-311.e220.
[159]Waudby CA,Dobson CM,Christodoulou J.Nature and regulation of protein folding on the ribosome.Trends Biochem Sci,2019,44(11):914-926.
[160]Liu KX,Maciuba K,Kaiser CM.The ribosome cooperates with a chaperone to guide multi-domain protein folding.Mol Cell,2019,74(2):310-319.e7.