摘 要: 除了作为遗传信息的载体,DNA所展现出的特殊材料性能引起了广泛关注。基于碱基互补配对原则的精确性和可编程性使得核酸纳米结构的构建逐步从一维单链发展到二维平面以及三维立体结构。计算机辅助工具的进步也促进了各种大小和形状的DNA纳米结构的自动化设计,而近年来构建的“框架核酸(FNAs)”为生物大分子纳米尺度上的精确排列提供了新方法,其固有的生物学功能以及可定制的特性使得其在物理、化学和生物等领域具有十分广阔的应用前景。本文阐述了精确自组装的FNAs的概念,并概述了FNAs在蛋白精确组装等领域的最新进展;重点论述了FNAs的优势所带来的对蛋白空间排布及其性能的调控能力,讨论了该领域存在的挑战,并对其发展机遇进行了展望。
关键词: 框架核酸; DNA纳米技术; DNA折纸; 蛋白精确组装;
Abstract: In addition to being the carrier of genetic information, DNA has attracted wide interest as a new class of material in the synthetic realm. The exploitation of the precise and programmable Watson-Crick base pairing of DNA or RNA has led to the development of exquisite nucleic acid nanostructures from one to three dimensions. Advances in computer-aided tools have also facilitated the automated design of DNA nanostructures with various sizes and shapes. The “framework nucleic acids(FNAs)” constructed in recent years provide a new method for precise organization of biological macromolecules with nanometer precision. The intrinsic biological properties and tailorable functionalities of FNAs hold great promise for physical, chemical, and biological applications. This review elaborates the concept of precise self-assembled FNAs, and summarizes the recent advances of FNAs in the field of protein precise arrangement. The unique features of FNAs that benefit the arrangement of proteins and their performance are highlighted. The challenges and opportunities of this field are also discussed.
Keyword: Framework Nucleic Acids; DNA nanotechnology; DNA origami; Protein precise arrangement;
DNA双螺旋结构是生物体的遗传学基础[1],除了这种最着名的结构外,核酸在自然界中也存在着许多其他的构型,例如单双链核糖开关、核酶[2]、环状RNA[3]以及结构更加复杂的G-四链体[4]和i-motif[5]等(图1(a))。这些天然的特殊结构已经被证明在活细胞中起着重要作用,如细胞转录控制、信号通路调控等。以自然为灵感,研究人员仿生构建了一系列更加复杂多样的DNA结构,包括DNA瓦片[6,7]、DNA四面体[8]、DNA折纸[9,10]以及DNA和无机化合物杂交形成的球形核酸[11,12]等(图1(b))。这些人造的DNA结构在调控活体和细胞内的生物进程方面具有极大的潜力。
20世纪80年代,Seeman通过定量混合四个具有特定序列的人工合成DNA单链打破了细胞内非固定的Holliday交叉的序列对称性限制,实现了固定的Holliday交叉的体外自组装[6,7]。Seeman认为,具有N(N≥3)条手臂链的DNA交叉都能够通过这种方法实现组装,并且通过设计用于杂交的粘性末端,可以使具有多条手臂链的DNA交叉进一步自组装成为复杂的纳米结构[6]。这一成果使DNA成为了不仅限于一维的纳米级聚合物,基于这一设想,科学家设计并建立了越来越复杂多样、契合应用的DNA纳米结构。

DNA纳米结构的可编程性和纳米精准性以及结构丰富性等性质使其能够作为可控刚性支架在纳米尺度精确排列多个分子,从而构建具有级联效应的分子网络。同时,DNA纳米结构由于其不同位点的序列特异性,可以作为纳米尺度的空间可寻址性支架,在这种情况下,能够实现生物分子间距离和相对比率的精确调控[13,14]。这其中,具有特殊物理化学特性的“框架核酸(Framework nuclear acids,FNAs)”纳米结构为体内和体外分子组装提供了新的思路[15,16,17,18,19]。FNAs特殊的理化性质主要体现在以下方面:第一,FNAs本身是高度单分散纳米结构,与精度较差的无机和有机纳米颗粒相比,其具有接近原子级别的精确度;第二,FNAs能够实现小分子、大分子或是纳米颗粒在纳米尺度上的精确位点调控[20,21,22,23,24];第三,FNAs和单双链DNA在细胞内化和分布方面有着显着的差异[25,26,27,28,29,30]。以FNAs的机械性能为例,FNAs的刚性是高度可控和可编程的。由于聚合物的延伸长度与半径的4次方成正比,由半径为10~100 nm的FNAs所构建的DNA纳米结构在延伸长度上超过单双链DNA 4~6个数量级。因此,柔性的DNA手臂链介导的无缝杂交而获得的刚性FNA支架为纳米尺度结构的构建提供了明确的思路。
图1 种类丰富的核酸结构
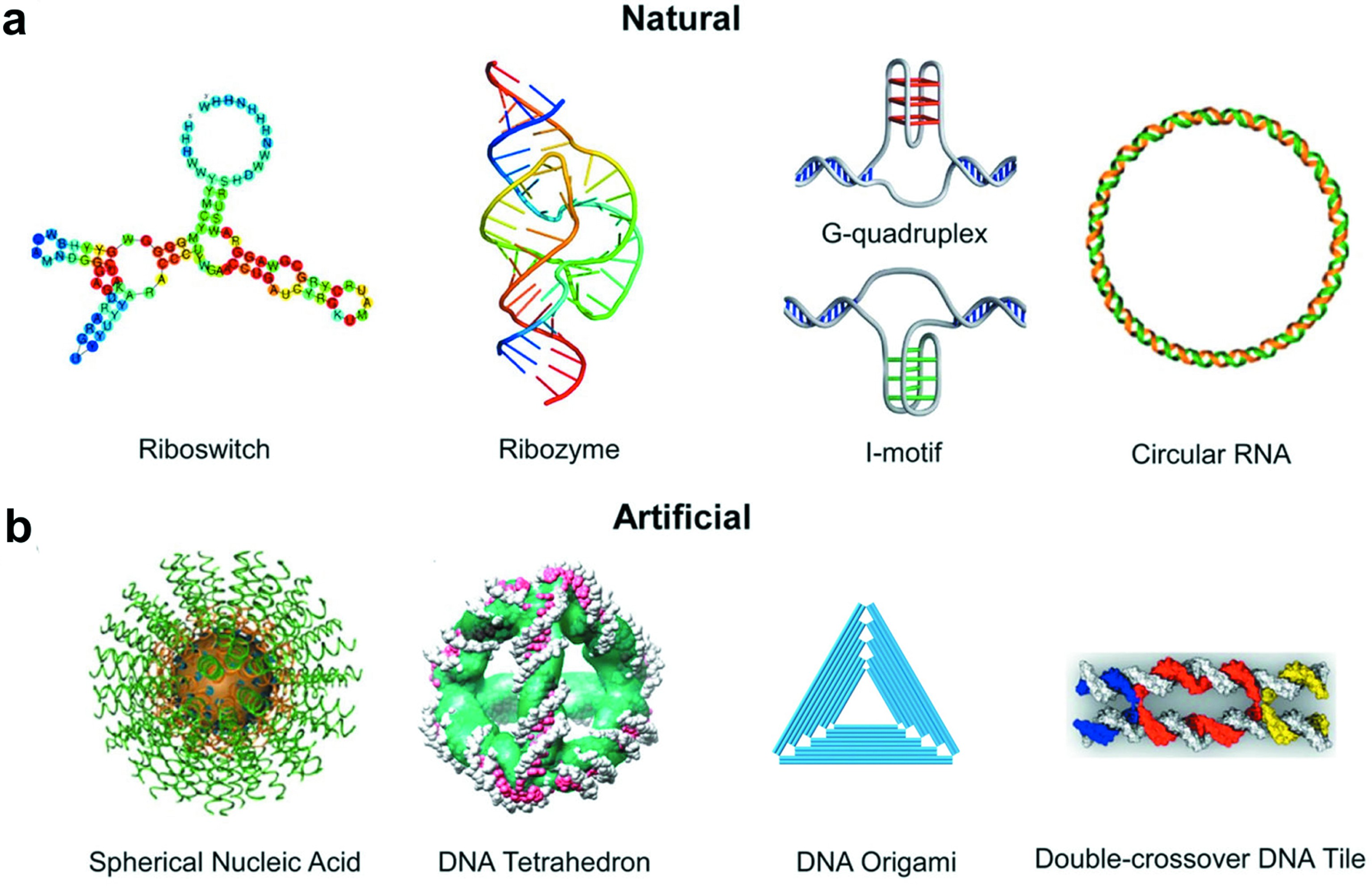
Fig.1 A wide variety of nucleic acid structures
(a)自然界中存在的核酸结构[2~5];(b)人工设计的核酸结构[6,8,9,11]
到目前为止,科学家已经研究了FNAs介导的距离依赖性分子相互作用、底物通道化和区室化效应等。此外FNAs已经被用于构建从体外小分子生物传感器到复杂的体内治疗性药物递送系统以及多酶网络等多种纳米器件[13]。本文分析了生物分子空间排列对其功能的影响,并进一步探讨了不同维度的FNAs在多肽和蛋白的精确组装方面的最新研究进展。
1、一维线性框架核酸
随着DNA化学合成的逐渐成熟,包括荧光染料、硫醇、生物素等在内的多种化学基团几乎可以与FNAs的组分链在任何位点实现共价连接[31],小分子官能团的共价修饰也成为了蛋白与核酸结合的基础,FNAs的精确自组装特性则可以实现对这些活性分子的精确空间调控。一维线性核酸作为最简单的框架结构与各类共价结合修饰技术往往被用于调控双蛋白之间的空间分布从而研究蛋白协同作用对其功能的影响。Freeman等[32]的研究表明,DNA双链能够调节多肽之间的间距,实现对多肽协同作用的调控。多肽RGDS和PHSRN是纤维连接蛋白的组分,它们协同调控细胞的黏附行为。以DNA双链作支架,他们优化了RGDS和PHSRN之间的距离,提高了多肽协同作用的效率,与对照组相比,细胞黏附效率提高了46%(图2(a))。
除了对不同蛋白协同作用的空间调控,一维FNAs也用于构建仿生的人工蛋白。以FNAs作为骨架,实现了蛋白功能亚基的自组装。Niemeyer等[33]利用FNAs距离调控实现了细胞色素P450 BM3亚基的组装,BM3含有两个亚基,即还原酶亚基BMR和卟啉亚基BMP,两个亚基分别与HaloTag蛋白融合,HaloTag蛋白分别与氯烷烃修饰的DNA偶联,当DNA通过碱基互补配对连接在一起时,在特定的间距下,两个蛋白亚基能够实现重组,并且保留了BM3的酶活性(图2(b))。
图2 一维线性框架核酸
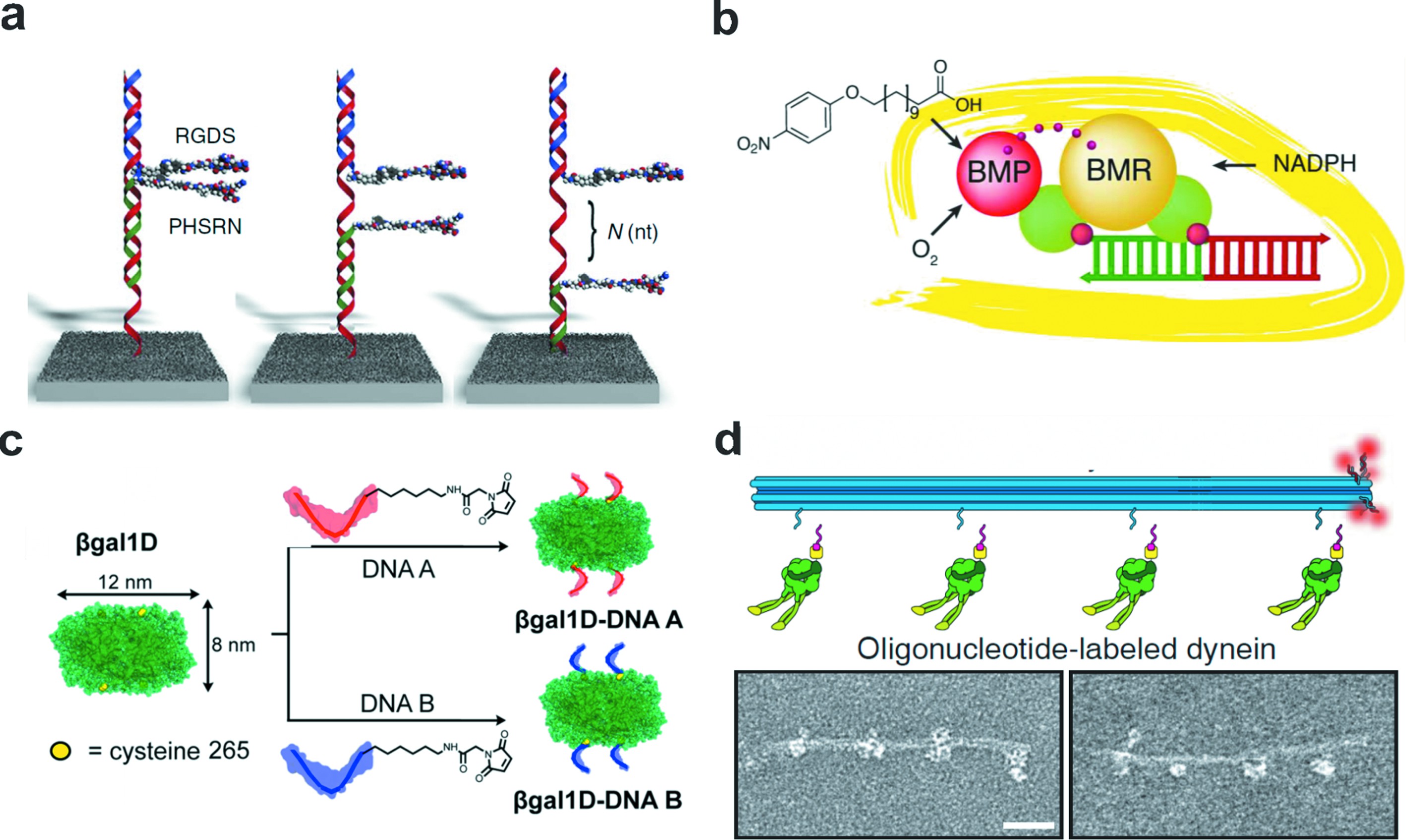
Fig.2 1D framework nucleic acids
(a)利用框架核酸调整多肽RGDS和PHSRN的间距而调控它们之间的协同作用[32];(b)在框架核酸支架上组装BMR还原酶结构域和BMP卟啉结构域,探究细胞色素P450 BM3的距离依赖性亚基自组装[33];(c)利用框架核酸实现β-半乳糖苷酶的有序自组装[34];(d)框架核酸上组装马达蛋白仿生模拟其生物学功能[35]
(a) FNAs were used to tune the distance between the peptides RGDS and PHSRN for study of their synergy[32];(b) Design of distance-dependence study of cytochrome P450 BM3 subunit self-assembly with organizing the BMR reductase domain and the BMP porphyrin domain on FNA[33];(c) Controllable self-assembly ofβ-galactosidase using FNA[34];(d) An ensemble of motor proteins on FNA for biomimicking of biological functions[35]
FNAs的高效自组装使得其在蛋白质工程领域也有着应用价值。依托一维FNAs的线形结构,McMillan等[34]使用对称四聚体蛋白质β-半乳糖苷酶作为模型蛋白,将265号位的苏氨酸突变成半胱氨酸,用于在蛋白顶部和底部分别交联马来酰亚胺修饰的DNA,利用DNA介导的方向控制促使β-半乳糖苷酶轴向排列成一维DNA-蛋白质纳米线,而不是非特异性聚集(图2(c))。这种FNAs介导的蛋白有序聚集也被应用于构建仿生的生物反应体系,进而研究生物体内的反应进程,例如马达蛋白的生物学功能。研究者在FNAs支架上组装多种马达蛋白分子来研究它们之间的相互作用,Derr等[35]利用DNA折纸研究了动力蛋白和驱动蛋白在细胞微管上运送货物的轨迹,在十二螺旋束DNA折纸上组装了极性相反的动力蛋白和驱动蛋白,仿生模拟了这两种蛋白在细胞微管上的“拔河”行为(图2(d))。还有研究者开发了基于马达蛋白和DNA复合物的人工自组装运输系统,在特定分子的诱导下,该系统能够实现组装和拆卸以及在分子轨道上装卸货物。
2、 二维平面框架核酸
天然存在的细胞通路以及级联反应大多需要多个蛋白参与,一维线性的FNAs本身修饰位点的匮乏限制了多蛋白协同以及蛋白级联反应的研究。基于DNA可编程性,科学家设计了更为丰富、维度更高的DNA纳米结构。二维FNAs由于其特殊的平面结构以及表面粘性末端的可设计性,实现了平面限域酶级联体系的精确组装,基于此对多蛋白协同作用进行研究,并通过分子开关实现了蛋白级联反应的人工干预。基于FNAs的精确自组装和可寻址性,Wilner等[36]率先在二维六角形支架上实现了葡萄糖氧化酶(GOX)/辣根过氧化物酶(HRP)蛋白纳米精度的自组装,以二维六角形DNA阵列作为支架,通过改变探针链的位置来实现蛋白空间分布的人工调控。由于六边形DNA框架的距离调控与表面限域作用,反应中间产物过氧化氢的扩散效率更高,因而实现更高的级联效率。因此,以两个六边形DNA框架作为支架时的酶活性比四个六边形DNA框架高出20%(图3(a))。有趣的是,这种距离决定的级联效率却不是完全随距离增加而逐渐降低的。基于DNA纳米材料的高纳米精度,科学家利用DNA平面结构进行了更精确的距离控制,系统地研究了酶间距对级联效率的影响。实验结果表明,在增加酶间距时,级联活性并未逐渐降低,而是在酶间距为10nm时出现了效率提高的现象,这是由于蛋白相互作用时形成的水和层促进反应中间体过氧化氢的扩散导致的。
Yan等[37]通过在GOX和HRP之间构建不同大小的蛋白质桥验证了这个机制,桥连蛋白将反应中间体的布朗运动转变成依赖蛋白间形成的水和层进行扩散(图3(b))。然而,除了GOX-HRP酶级联体系以外的其他酶级联反应,涉及到过氧化氢以外的其他反应中间体还需要进行进一步的研究,从而充分了解酶级联反应的距离依赖性。提高底物通道的中间体传递效率也有助于提高酶级联效率,从本质上来讲,底物通道用于将中间体从一种酶转移到另一种酶,以促进酶级联反应。Yan等[38]利用人工摆臂仿生构建了人工底物通道,建立了一种基于FNAs的多酶复合物,在两个双链刚性固定的脱氢酶葡萄糖-6-磷酸脱氢酶(G6pDH)和苹果酸脱氢酶(MDH)之间修饰了一条含20T的柔性人工摆臂在两种酶之间摆动,从而实现底物分子在酶级联体系中多个活性位点之间直接转移(图3(c))。利用FNAs支架对反应底物表面限域,实现了酶级联体系单个组分的精确位点控制。实验结果表明,与游离体系相比,基于FNAs的人工酶级联体系的反应活性提高了大约90倍,并且通过改变两种酶的相对比例能够进一步提高反应活性。除了调控酶级联反应活性外,FNAs的可编程性和生物相容性使得其在仿生领域也有着应用价值。Nielsen等[39]利用星形FNAs与抗体特定位点共价结合,在不影响抗体功能的情况下仿生构建抗体蛋白五聚体,模拟IgM的结构以及其生物学功能(图3(d))。
图3 二维平面FNAs
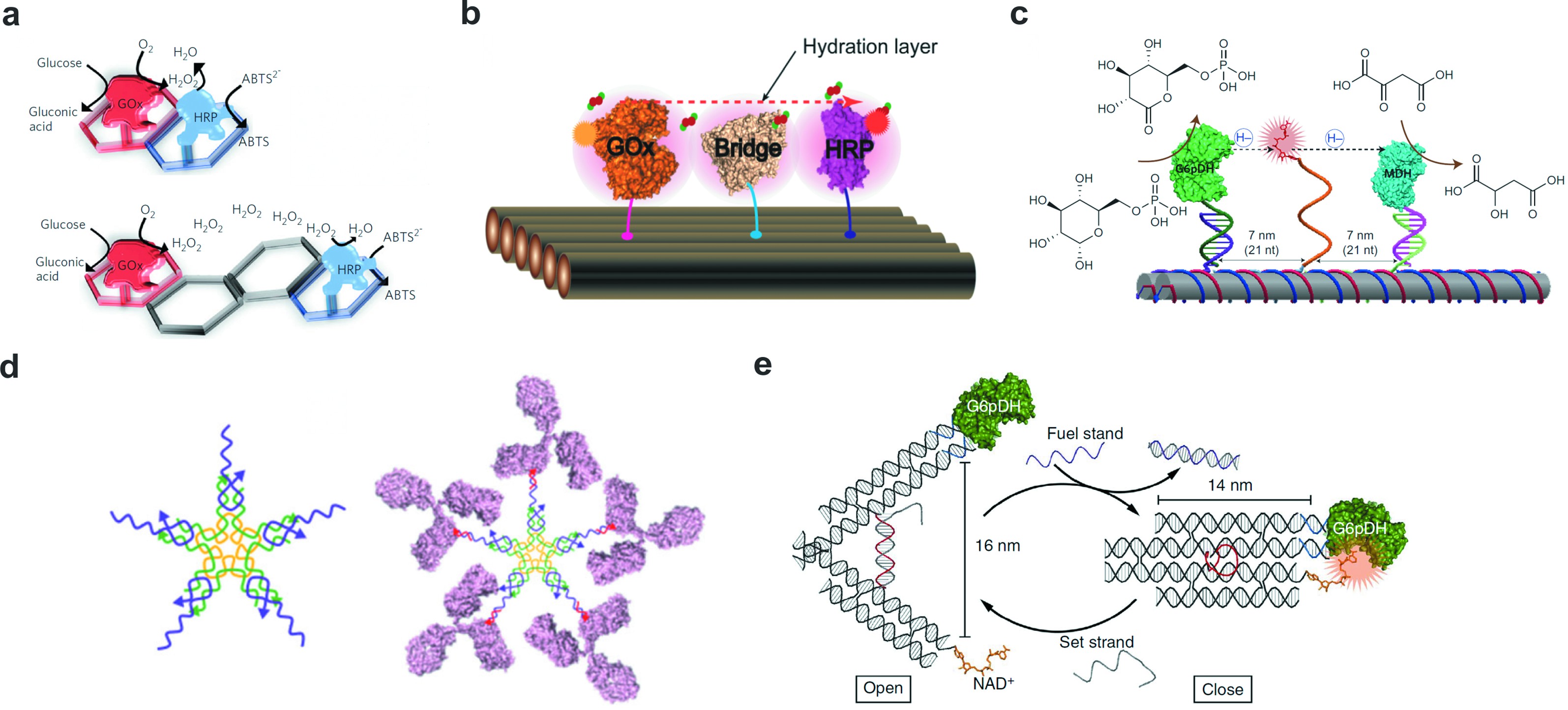
Fig.3 2D framework nucleic acids
(a)葡萄糖氧化酶(GOx)和辣根过氧化物酶(HRP)组装在六边形FNAs上,利用六边形的个数调节蛋白之间的间距[36];(b) FNAs表面构建GOx/HRP酶级联体系,蛋白质桥作为水合层促进过氧化氢扩散[37];(c) FNAs表面构建葡萄糖磷酸脱氢酶(G6p DH)和苹果酸脱氢酶(MDH)的多酶复合物,并利用腺嘌呤二核苷酸(NAD+)修饰的单链DNA作为摆动臂促进反应中间产物的转移[38];(d) FNAs用于仿生构建免疫球蛋白Ig M[39];(e) FNAs构建酶催化反应分子开关[41]
(a) Scheme of glucose oxidase (GOx) and horseradish peroxidase (HRP) cascade organized with two different distances between GOxand HRP on two-hexagon and four-hexagon strips[36];(b) Scheme of the assembled GOx/HRP pair with a protein bridge working as a connected hydration layer to facilitate H2O2diffusion[37];(c) An artificial swinging-arm multienzyme complex consisting of glucose-6-phosphate dehydrogenase (G6p DH) and malic dehydrogenase(MDH) organized on FNA,with a nicotinamide adenine dinucleotide (NAD+)-modified ss DNA as the swinging arm,facilitating the transfer of hydrides[38];(d) Bio mimic construction of immunoglobulin Ig M using FNA[39];(e) FNA constructs enzyme catalytic reaction molecular switch[41]
基于蛋白空间分布系统研究,科学家也建立了一系列的分子开关,对酶级联反应进行人工干预,而相较于一维线形结构,二维平面结构的限域作用实现了级联反应的可逆干预。作为一个经典的二维分子开关,DNA镊子被广泛用于组分之间的距离调控从而实现蛋白级联反应的激活。镊子通过DNA双链连接,在加入取代链之后形成稳定的茎环结构,基于此,研究者们探究了不同状态下GOX/HRP级联系统的活性。结果表明,DNA镊子的两个臂在打开和关闭的状态下酶级联活性相差约2倍[40]。而由于DNA分子开关的可逆性,DNA镊子可以在打开和关闭状态之间切换至少4个周期,因此DNA镊子也被应用于控制酶及其辅酶的结合和解离。Yan等[41]将G6pDH和烟酰胺腺嘌呤二核苷酸(NAD+)分别连接到DNA镊子的两个臂上,由于酶仅在直接“接触”辅酶时才具有活性,因此与开放状态相比,闭合状态下的酶活性提高了约5倍,并且这个开关状态是可以通过添加置换链实现可逆调节的(图3(e))。
3、 三维立体框架核酸
由于一维、二维FNAs蛋白修饰均暴露在结构表面,使得其难以实现蛋白簇空间限域研究。三维FNAs由于其立体结构能够精确地实现分子的空间排布,弥补了一维或二维FNAs低维度的不足。三维纳米结构介导的笼子状FNAs能够模拟天然区室构建纳米反应器,然后将代谢酶有序的封装进去,从而增强催化活性并提高蛋白酶消化条件下的稳定性(图4(a))[42]。
利用三维DNA晶体构建蛋白网络也是一件十分有趣的事情,自从第一个可自我组装的DNA晶体结构被设计出来,研究人员不断尝试对DNA晶体进行功能化(图4(b))[43]。使用DNA晶体实现蛋白的空间精确调控是非常有意义的,与单分子系统相比,研究三维网络如何影响蛋白功能有利于更加深入了解体内相关生物学功能。
三维立体FNAs的空间结构使得其能够将分子封装起来,避免分子受到周围环境的影响,因此基于三维FNAs的纳米载药系统在疾病诊疗领域也有着其应用价值。传统的药物载体或多或少地存在一些固有的局限性,例如,脂质体由于其尺寸不均一而可能诱导免疫毒性、无机纳米颗粒毒性元素或不可降解的残留物也存在安全隐患。而FNAs是由核酸组成的,因此具有天然的生物相容性和生物可降解性的优势,此外,FNAs能够以完全统一的尺寸进行组装并且具有良好的药物装载能力,它们的三维几何形状、表面化学和动态功能都是精准可控的。另外有“人工抗体”之称的DNA适配体的发展赋予了DNA识别功能,提高了药物递送的靶向性,这些优势使得FNAs成为药物递送的独特载体。Douglas等[44]2012年首先构建了能够靶向递送以及控制药物释放的DNA纳米结构,通过在DNA纳米盒表面修饰一个交互式“锁”,而解开这个“锁”的“钥匙”分子正是位于目标细胞的表面,实现了分子靶向以及药物释放的精确调控(图4(c))。Li等[45]在此基础上合作开发了一种纳米机器人用于治疗肿瘤(图4(d))。这样一个开创性的工作正是基于FNAs完成的,首先在90nm×60nm的折纸表面修饰4个凝血酶,然后将二维折纸卷成中空管(直径约19nm),这样凝血酶就附着在中空管内部,再用一系列的“紧固件”将其固定,同时,“紧固件”作为核仁素适配体还具有分子传感器的功能,它能特异性结合肿瘤血管标记物核仁素蛋白。静脉注射之后,在经过正常组织血管时,DNA纳米结构保护凝血酶使其不受血小板和血浆纤维原的影响,在到达肿瘤血管组织时,核仁蛋白与“紧固件”结合使其解离从而导致管状结构破裂,FNAs的构象改变使凝血酶暴露在血液中,诱导肿瘤部位的血液凝固,抑制肿瘤生长并导致肿瘤坏死。由于所有的实体瘤组织血管都是基本一致的,所以原则上这种方法可以有效地对抗多种类型的肿瘤,具有较高的普适性。尽管整个过程需要多个组件的参与,但其实整个体系是易于合成的,因为这些组件(紧固件,药物和成像标志物)可以很容易地集成到精心设计的FNAs上,并且均一度及产率很高。将多个功能基团组合在一起有效地提高了纳米机器人的人工智能,它可以读取环境信息并决定何时、何地以及是否采取行动。在肿瘤研究中,凝血酶由于其非特异性的凝结作用和短暂的血液循环半衰期而限制了在肿瘤治疗上的应用,但是通过设计特定结构和功能的FNAs能够完美的规避这些限制。将凝血酶这种不稳定分子精确组装在保护壳框架内,同时将多个分子传感器以纳米级分辨率集成到框架的适当位置,借助特异性的肿瘤靶向配体,基于三维FNAs的纳米机器人可以在肿瘤部位特异性聚集并以自主方式释放携带的凝血酶,从而最大程度地发挥药效,并最大程度地降低其毒性。这个体系也同样应用于由于存在稳定性问题或毒副作用而被排除在临床之外的其他备选药物。此外,该体系在治疗具有化学耐药性的肿瘤方面具有更高的价值,因为它的功能机制和癌细胞的分子背景无关,并且针对一种疗法产生的耐药性也不太可能与另一种疗法重叠。Xu等[46]也基于这个思路将蛋白和FNAs作为模块共同构建了四面体框架结构,以蛋白质作为顶点,能够在载药的同时实现位点特异性靶向识别(图4(e))。
图4 三维立体框架核酸
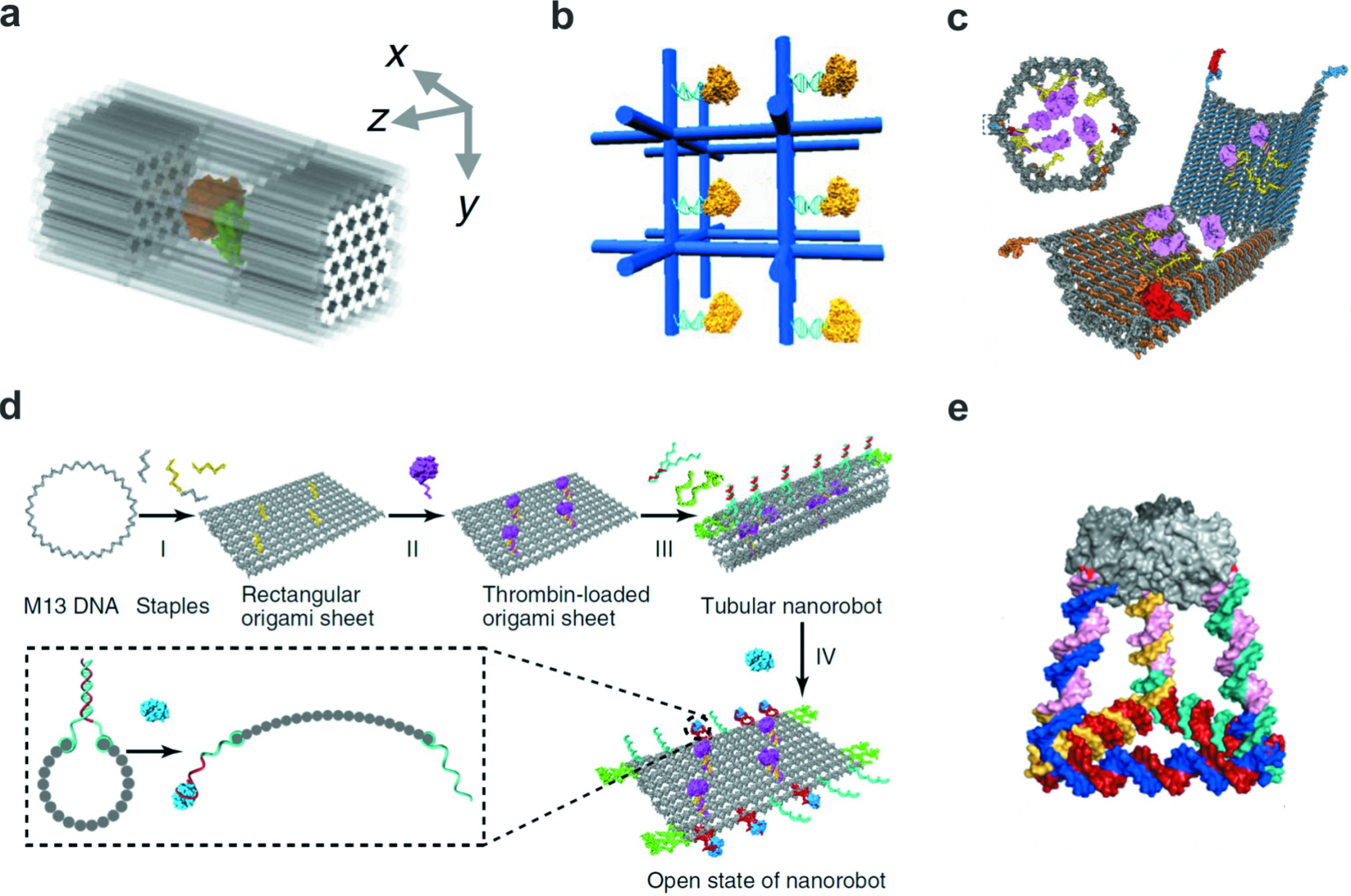
Fig.4 3D framework nucleic acids
(a)在DNA纳米笼子中精确组装一对蛋白酶[42];(b)三维DNA晶体中精确组装蛋白[43];(c)适配体作为分子开关的DNA纳米机器人用于载运蛋白[44];(d)框架核酸构建的纳米机器人用于肿瘤治疗[45];(e)蛋白和DNA共同构建的四面体纳米笼子用于药物靶向递送[46]
(a) A pair of protein enzymes precisely organized in a DNA cage[42];(c) Scheme of aptamer-gated DNA nanorobot loaded with a protein payload.Two DNA-aptamer locks are used to control opening and closing of the device[44];(d) A FNA-based nanorobot for cancer therapy[45];(e) Tetrahedral nanocages constructed by proteins and DNA are used for targeted drug delivery[46]
4 、总结与展望
本文概括了DNA框架核酸的概念,并简要总结了框架核酸在蛋白调控方面的最新进展。自然界中的许多信息处理和功能调节都是由核酸和蛋白质共同实现的,人工设计的DNA-蛋白质纳米结构的开发在许多领域都有着重要意义,包括合成生物学、纳米技术和生物医学等。近年来,DNA纳米技术的快速发展引起了蛋白质的精确调控的研究热潮,框架核酸的可编程性和生物相容性对于构建和模拟蛋白质体系方面有着广阔的前景。例如,利用框架核酸调控蛋白间距从而实现蛋白功能以及有序组装的调控,以框架核酸作为支架构建酶级联体系并探索其反应活性的影响因素,构建基于框架核酸的纳米机器人用于药物靶向和释放等。
设计和构建高度复杂的DNA纳米结构的高速发展为调控各种生物分子的组装提供了一个巨大的机会。然而,DNA与这些生物分子之间的整合仍有待进一步探索,寻找更为普适的在不影响蛋白功能域的情况下对DNA进行蛋白修饰的方法是科学家亟待解决的问题。尽管利用各种DNA支架构建蛋白质网络已经部分实现了天然体系的功能,但还有诸如方向性等多种相关参数有待进一步进行理论建模和系统实验。事实上,DNA纳米技术已经提供了一个高度可编程的平台,研究者们需要思考如何基于核酸框架进行更深入地探索与发现。
参考文献
[1] Serganov A,Nudler E.Cell,2013,152:17~24.
[2] Forster A C,Symons R H.Cell,1987,49:211~220.
[3] Sen D,Gilbert W.Nature,1988,334:364~366.
[4] 张素格,孙红霞,唐亚林.化学通报,2016,79(5):387~394.
[5] Zeraati M,Langley D B,Schofield P,et al.Nat.Chem.,2018,10:631~637.
[6] Seeman N C.J.Theor.Biol.,1982,99:237~247.
[7] Kallenbach N R,Ma R I,Seeman N C.Nature,1983,305:829~831.
[8] Kato T,Goodman R P,Erben C M,et al.Nano Ltt.,2009,9:2747~2750.
[9] Liu X,Zhang F,Jing X,et al.Nature,2018,559:593~598.
[10] Rothemund P W K.Nature,2006,440:297~302.
[11] Cutler J I,Auyeung E,Mirkin C A.J.Am.Chem.Soc.,2012,134:1376~1391.
[12] 张希伟,单志,王显祥.化学通报,2014,77(9):848~852.
[13] Yang Y R,Liu Y,Yan H.Bioconjug.Chem.,2015,26:1381~1395.
[14] Deng M,Li M,Li F,et al.ACS Mater.Lett.,2019,1:671~676.
[15] Ge Z,Gu H,Li Q,et al.J.Am.Chem.Soc.,2018,140:17808~17819.
[16] Lu N,Pei H,Ge Z,et al.J.Am.Chem.Soc.,2012,134:13148~13151.
[17] Ye D,Zuo X,Fan C.Ann.Rev.Anal.Chem.,2018,11:171~195.
[18] Yang F,Zuo X,Fan C,et al.Natl.Sci.Rev.,2018,5:740~755.
[19] 谢茉,谷沛霖,李宇,等.辐射研究与辐射工艺学报,2019,37:050101(10).
[20] Zhang H,Chao J,Pan D,et al.Nat.Commun.,2017,8:14738.
[21] Chao J,Wang J,Wang F,et al.Nat.Mater.,2019,18:273~279.
[22] Qi X,Zhang F,Su Z,et al.Nat.Commun.,2018,9:4579.
[23] Zhang F,Jiang S,Wu S,et al.Nat.Nanotechnol.,2015,10:779~784.
[24] Liu W,Halverson J,Tian Y,et al.Nat.Chem.,2016,8:867~873.
[25] Li J,Pei H,Zhu B,et al.ACS Nano,2011,5:8783~8789.
[26] Walsh A S,Yin H,Erben C M,et al.ACS Nano,2011,5:5427~5432.
[27] Jiang D,Ge Z,Im H J,et al.Nat.Biomed.Eng.,2018,2:865~877.
[28] He L,Lu D,Liang H,et al.J.Am.Chem.Soc.,2018,140:258~263.
[29] Zhu G,Zheng J,Song E,et al.PNAS,2013,110:7998~8003.
[30] Hao Y Y,Liu L,Zhang L H,et al.Nucl.Sci.Tech.,2018,29:83~89.
[31] Chen Q,Liu H,Lee W,et al.Lab Chip,2013,13:3351~3354.
[32] Freeman R,Stephanopoulos N,Alvarez Z,et al.Nat.Commun.,2017,8:15982.
[33] Erkelenz M,Kuo C H,Niemeyer C M.J.Am.Chem.Soc.,2011,133:16111~16118.
[34] McMillan J R,Mirkin C A.J.Am.Chem.Soc.,2018,140:6776~6779.
[35] Derr N D,Goodman B S,Jungmann R,et al.Science,2012,338:662~665.
[36] Wilner O I,Weizmann Y,Gill R,et al.Nat.Nanotechnol.,2009,4:249~254.
[37] Fu J,Liu M,Liu Y,et al.J.Am.Chem.Soc.,2012,134:5516~5519.
[38] Fu J,Yang Y R,Johnson-Buck A,et al.Nat.Nanotechnol.,2014,9:531~536.
[39] Nielsen T B,Thomsen R P,Mortensen M R,et al.Angew.Chem.Int.Ed.,2019,58:9068~9072.
[40] Xin L,Zhou C,Yang Z,et al.Small,2013,9:3088~3091.
[41] Liu M,Fu J,Hejesen C,et al.Nat.Commun.,2013,4:2127.
[42] Zhao Z,Fu J,Dhakal S,et al.Nat.Commun.,2016,7:10619.
[43] Rusling D A,Chandrasekaran A R,Ohayon Y P,et al.Angew.Chem.Int.Ed.,2014,53:3979~3982.
[44] Douglas S M,Bachelet I,Church G M.Science,2012,335:831~834.
[45] Li S,Jiang Q,Liu S,et al.Nat.Biotechnol.,2018,36:258~264.
[46] Xu Y,Jiang S,Simmons C R,et al.ACS Nano,2019,13:3545~3554.