含酚废水是水体重要污染物之一,其主要来源于焦化或以苯酚或酚醛为原料的化工生产过程。废水中的酚类物质可直接抑制土壤及水体中生物的正常生长,严重破坏环境生态系统。在含酚废水中,以苯酚毒性最大,通常含量也最高。现已经证明低浓度苯酚能使蛋白变性,而高浓度苯酚则直接导致蛋白质沉淀,引起生物组织深部损伤、坏死乃至整体中毒。
正是由于苯酚具有的生物危害性,美国环保署(EPA)把苯酚列入水体 129 种优先污染物中的第 65 位,并且规定废水中酚的浓度不得超过 1 mg/L;我国也把苯酚列入中国环境优先污染物黑名单之中,并且对含酚废水的排放有严格的规定:一般条件下,规定饮用水的含挥发性酚的浓度为 0.001 mg/L,水源水体中含酚最高容许浓度为 0.002 mg/L。
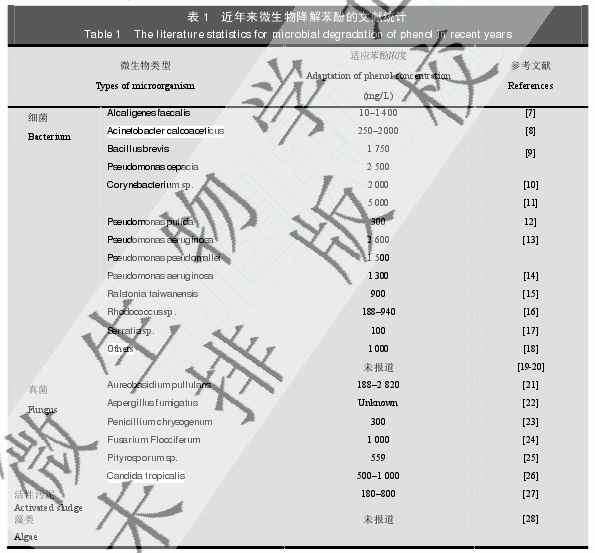
世界各国普遍重视含酚废水的处理,人们运用各种物理化学方法去除废水中的苯酚,例如溶剂萃取法、吸附法、化学氧化法及焚烧法等。但这些方法都具有一定的缺陷,例如成本高,耗能,排放更毒的副产物及不能完全去除苯酚等。在过去的几十年间,通过研究发现,许多微生物能以苯酚为唯一的碳源进行生长(表 1)。目前己基本了解微生物降解苯酚的生物学途径主要有两条:一条是好氧微生物利用苯酚羟化酶将苯酚氧化为邻苯二酚、乙酰辅酶A、琥珀酸及丙酮酸等,然后这些物质进入三羧酸循环继续被微生物利用;另一条则是通过厌氧微生物将苯酚羧化为 4-羟基苯甲酸,然后经过相关酶的作用,通过苯甲酸途径,生成乙酰辅酶 A,最终转化成乙酸,继续被微生物利用。与物理化学法相比,微生物降解苯酚的成本较低,环境友好,操作简单,但是高浓度的苯酚会抑制微生物的生长,从而影响苯酚的生物降解速率。因此筛选出能够耐高浓度苯酚的微生物是实现苯酚生物降解的关键。
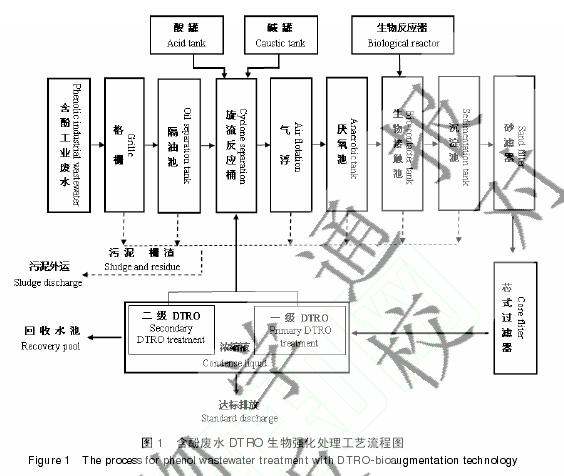
碟管式反渗透技术(DTRO)是一种新的水处理技术,通过碟管式的装置实现反渗透以达到高效处理污水的效果。该技术运用反渗透原理,即在浓溶液一边加上比自然渗透压更高的压力,扭转自然渗透方向,把浓溶液中的溶剂(水)压到半透膜另一边的稀溶液中。目前该技术多应用于垃圾填埋场的渗透液处理和海水的淡化上,本实验首次将这种技术用于含酚废水的处理。单独使用这种技术虽然能够有效地降低废水中苯酚的浓度,但在此过程中膜容易被苯酚腐蚀,从而导致膜使用寿命降低。
本实验利用煤化工环境废水及附近土样微生物细菌群,通过在以苯酚为唯一的碳源培养基上进行筛选、驯化和分离得到高效苯酚降解菌,并将此菌种制备成相应的水处理剂与DTRO 技术结合形成一种新的生物强化 DTRO 技术,为创制降解苯酚工业菌剂及其在煤化工领域的用途提供相关基础数据。
1 材料与方法
1.1 土壤浸渍液的准备
从云南开远解化化工公司生产部污水处理系统底部取出污泥样品,装入已灭菌的自封袋中,带回实验室置于 4 °C 冰箱中保存备用。然后各称取污泥 10 g,放入装有 90 mL 无菌水的锥形瓶中,振荡 4 h 后 2 000 r/min 低转速离心,取上层液体备用。
1.2 培养基
1.2.1 无机盐基础培养基(g/L):KH2PO41.0,K2HPO41.0,MgSO40.4,CaCl20.4,NaCl 0.2,(NH4)2SO40.1,FeSO4微量,琼脂 20,用 NaOH 调整其 pH 8.0;1×105Pa 灭菌 20 min。
1.2.2 纯培养斜面培养基:参照文献[30]进行配制。
1.2.3 NA 培养基:参照文献[31]进行配制。
1.2.4 液体种子培养基(g/L):蛋白胨 1.0,酵母膏 0.5,NaCl 1.0,用 NaOH 调整 pH 为 7.5 7.8;1×105Pa 灭菌 30 min。
1.2.5 苯酚降解培养基:专用菌剂培养基(g/L) (酵母膏 0.2,蛋白胨 0.8,(NH4)2SO40.2,MgSO40.6。酵母膏和蛋白胨成分在 40 h 后添加。摇瓶实验时还需补充 1.2.1 中其他无机成分,但在组合验证时则无须补充)。1×105Pa 灭菌 30 min 后添加不同浓度苯酚溶液(苯酚浓度分别为 1000、1 500、2 000 mg/L)。
1.3 苯酚降解菌的富集培养
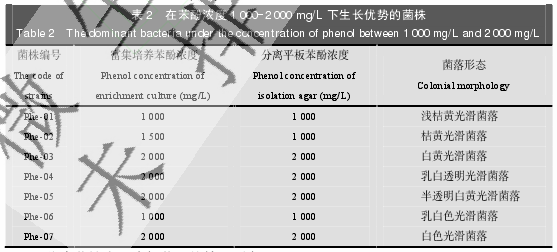
5 mL 的污泥浸渍液分别加入 100 mL 具有不同苯酚浓度的无机盐培养基中(苯酚浓度分别为 500、1 000、1 500、2 000、2 500 mg/L),于 28 °C、210 r/min 的摇床中培养 240 h。然后将各苯酚浓度富集培养液涂布于不同苯酚浓度无机盐基础培养基琼脂平板上,在 28 °C 的恒温箱中培养 48-96 h 后,将单菌落采用四分区划线法接种在纯化培养基的固体平板上。用保鲜膜封存好,置于 28 °C 的恒温箱中培养。最后挑取单一菌落于纯化斜面备用。
1.4 苯酚高效降解菌的分类鉴定
1.4.1 菌株 Phe-3 和 Phe-5 的生理和生物化学特征:参照文献[32]的方法来进行。
1.4.2 菌株的电镜显微照片:由扫描电子显微镜拍摄(Quanta 200,FEI 公司,美国)。
1.4.3 16S rRNA 基因的扩增与测序:菌株 Phe-03 和 Phe-05 在 NA 平板培养基上 28 °C 培养 3 d,然后挑取适量的菌体于微量离心管中,基因组 DNA 的提取按照参考文献[33]进行。
16S rRNA 基因的扩增与测序:16S rRNA 基因由两个引物(27F:5′-AGA GTT TGA TCC TGGGCG CTC AG-3′;1541R:5′-AAG GAG GTG ATC CAG CCG CAG-3′)来扩增。扩增体系含有 DNA 模板(大约 20 100 ng DNA) 1 μL;10×PCR 缓冲溶液(100 mmol/L Tris-HCl,pH 8.3;500 mmol/L KCl;15 mmol/L MgCl2) 5 μL,dNTPs 混合物(每个 dNTPs 浓度为 2.5 mmol/L,TaKaRa) 4 μL, 1.25 U Taq 酶(TaKaRa), 两个引物(终浓度为 0.4 μmol/L)各 1 μL,补足无菌去离子水 50 μL。PCR 扩增仪器为 GeneAmp PCR system (美国 PE 公司),扩增程序为:95 °C4 min;94 °C 1 min,60 °C 1 min,72 °C 1 min,35 个循环;72 °C 10 min。PCR 产物用 1%琼脂糖凝胶电泳进行检测,然后用手术刀切出 1 500 bp 大小的片段,用胶回收试剂盒(WATSON Gel Extraction Mini Kit)回收并纯化目的片段。然后用 AB I PR ISM 377–96sequencer 直接测定序列, BigDye ( Perkin2Elmer)作为终止剂,测序引物为 27F 和 1541R。
测序反应体系:纯化后的扩增产物约 1 μL (约 30 ng),BigDye 2.5 μL,引物 1 μL (约 3.2pmol/L),用水补足 10 μL。扩增程序为:95 °C 1 min,56 °C 1 min,72 °C 1 min,35 个循环;72 °C,5 min;4 °C 保存。
1.4.4 系统发育树的构建:测得的 DNA 序列经 NCBI (National Center for BiotechnologyInformation) BLAST 引擎搜索后获取相关种属的 16S rRNA 基因序列,用 ClustalX 1.8 软件进行排列。系统发育分析时排除碱基缺失位点,用邻接法(Neighbor-Joining analysis)构建系统发育树。距离矩阵按照 Kimura’s 双参数模型进行计算,Bootstrap 检验进行 1 000 次取样。
1.5 苯酚分析方法
采用高效液相分析方法测定苯酚浓度。HPLC 分析柱规格:YMC HPLC 苯酚专用分析柱(Hydrosphere C18250 mm×4.6 mm,5 μm,Japan);HPLC 分析系统为乙腈:水=80:20(体积比)等度;流速 1.0 mL/min;柱温 37 °C;在此条件下苯酚的出峰时间为 10.75 min。在 0-1 000mg/L 范围内,苯酚浓度与峰面积呈线性关系(R2=0.998 9)。苯酚浓度计算按回归方程进行计算:苯酚浓度(mg/L)=(测定峰面积 24 055)/4 382.7。
1.6 菌株降解苯酚分析
1.6.1 分离菌株降解苯酚性能初筛:配制无机盐培养基(每 250 mL 摇瓶中装 82 mL 培养液)。待灭菌冷却至适温后向摇瓶中均加入 18 mL10 g/L 的苯酚浓溶液,使得发酵瓶中含量达到 1800 mg/L。然后在无菌条件下,用消毒过的吸管从种子瓶中分别吸取菌液 5 mL 转接入摇瓶中(对照不进行接种),于 28°C、210 r/min 培养 120 h。各取样 1 mL 发酵液,12 000 r/min 离心 2 min 后,上清液进行高效液相色谱分析,每样重复 3 次测定,取平均值进行分析。
1.6.2 性能良好菌株降解苯酚性能考察:配制无机盐培养基(每 250 mL 摇瓶中装 87 mL 培养液)。待灭菌冷却至适温后向摇瓶中均加入 13 mL 10 g/L 的苯酚浓溶液,使得发酵瓶中含量达到 1 300 mg/L。然后在无菌条件下,用灭菌过的吸管从种子瓶中分别吸取菌液 5 mL 转接入摇瓶中(对照不进行接种),于 28 °C、210 r/min 培养 120 h。每 12 h 各取样 1 mL 发酵液,12 000 r/min 离心 2 min 后,上清液进行高效液相色谱分析,每样重复 3 次测定,取平均值进行分析。菌体量的测定通过分光光度计(OD600)进行测量。
1.7 苯酚降解菌DTRO组合工艺
将筛选出来的苯酚降解菌在专用生物培养器内扩繁(已申请中国发明专利保护。申请号:201210078150),添加专用营养剂(数据见 1.2.5)后制备成相应的水处理剂,然后与碟管式反渗透技术相结合用于对含苯酚废水的处理,即在 DTRO 工艺基础上,嫁接高效生物降解酚类物质的强化技术。观察反渗透膜的反应,并与单纯使用 DTRO 技术的结果进行对比。图 1是含酚废水 DTRO 生物强化技术处理的工艺流程图。
2 结果与分析
2.1 苯酚降解菌的获得
通过平板分离,我们观察到在 500-2 500 mg/L 苯酚浓度下,随着苯酚浓度的不断提高,平板上可培养微生物的种类越单一。通过观察,我们最终选择分离纯化在 1 000-2 000 mg/L苯酚浓度下生长呈优势的菌株(表 2)
2.2 分离菌株实际降解苯酚的效果测定
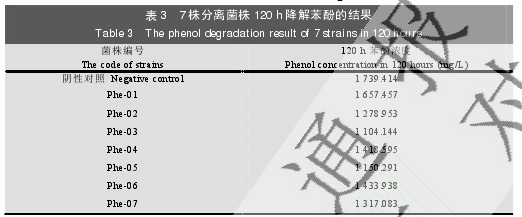
2.2.1 分离菌株苯酚降解性能初筛:从分离获得菌株 120 h 降解苯酚情况看,所有菌株均具备一定的降解苯酚能力(表 3)。其中,Phe-03 及 Phe-05 菌株降解能力较强。另外,考虑到本研究今后涉及的煤化工含酚污水中苯酚的浓度一般在 800-1 000 mg/L,且 1 800 mg/L 浓度的苯酚可能极大影响菌株的生长,影响苯酚的降解效率,故选择 Phe-03 及 Phe-05 菌株进行后续验证实验,并进一步将苯酚的起始浓度降低为 1 300 mg/L。
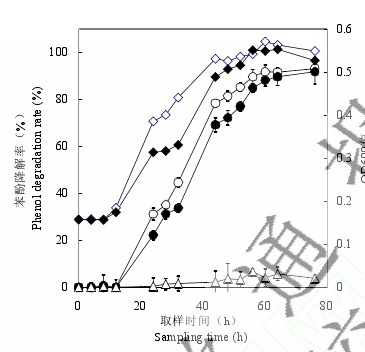
2.2.2 Phe-03 及 Phe-05 菌株苯酚降解性能比较:通过测定 Phe-03 及 Phe-05 在不同时段下的苯酚降解量情况(图 2);可以看出在培养 76 h 后,菌株 Phe-05 及 Phe-03 苯酚降解率均达到了 90%以上。但 Phe-05 菌株更适应培养环境,从图 2 中可以明显发现 Phe-05 菌株在经过12 h 的延迟生长后,OD600值呈快速增长趋势,表明在 12–44 h 期间菌株生长活力旺盛;与此同时,与 OD600生长曲线对应的苯酚降解曲线也逐级走高。这些现象表明:伴随着 Phe-05菌株的快速生长,苯酚作为唯一碳源物质也正在被快速消耗。44 h 以后,Phe-05 菌株的生长接近对数生长末期,也随着培养系统内苯酚残存浓度的降低,苯酚的降解率趋于稳定,苯酚降解曲线也趋于平缓。Phe-03 菌株的在 44 h 前的生长情况明显要比 Phe-05 菌株要逊色。
特别的是该菌株在 24–32 h 期间有一个明显的生长平台期,而且苯酚的降解在该阶段也有趋缓态势。我们初步判断可能与菌株培养系统内(NH4)2SO4浓度过低(与 Phe-05 菌株相比,Phe-03 培养系统内(NH4)2SO4浓度减少了 60%)有关。作为一种无机速效氮源,(NH4)2SO4的快速消耗必定会严重影响菌株一些重要生理物质比如与苯酚降解酶的生物合成,从而进一步影响了菌株的生长状况;而在 32 h 后 Phe-03 菌株生长又突破了前期平台继续呈增长趋势,针对该现象,我们认为可能是在此阶段添加在培养系统中的有机氮源物质发挥了作用,从而继续推动菌株发挥其生理功能。而未加菌的苯酚同步阴性对照在整个反应过程中浓度变化有一定起伏,但变化不大。说明仅仅依靠苯酚的自身挥发不能有效降低苯酚在水中的浓度。该结果也从一个方面证实微生物对苯酚降解的推动作用。
2.3 苯酚高效降解菌株 Phe-03 与 Phe-05 的分子生物学鉴定
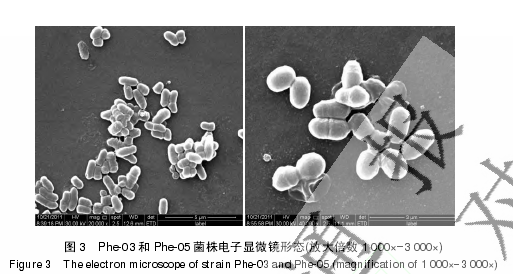
从平板培养上看 Phe-03 菌落质地柔软,带有白色微黄的颜色,而 Phe-05 菌落成片,质地柔软,呈白色。Phe-03 菌株呈短杆状[(0.1–0.2) μm×0.5 μm。见图 3];革兰氏染色呈阳性;检测生长温度 20–38 °C (适宜生长温度为 28–32 °C);检测生长 pH 4.5–9.0 (适宜 pH 6.0–8.0);纤维素、淀粉、酪蛋白水解阴性;可利用葡萄糖、蔗糖,不产 H2S;甲基红实验阳性;吲哚实验阴性;VP 实验阴性;柠檬酸盐利用阴性。Phe-05 菌株呈短球杆状[(0.2–0.3) μm×0.5 μm。见图 3];革兰氏染色呈阳性;检测生长温度 20–38°C (适宜生长温度为 28 °C);检测生长 pH4.5–9.0 (适宜 pH 6–8);纤维素、淀粉、酪蛋白水解阴性;可利用葡萄糖、蔗糖。H2S 实验弱阳性;甲基红实验阴性;吲哚实验阴性;VP 实验阴性;柠檬酸盐利用阴性。2 株菌均可以苯酚为唯一碳源生长。
根据 16S rRNA 基因有效序列分析构建的系统进化树。所有分支均采用最小二乘法(Least-squares)和极大似然(Maximum-likelihood)构树算法进行过计算和验证。分支节点上的数字表示经过 1 000 次自举(Bootstrap)分析所支持的结果。该结果表明所有菌株在进化树上的位置可靠(Bootstrap 值>70)。线段标尺(0.005)表示 0.5%序列差异的分支长度。由系统进化树可
以得知 Phe-03 与 Agromyces soli (GQ241325)的序列最大相似性达到 100%,故可推断 Phe-03 属于壤霉菌属(Agromyces)属的菌株;Phe-05 与最接近菌株 Corynebacterium lubricantis (FM173119)序列最大相似性小于 98%,推断 Phe-05 为棒杆菌属(Corynebacterium)的菌株。
2.4 苯酚降解菌-DTRO 工艺集成试验
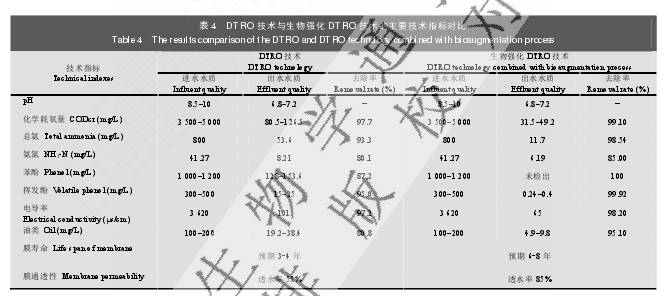
实验结果表明(表 4):与单纯使用 DTRO 技术相比,这种生物强化 DTRO 集成技术对含酚废水的处理效果更好,可有效控制含酚废水中酚类物质的浓度,减少膜污染,增大通透性,延长膜寿命。新工艺对高浓度含酚废水进行深度处理,处理效果好,出水水质达到《污水综合排放标准》GB8978-1996 一级标准,可用于含酚废水处理。
3 讨论
从煤化工污水处理系统污泥样中分离纯化了 2 株高效降解苯酚的菌株(分别编号为Phe-03 和 Phe-05)。它们均可以苯酚为唯一碳源生长。根据 16S rRNA 基因序列分析,初步判定 Phe-03 为壤霉菌属(Agromyces)的菌株;而 Phe-05 为棒状杆菌属(Corynebacterium)的菌株。到目前为止,壤霉菌属(Agromyces)微生物降解苯酚尚未见报道,因此 Phe-03 是苯酚降解菌的新成员。这为今后研究微生物降解苯酚提供了更多的选择,目前该两株菌已经申请了中国发明专利保护(Phe-05 菌株保藏编号为 CCTCC No:M2012031。专利申请号:
201310285990;Phe-03 菌株保藏编号为 CCTCC No:M2012030。专利申请号:201210057537)。根据相关文献可知棒状杆菌属(Corynebacterium)中的一些微生物可以利用苯酚作为唯一的碳源和能源,本研究也证实了这一点。在这其中最典型的例子就是谷氨酸棒状杆菌。许多文献揭示了谷氨酸棒状杆菌可以利用包括苯酚在内的各种芳香族化合物;目前对棒状杆菌属(Corynebacterium)微生物代谢苯酚的机制及其对苯酚污染环境修复研究已经有一系列深入报道。总之,该属微生物菌株在环境修复领域可发挥重要作用。
另外,DTRO 作为一种新型的水处理技术,目前多应用在垃圾填埋场的渗透液处理和海水淡化上。但是由于其反渗透膜成本高,投资大,易受污染,常需清理,日处理量小等缺陷致使该技术推广难度很大。因此 DTRO 技术目前还没有运用到对含酚废水的处理上。本研究首次将反渗透技术用于对含酚废水的处理,并且结合由苯酚高效降解菌 Phe-03 与 Phe-05制备成的相应的水处理剂,形成生物强化 DTRO 技术。这种技术不仅可以有效的除去废水中的苯酚含量,使得出水中未检测到苯酚,而且对污水中 CODcr、NH3-N 和油类等常规性指标去除率也都达到了较为理想状态。更重要的一点是这种技术的运用可以减少反渗透膜的污染,增加膜通透性提高了工业废水的处理水平,在一定程度上解决了将 DTRO 技术运用到含酚废水处理上的可能性。但要真正实现微生物技术与 DTRO 的有效对接还必须解决以下问题:(1) 微生物菌株在系统中的有效定植;(2) 微生物菌株在水处理系统温度变化情况下苯酚降解效率的稳定性;(3) 外加有机氮源是否造成二次污染;(4) 菌株的生物安全性评价。通过本研究工作,表明微生物技术可与 DTRO 技术联用,构建含酚废水生物强化处理工艺;本工作可为含酚废水处理研究提供一种选择思路。
参 考 文 献
[1] 琳稚兰, 黄秀梨. 现代微生物学与实验技术[M]. 北京: 科学出版社, 2000: 202-216.
[2] 张芳西. 含酚废水的处理与利用[M]. 北京: 化学工业出版社, 1983: 1-12.
[3] Nair CI, Jayachandran K, Shashidhar S. Biodegradation of phenol[J]. African Journal of Biotechnology, 2008, 7(25):4951-4958.
[4] Dutta NN, Borthakur S , Patil GS. Phase transfer catalyzed extraction of phenolic substances from aqueous alkalinestream[J]. Separation Science and Technology, 1992, 27(11): 1435-1448.
[5] 马文漪, 杨柳燕. 环境微生物工程[M]. 南京: 南京大学出版社, 1998: 131-132.
[6] 狄军贞. 苯酚降酚菌处理含酚废水的实验研究[D]. 阜新: 辽宁工程技术大学硕士学位论文, 2003.
[7] Bai J, Wen JP, Li HM, et al. Kinetic modeling of growth and biodegradation of phenol and m-cresol usingAlcaligenes faecalis[J]. Process Biochemistry, 2007, 42(4): 510-517.
[8] Adav SS, Lee DJ. Single-culture aerobic granules with Acinetobacter calcoaceticus[J]. Applied Microbiology andBiotechnology, 2008, 78(3): 551-557.13
[9] Arutchelvan V, Kanakasabai V, Nagarajan S, et al. Isolation and identification of novel high strength phenoldegrading bacterial strains from phenol-formaldehyde resin manufacturing industrial wastewater[J]. Journal ofHazardous Materials, 2005, 127(1/3): 238-243.
[10] Ho KL, Lin B, Chen YY, et al. Biodegradation of phenol using Corynebacterium sp. DJ1 aerobic granules[J].Bioresource Technology, 2009, 100(21): 5051-5055.
[11] Dalal S, Panigrahi DP, Randhawa GS, et al. CatA gene in a potential Corynebacterium strain is responsible for itsefficiency in phenol bioremoval[J]. Polycyclic Aromatic Compounds, 2012, 32(4): 423-438.
[12] El-Naas MH, Al-Muhtaseb SA, Makhlouf S. Biodegradation of phenol by Pseudomonas putida immobilized inpolyvinyl alcohol (PVA) gel[J]. Journal of Hazardous Materials, 2009, 164(2/3): 720-725.
[13] Afzal M, Iqbal S, Rauf S, et al. Characteristics of phenol biodegradation in saline solutions by monocultures ofPseudomonas aeruginosa and Pseudomonas pseudomallei[J]. Journal of Hazardous Materials, 2007, 149(1): 60-66.
[14] Kotresha D, Vidyasagar GM. Isolation and characterisation of phenol-degrading Pseudomonas aeruginosa MTCC4996[J]. World Journal of Microbiology and Biotechnology, 2008,24(4): 541-547.
[15] Chen BY, Chen WM, Chang JS. Optimal biostimulation strategy for phenol degradation with indigenous rhizobiumRalstonia taiwanensis[J]. Journal of Hazardous Materials, 2007,139(2): 232-237.
[16] 沈锡辉, 刘志培, 王保军, 等. 苯酚降解菌红球菌 PNAN5 菌株(Rhodococcus sp. strain PNAN5)的分离鉴定、降解特性及其开环双加氧酶性质研究[J]. 环境科学学报, 2004, 24(3): 482-486.
[17] Kobayashi F, Daidai M, Suzuki N, et al. Degradation of phenol in seawater using a novel microorganism isolatedfrom the intestine of Aplysia kurodai[J]. International Biodeterioration & Biodegradation, 2007, 59(3): 252-254.
[18] Geng A, Soh AEW, Lim CJ, et al. Isolation and characterization of a phenol-degrading bacterium from an industrialactivated sludge[J]. Applied Microbiology and Biotechnology, 2006, 71(5): 728-735.
[19] Jiang HL, Tay JH, Maszenan AM, et al. Enhanced phenol biodegradation and aerobic granulation by twocoaggregating bacterial strains[J]. Environmental Science & Technology, 2006, 40(19): 6137-6142.
[20] Jiang HL, Tay STL, Maszenan AM, et al. Physiological traits of bacterial strains isolated from phenol-degradingaerobic granules[J]. FEMS Microbiology Ecology, 2006, 57(2): 182-191.
[21] Dos Santos VL, Monteiro AS, Braga DT, et al. Phenol degradation by Aureobasidium pullulans FE13 isolated fromindustrial effluents[J]. Journal of Hazardous Materials, 2009, 161(2/3): 1413-1420.
[22] Jones KH, Trudgill PW, Hopper DJ. Evidence of two pathways for the metabolism of phenol by Aspergillusfumigates[J]. Archives of Microbiology, 1995,163(3): 176-181.
[23] Leitao AL, Duarte MP, Oliveira JS. Degradation of phenol by a halotolerant strain of Penicillium chrysogenum[J].
International Biodeterioration & Biodegradation, 2007, 59(3): 220-225.
[24] Anselmo AM, Novais JM. Degradation of phenol by immobilized mycelium of Fusarium flocciferum in continuousculture[J]. Water Science and Technology, 1992, 25(1): 161-168.
[25] 刘慧, 杨柳燕, 肖琳, 等. 酵母菌降解苯酚同时生产单细胞蛋白[J]. 农业环境科学学报, 2004, 23(4): 810-813.
[26] Rocha LL, Cordeiro RDA, Cavalcante RM , et al. Isolation and characterization of phenol-degrading yeasts from anoil refinery wastewater in Brazil[J]. Mycopathologia, 2007,164(4): 183-188.
[27] Vazquez-Rodriguez G, Youssef CB, Waissman-Vilanova J. Two-step modeling of the biodegradation of phenol byan acclimated activated sludge[J]. Chemical Engineering Journal, 2006, 117(3): 245-252.
[28] Semple KT, Cain RB. Biodegradation of phenols by the Alga Ochromonas danica[J]. Applied Microbiology andBiotechnology, 1996, 62(4): 1265-1273.
[29] 齐奇. 特种膜分离装备—碟管式反渗透[J]. 装备机械, 2011(3): 14-18.
[30] 沈萍, 陈向东. 微生物学实验[M]. 北京: 高等教育出版社, 2007: 246-247.
[31] 杨欢欢, 胡中泽, 熊魏, 等. 常用防腐剂在鱼糕保鲜中的应用研究[J]. 现代食品科技, 2011, 27(1): 90-95.
[32] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 349-417.
[33] Babu TG, Nithyanand C, Babu NKC, et al. Evaluation of cetyltrimethylammonium bromide as a potential short-termpreservative agent for stripped goat skin[J]. World Journal of Microbiology & Biotechnology, 2009, 25(5): 901-907.
[34] Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies ofnucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120.
[35] Fitch WM, Margoliash E. Construction of phylogenetic trees[J]. Science, 1967, 155(3760): 279-284.
[36] Felsenstein J. Evolutionary trees from DNA sequences: a maximum likelihood approach[J]. Journal of MolecularEvolution, 1981, 17(6): 368-376.
[37] Lee SY, Kim YH, Min J. Conversion of phenol to glutamate and proline in Corynebacterium glutamicum isregulated by transcriptional regulator ArgR[J]. Applied Microbiology and Biotechnology, 2010, 85(3): 713-720.
[38] Lee SY, Kim BN, Choi YW, et al. Growth response of avena sativa in amino-acids-rich soils converted fromphenol-contaminated soils by Corynebacterium glutamicum[J]. Journal of Microbiology and Biotechnology, 2012,22(4): 541-546.
[39] Lee SY, Kim BN, Han JH, et al. Treatment of phenol-contaminated soil by Corynebacterium glutamicum andtoxicity removal evaluation[J]. Journal of Hazardous Materials, 2010, 182(1/3): 937-940.
[40] Nardi A, Avrahami R, Zussman E, et al. Phenol biodegradation by Corynebacterium glutamicum encapsulated inelectrospun fibers[J]. Journal of Environmental Protection, 2012, 3(2): 164-168.