摘 要: 优质的微生物菌种对提高发酵工业产品的品质与产量有重要意义,而高效的育种技术对微生物选育工作至关重要。基因编辑技术作为现代遗传学研究的热点技术,极大地提升了现代工业微生物育种效率。基因编辑技术主要包含锌指核酸酶技术(ZFN)、转录激活因子样效应物核酸酶技术(TALEN)和成簇的规律间隔的短回文重复序列相关蛋白技术(CRISPR)3类。ZFN由于技术的复杂性,在工业微生物育种中鲜有报道。TALEN和CRISPR技术在酿酒酵母、米曲霉菌、黑曲霉、谷氨酸棒状杆菌和产甘油假丝酵母等主要工业微生物育种中相继展开应用,尤以CRISPR技术应用最为广泛。
关键词: 工业微生物育种; 基因编辑; TALEN; CRISPR;
Abstract: High-quality microbial strains are important to improve the quality and yield of industrial fermentation products, and high efficient breeding technology is also essential to microbial breeding. As a hot technology in modern genetics research, gene editing has greatly improved the efficiency of modern industrial microbial breeding. Three kinds of technology are contained in gene editing, which are ZFN, TALEN and CRISPR system respectively. ZFN is rarely used for industrial microbial breeding for its complexity of technology. TALEN and CRISPR technology have been gradually applied in breeding of major industrial microbes, such as S.cerevisiae, A.oryzae, A.niger, C.glutamicum, C.glycerinogenes, etc, especially CRISPR.
Keyword: industrial microbe breeding; gene editing; TALEN; CRISPR;
工业微生物育种学是一门通过遗传改良技术为工业生产提供有益微生物的学科。工业微生物育种对挖掘微生物应用潜力,提高发酵工业产品的品质与产量具有重要意义。工业微生物育种技术经历了常规突变体筛选、基因诱变、基因重组和基因工程4个重要阶段[1]。常规突变体筛选又称自然随机筛选,在非人工处理条件下,菌株自发突变,经筛选获得目标菌株;基因诱变是通过人为诱导使菌株发生突变,主要方法有物理诱变和化学诱变。物理诱变包括60Coγ射线、α射线、紫外线和快中子等方式,化学诱变主要利用NTG,EMS和DES等烷化剂,5-氟尿嘧啶、2-氨基嘌呤和5-溴去氧尿核等碱基类似物,吖啶橙和吖啶黄移码突变剂。基因重组是通过将不同特征的菌株进行遗传重组获取聚合优良性状的新菌株,主要技术方法有原生质体融合以及衍生出的原生质体诱变法、紫外处理原生质体法和原生质体电子融合技术等。这些技术均为非靶向诱变,突变的方向和性状是随机的,获得优良生产性能的菌株后,还要经过背景纯化,恢复其他正常功能才能进行工业化生产。因此,通过以上技术获取理想菌株存在的缺点是难度较大、效率较低。随着分子生物学研究技术的发展,微生物育种技术也从非靶向诱变向靶向型遗传改良转变。在不改变菌株背景性状的前提下,实现了育种目标的定向改良,是育种技术的里程碑式发展,极大地提高了工业微生物育种效率。

基因编辑技术作为现代生物学研究的热点技术,也是定向遗传改良技术的代表,在遗传改良育种、基因组学研究和遗传病治疗等方面均展现出巨大的应用潜力。基因编辑技术主要包含三代:第一代为锌指核酸酶技术(Zinc finger nuclease,ZFN),第二代为转录激活因子样效应物核酸酶技术(Transcription activator-like effector nuclease,TALEN),第三代为成簇的规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)相关蛋白(CRISPR associated proteins,Cas)技术。TALEN技术被《科学》杂志评为2012年度10大科学突破之一,CRISPR技术更是在2013年度、2015年度两次荣登该榜单。三代技术虽然原理不同,但都是通过识别模块定位特异性基因组序列,再利用核酸内切酶进行靶标序列切割形成双链断裂,最后通过引发内源性基因组修复机制实现基因组编辑目的[2]。ZFN由于技术的复杂性,在工业微生物育种应用中鲜有报道,TALEN技术和CRISPR技术则在酿酒酵母、黑曲霉和米曲霉等重要工业微生物遗传改良中相继展开应用。笔者旨在综述TALEN和CRISPR基因编辑技术的原理、发展及其在工业微生物育种中的应用。
1、转录激活因子样效应物核酸酶技术
1.1、技术原理
2007年,Sugio等[3]在植物病原菌黄单胞菌(Xanthomonas oryzae pv. oryzae)菌株PXO99A中,Ⅲ型效应基因pthXo6和pthXo7可编码转录激活因子样效应物(Transcription activator-like effector,TALE),该蛋白可诱导宿主水稻基因Os8N3的表达,增加水稻白叶枯病易感性。黄单胞菌属植物病原菌会在多种作物上引起严重病害,其毒性因子主要通过Ⅲ型分泌系统进入宿主细胞内。在结合到宿主DNA后诱导宿主特定基因表达,以破坏宿主防御系统或增加宿主对病原菌的敏感性,最终导致宿主生长发育异常或感染病害。
TALE家族蛋白是黄单胞菌的关键毒性因子,通过模拟真核转录因子对宿主基因组表达进行调控。科学家们就TALE蛋白结构及其结合DNA的分子机制展开研究[4,5]。TALEs家族蛋白序列较为保守,主要包含4个功能结构域:串联重复序列中心结构域、C端核定位信号(Nuclear localization signal,NLS)、N端分泌信号肽和转录激活结构域(Activating domain,AD)[6]。串联重复序列中心结构域一般由17个重复序列(Repeats)串联形成,每个重复序列由33~35个氨基酸组成。其中,位于12和13位置上的可变残基(Repeat variable diresidues/RVDs)决定了靶向序列的特异性,其余位置均为保守氨基酸(图1)[7]。
图1 TALE蛋白结构
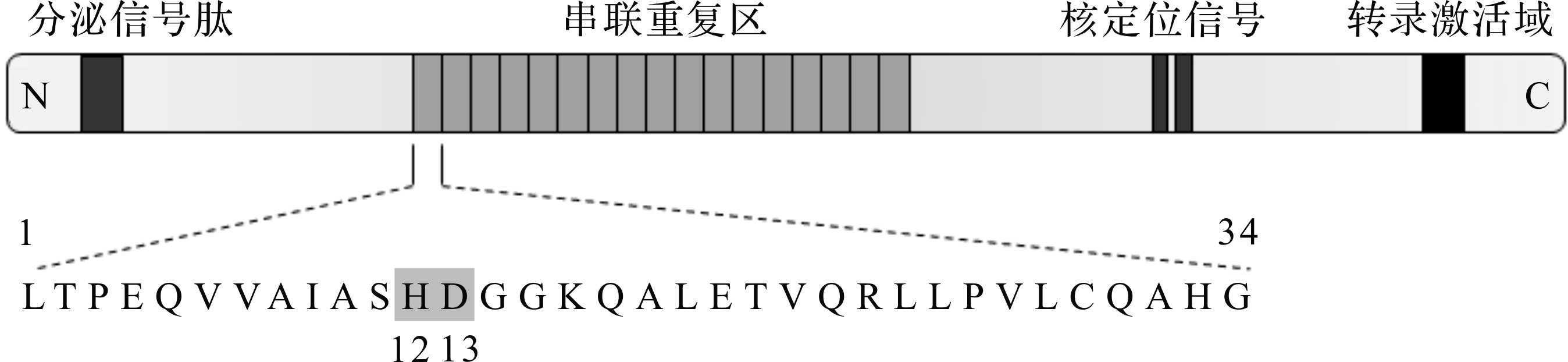
Fig.1 The structure of TALE protein
经研究,RVDs的识别关系主要为NI=腺嘌呤、NH=鸟嘌呤、HD=胞嘧啶、NN=腺嘌呤或者鸟嘌呤、NG=胸腺嘧啶,NS对4种碱基都可以识别[4,5]。C端的核定位信号主要负责将TALE引入宿主细胞核内,N端分泌信号肽负责TALE跨膜转运,转录激活结构域(AD)则负责启动靶标序列转录活动。因此,TALE蛋白进入宿主细胞后,由NLS引导靠近细胞核,在N端分泌信号肽协助下进入核内。然后,根据不同RVDs,串联重复序列识别并结合到宿主特定DNA位点,AD则激活靶点附近基因转录活动,引起宿主基因表达异常,从而导致宿主异常发育或感染病害。
随着TALE结构与功能的深入解析,TALEN基因编辑技术出现,并逐步取代ZFN技术。TALEN复合物主要包含两部分:1) 用于识别与定位基因组靶标序列的TALE;2) 用于切割基因组靶标序列的非特异性核酸内切结构域Fok I如图2(a)所示。Fok I来自海床黄杆菌,为IIS型内切酶,该酶只在二聚体状态时才有活性,行使靶序列切割功能。Fok I与TALEs的C端相连,由TALEs导向靶标序列。为使Fok I具有切割活性,TALEN二聚体需分别附着于双链两侧,且中间预留出14~20 bp的切割空间如图2(a)所示[8]。TALEN技术的工作原理与ZFN近似,首先是由TALE蛋白靶向识别宿主DNA序列,然后Fok I切割目标序列,产生双链断裂(Double-strand breaks,DSBs),激活细胞内源性DNA修复机制引入突变。当细胞内存在DSB侧翼序列的同源序列时,引发同源重组(Homologous recombination,HR)修复,可将外源DNA片段导入宿主目标DNA区域,形成插入突变;反之,细胞内不存在同源序列时,引发非同源末端连接(Non-homologous end joining,NHEJ)修复,NHEJ修复易导致DSB侧翼序列接合时发生核苷酸的缺失、插入或改变,形成无法预料的位点突变[4,5]如图2(c)所示。
图2 基因编辑工作原理
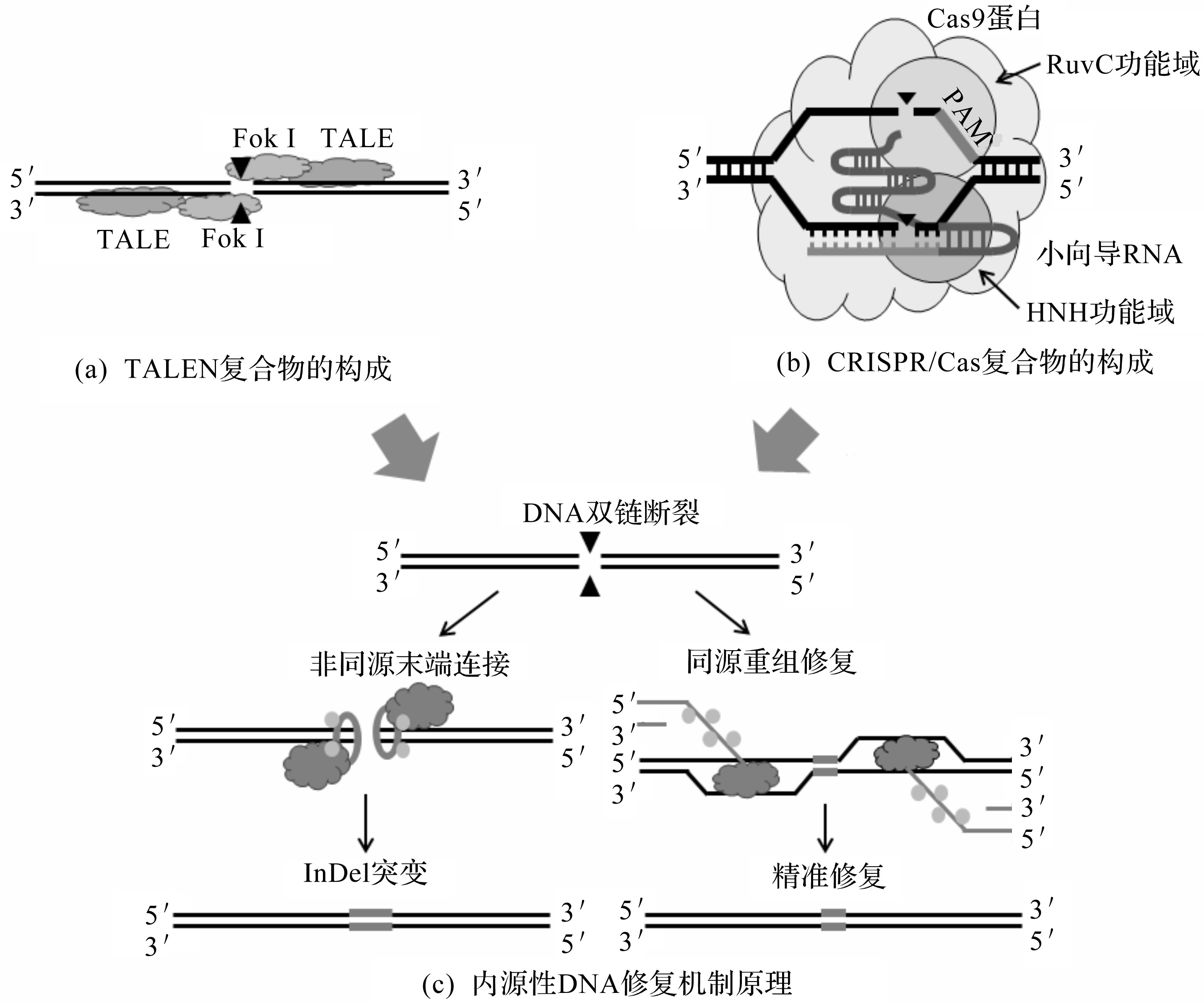
Fig.2 Operating machinery of gene editing
1.2、 应用进展
TALEN技术出现后,工业微生物育种学家们也展开了新技术的应用探索。Li等[9]9]基于TALEN的4个最常见RVDs(NI,NG,HD,NN)开发出一种“模块组装”方法,称为designer TALEN(dTALEN)。通过该方法设计了10个dTALEN,测试结果表明:10个dTALEN均获得了预期突变菌株,突变菌株中只有较低的非靶向突变,无细胞毒性。dTALEN方法极大地提高了酵母菌株基因编辑效率,为微生物基因改良奠定了基础;Arazoe等[10]10]首次将TALEN技术应用到稻瘟病菌(Pyricularia oryzae)基因编辑中,研究结果表明:通过同源重组成功将SRS2基因替换为潮霉素B抗性基因hph,且基因替换效率高达100%;叶伟等[11]11]在中国菌物学会学术年会上报道,利用TALEN技术成功将啤酒酵母菌株AS2.4的ADH2基因敲除,该基因编码乙醇脱氢酶。LC-MS和GC检测结果表明:基因敲除菌株的乙醇产量提高了50%左右,极大地促进了酿酒酵母的遗传改良;米曲霉菌是发酵食品饮料生产和食品加工的重要商业用酶。Mizutani等[12]首次将TALEN技术应用到米曲霉菌(Aspergillus oryzae)遗传改良中,发现在正常野生型菌株中,基因编辑材料约有一半为靶标区域的大片段缺失,而在LigD缺失型的NEHJ功能缺失菌株中,基因编辑材料的大片段缺失率明显降低。黑曲霉(Aspergillus niger)CBS 513.88是重要的有机酸、酶类工业生产菌株,李丽莎[13]利用TALEN技术对该菌株进行GlaA基因敲除,敲除后的菌株产糖化酶能力降低。将TALEN技术与电击转化法相结合,构建了成熟的A.niger CBS 513.88转化体系,为丝状真菌基因编辑应用提供了宝贵的技术支持,TALEN技术的应用,极大地促进了工业微生物精准育种进程。
2 、成簇的规律间隔的短回文重复序列相关蛋白技术
2.1 、技术原理
噬菌体感染是细菌生存的一大压力,海洋中每天约有4%~50%的细菌死于噬菌体攻击[14]。为避免噬菌体感染,细菌进化出包括CRISPR/Cas系统等获得性免疫系统[15]。研究发现:CRISPR/Cas系统分为Ⅰ~Ⅴ型,其中最常用的Cas9蛋白属Ⅱ型系统[16]。随后,对CRISPR/Cas结构功能与技术原理解析、技术优化研究相继开展。
CRISPR/Cas系统由CRISPR序列和Cas基因序列组成。CRISPR是一段具有前导区(leader sequence)和重复-间隔区(repeat-spacer)的DNA序列如图3所示[17]。前导区为一段富含AT的序列,被认为是CRISPR簇的启动子区。重复序列(repeats)是一段有回文或发卡结构的21~48 bp的序列,间隔序列(spacers)是一段特异性序列,其决定了CRISPR/Cas系统的靶向性。Cas基因序列经转录翻译合成CAS家族蛋白,CAS蛋白具有核酸内切酶活性、解旋酶活性和DNA结合功能域。其核酸酶结构域为HNH和RuvC,分别负责切断靶标序列的互补链与非互补链。
图3 CRISPR位点结构
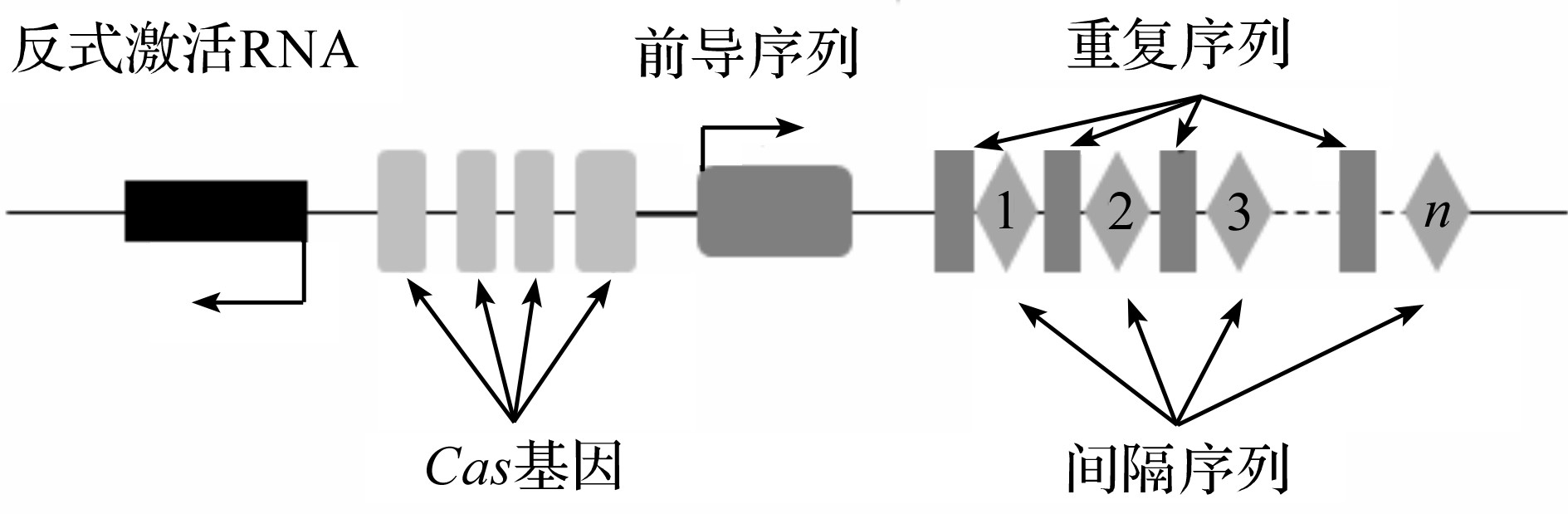
Fig.3 The structure of CRISPR loci
与TALEN技术原理不同,CRISPR/Cas系统以转录得到的crRNA为向导,与CAS蛋白形成CRISPR效应核蛋白复合体(crRNP),crRNP在crRNA的引导下与靶序列结合,并识别靶序列周边的原间隔相邻基序(Protospacer-adjacent motifs,PAM),在NGG-PAM前3~8 bp位置处进行切割,产生双链断裂诱发宿主细胞内源性HR修复或NHEJ修复引入突变见图2(b,c))[2,18,19]。
CRISPR技术出现后,在遗传病治疗、遗传育种改良和基因组研究上展现出极大的应用价值和潜力,全球科学家也纷纷开展CRISPR/Cas9的技术升级与应用研究。Cas9D10A突变体为RuvC失活和HNH激活;Cas9H840A突变体则为RuvC激活和HNH失活[20,21]。这2种Cas9突变体都有酶切活性,可对靶向序列进行剪切。dCas9(dead Cas9)的RuvC和HNH功能域同时失活,不具有核酸酶活性,但仍有DNA解旋与结合功能[22]。科学家通过将tRNA腺苷脱氨酶、胞苷脱氨酶分别与dCas9形成融合蛋白,成功实现了A与G和T与C间的相互转换[23,24]。为消除对NGG-PAM的依赖性,科学家设计出化脓性链球菌Cas9(SpCas9)变种SpG,该变异株能够靶向NGN-PAMs,并进一步优化该酶以开发一种几近无PAM依赖性的SpCas9变异株SpRY(NRN>NYN PAMs),极大地增加了CRISPR/Cas9的靶点范围[25]。此外,科学家还开发出CRISPR/Cas12a,CRISPR/Cas13a和CRISPR/Cpf1等其他CRISPR系统,拓宽了CRISPR/Cas系统应用范围。例如CRISPR/Cpf1系统,相较Cas9蛋白,Cpf1体积更小,更利于转化宿主。同时,Cpf1剪切产生黏性末端,更利于基因编辑后的修复,进一步扩充了靶点范围[26]。Wu等[27]更是利用CRISPR/Cpf1构建反式酶切体系,结合LAMP反应搭建了转基因大豆荧光检测系统,为快检行业提供了新思路。
2.2 、应用进展
在工业微生物育种研究中,CRISPR技术的应用也非常广泛,酿酒酵母、产甘油假丝酵母、谷氨酸棒状杆菌等重要微生物工业菌种的CRISPR技术体系相继建立。Dicarlo等[28]28]首次在模式生物酿酒酵母中构建CRISPR/Cas9系统,通过组成型表达Cas9蛋白与瞬时表达gRNA,为酵母基因编辑提供了一个高效的工具。研究表明:通过上述系统靶向双链断裂可使单链和双链供体的同源重组率分别提高5倍和130倍。Tsai等[29]29]为了向酿酒酵母导入外源木糖还原酶、木糖醇脱氢酶和木糖激酶基因(XYL1,XYL2,XYL3)并删除内源PHO13和ALD6,构建了一个以PHO13和ALD6为靶点的CRISPR/Cas9质粒,与含有XYL1,XYL2和XYL3的线性化靶点DNA同源片段共转化进酿酒酵母。最终,XYL1,XYL2和XYL3被整合到PHO13和ALD6的基因座上,实现了外源基因的插入和内源基因的敲除。该研究通过CRISPR/Cas9技术将来源于树干毕赤酵母的木糖合成相关基因导入酿酒酵母,并删除PHO13和ALD6,构建出一株无抗性筛选标记、木糖发酵能力强的工程菌株,该菌株非常适合工业化发酵使用。
甘油在医学、化工、食品和化妆品等行业均有重要应用。产甘油假丝酵母可以合成高浓度甘油,同时具有繁殖速度快、抗逆性强和转化率高等优点,是我国甘油工业化发酵生产的重要菌株,也是用于遗传改造的优良底盘细胞。朱敏阳等[30]构建了产甘油假丝酵母CRISPR/CgCas9瞬时表达系统,以TRP1和URA3为靶点设计sgRNA,未形成CgCas9-sgRNA复合体,而是通过单体元件形式导入宿主,实现了双基因敲除;通过在TRP1基因位点编辑表达木糖脱氢酶基因xylB获得了木糖酸生产菌株。该研究还表明同源臂长度在50 bp时修复效率最高。
谷氨酸棒状杆菌是目前全球氨基酸发酵工业的主要生产用菌,但无法引入CRISPR/SpCas9基因编辑系统。Jiang等[31]则通过CRISPR-FnCpf1系统对谷氨酸棒状杆菌的g-谷氨酰激酶G149进行密码子饱和突变减轻了L-脯氨酸抑制,使谷氨酸棒状杆菌基因编辑育种成为可能。夏婷婷[32]实验室收集到一株维吉尼亚链霉菌IBL14,经测序发现其基因组存在I-B-Svi型CRISPR/Cas系统,经验证,来源于IBL14的CRISPR-SviCas3-5-7和CRISPR-SviCas3系统可以在谷氨酸棒状杆菌中进行基因编辑,但CRISPR-SviCas3编辑效率较低,而SpCas9作为对照未出现基因编辑。该研究为谷氨酸棒状杆菌基因编辑提供了另一新工具,同时也为其他SpCas9不兼容菌株提供了一个新的可能。
光合微生物作为自然界重要的存在,具有高效能、高营养和分布广泛等特点应用于养殖、环保和医疗等行业,是重要的工业微生物。Jiang等[33]首次在光合微生物莱茵衣藻中应用CRISPR/Cas9技术,发现Cas9单独转化到莱茵衣藻中存在毒性效应,仅获得了一个成功编辑的克隆,仍为光合微生物基因编辑打开了思路。此后,科学家们在微拟球藻、三角褐指藻、海链藻、长链链球菌和集胞藻等光合微生物中相继建立了CRISPR/Cas9基因编辑体系[34]。
3 、TALEN与CRISPR/Cas9技术对比
TALEN技术与CRISPR技术均为近年来的热点基因编辑技术,在基因治疗、基因功能研究和种质遗传改良等领域均有广泛的应用。这2个技术体系各有优势,对比结果如表1所示。
表1 TALEN与CRISPR/Cas9技术对比
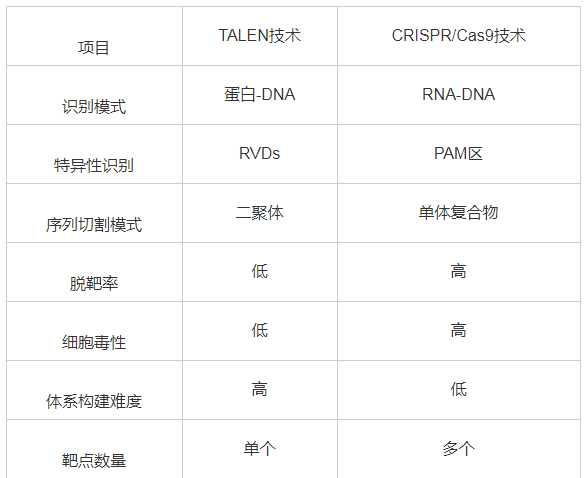
由表1可知:1) TALEN技术以蛋白-DNA模式识别靶标序列,CRISPR技术为RNA-DNA识别模式。TALEN技术需要根据靶标序列构建不同的TALEs蛋白,体系构建耗时耗力且成本较高,而CRISPR技术只需改变一段短sgRNA序列即可实现靶标位点识别功能,体系构建更加简单高效。同时,由于蛋白与DNA结合效果受DNA甲基化影响,TALEN技术的靶点范围受限于DNA甲基化,CRISPR技术的RNA-DNA识别模式则不受DNA甲基化影响。2) TALEN技术靶标序列的特异性由串联重复序列结构域的RVDs决定,这4种核苷酸均有相应RVDs进行识别。理论来讲,TALEN可以靶向基因组的任何一段特异性序列。CRISPR技术则通过识别PAM区进行定点编辑,虽经科学家改造,PAM区对靶点序列识别的范围增大很多,但仍受靶点范围限制。3) TALEN技术只有形成二聚体才能进行基因组编辑,但TALEN的二聚体基因编辑模式决定了其靶点特异性非常高,编辑效率高,脱靶率低,对细胞的毒性效果较低。CRISPR只需单个CRISPR/Cas9蛋白复合体即可启动定点编辑,但存在较高的脱靶率,易出现预料之外的编辑效果,有较高的细胞毒性作用。因此,TALEN技术在微生物发酵食品工业方向具有较高的应用潜力。4) TALEN主要用于基因片段编辑,尚不能实现单碱基编辑功能。CRISPR技术则通过融合tRNA腺苷脱氨酶、胞苷脱氨酶实现了A与G和T与C间的单位点精确编辑,随着基因功能位点的精确研究,CRISPR在精准位点编辑上的应用前景更广阔。5) TALEN技术每次只能进行单个靶点的基因编辑,而CRISPR技术可以同时进行多靶点定向基因编辑,在创制微生物育种突变体材料时更加高效。
4 、基因编辑技术应用面临的问题
基因编辑技术虽然应用前景广阔,但在技术提升与工业微生物遗传改良应用上仍面临诸多困难,如存在脱靶效果、细胞毒性、传递系统的效率和安全性、微生物的系统不兼容性等。
CRISPR/Cas技术虽然依靠融合腺苷脱氨酶、胞苷脱氨酶实现A与G和T与C转换,但未能实现嘌呤与嘧啶间的自由转换。脱靶效果也是限制CRISPR系统应用的一大问题,缩短sgRNA可以一定程度上提高靶点结合效率,但sgRNA过短也会降低靶点的特异性。Davis等[35]将4-羟基三苯氧胺反应性内含肽插入Cas9特定位置,发现Cas9编辑靶基因位点的特异性比野生型Cas9高25倍,提高靶向效率依旧是基因编辑应用必须解决的问题之一。
常见的传递载体工具主要有质粒、病毒和蛋白质等。病毒载体虽然传递效率高,但对转运对象的大小有要求;质粒载体可承载大片段转运,适合多靶点编辑,但同样会面临携带外基因同源片段进入宿主内的问题;蛋白核酸复合物作为常用载体,普遍认为其安全高效,但持续表达时间较短[36]。如何将基因编辑系统的高效安全地传递进宿主,并在编辑事件完成后从细胞中完全清除也需要进一步探索。
据统计,已知的微生物代谢产物虽然有近1 000种,但工业化生产应用的仅100种左右;微生物的酶也有近1 000种,但工业化生产应用的仅10种。很多微生物由于自身的遗传特性限制,尚不能被开发应用。目前,研究人员仅在少数工业微生物中建立了基因编辑系统[37,38],但微生物种类繁多,且很多物种具有系统不兼容性。因此,改造与开发更多高兼容性核酸酶,并在各微生物中建立成熟的应用体系,对充分利用微生物资源,创造高价值的工业微生物菌种有重要意义。
5、 结 论
优良菌种是微生物高效工业化生产的基础,菌种遗传改良技术决定了育种的效率。传统育种技术由于突变方向和性状随机性问题,致使优良菌种筛选效率低、过程繁琐。同时,射线辐射与诱变剂等物理化学诱变方法存在一定的安全隐患,对环境防护及操作者安全防护有一定要求。此外,通过传统方法将外源基因表达模块整合到菌株中,不可避免地会在酵母染色体上沉积不良的遗传元素,如质粒携带的多克隆位点和抗生素抗性基因之类的不良标记。由于健康问题,这些用于筛选菌株的耐药标记可能会阻碍菌株的工业化应用。TALEN和CRISPR技术作为定向遗传改良技术的代表,具有高度靶向性、无需筛选标记、体外组装、操作简单高效和单核苷酸精准编辑等优势,对环境和操作者几乎无防护要求,安全度更高,是未来工业微生物遗传改良的主流技术。综观近年来工业微生物育种研究报道,研究人员正在积极探索和研究TALEN技术和CRISPR技术在微生物育种中的应用。随着技术体系的优化和工业微生物基因功能位点的深入研究,基因编辑技术将在遗传改良方面发挥更大的作用,尽管近年已有基因编辑技术在微生物育种应用方面的综述,但CRISPR/Cas技术研究与应用进展极快,需不断更新。希望通过本综述,让育种者了解基因编辑技术最新进展及在工业微生物育种研究上的应用情况,以更好地应用基因编辑技术提升工业微生物育种效率。
参考文献
[1] 房耀维,范琳,牛艳芳,等.工业微生物育种技术研究进展[J].内蒙古师范大学学报(自然科学版),2003(2):158-161.
[2] HSU P D,LANDER E S,ZHANG F.Development and applications of CRISPR-Cas9 for genome engineering[J].Cell,2014,157(6):1262-1278.
[3] SUGIO A,YANG B,ZHU T,et al.Two type Ⅲ effector genes of Xanthomonas oryzae pv.oryzae control the induction of the host genes OsTFIIAgamma1 and OsTFX1 during bacterial blight of rice[J].Proceedings of the national academy of sciences of the United States of America,2007,104(25):10720-10725.
[4] BOCH J,SCHOLZE H,SCHORNACK S,et al.Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors[J].Science,2009,326(5959):1509-1512.
[5] MOSCOU M J,BOGDANOVE A J.A simple cipher governs DNA recognition by TAL effectors[J].Science,2009,326(5959):1501.
[6] MAK A N,BRADLEY P,CERNADAS R A,et al.The crystal structure of TAL effector PthXo1 bound to its DNA target[J].Science,2012,335(6069):716-719.
[7] DENG D,YAN C,PAN X,et al.Structural basis for sequence-specific recognition of DNA by TAL effectors[J].Science,2012,335(6069):720-723.
[8] LEE H B,SUNDBERG B N,SIGAFOOS A N,et al.Genome engineering with TALE and CRISPR systems in neuroscience[J].Frontiers in genetics,2016 (7):47.
[9] LI T,HUANG S,ZHAO X,et al.Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes[J].Nucleic acids research,2011,39(14):6315-6325.
[10] ARAZOE T,OGAWA T,MIYOSHI K,et al.Tailor-made TALEN system for highly efficient targeted gene replacement in the rice blast fungus[J].Biotechnology and bioengineering,2015,112(7):1335-1342.
[11] 叶伟,章卫民,刘桃妹,等.TALEN技术在提高啤酒酵母乙醇产量中的应用[C]//中国菌物学会.中国菌物学会2015年学术年会论文摘要集.上海:中国菌物学会,2015:105.
[12] MIZUTANI O,ARAZOE T,TOSHIDA K,et al.Detailed analysis of targeted gene mutations caused by the Platinum-Fungal TALENs in Aspergillus oryzae RIB40 strain and a ligD disruptant[J].Journal of bioscience and bioengineering,2017,123(3):287-293.
[13] 李丽莎.基于TALENs的丝状真菌基因组精确编辑[D].福州:福州大学,2017.
[14] BREITBART M,ROHWER F.Here a virus,there a virus,everywhere the same virus[J].Trends in microbiology,2005,13(6):278-284.
[15] SOREK R,LAWRENCE C M,WIEDENHEFT B.CRISPR-mediated adaptive immune systems in bacteria and archaea[J].Annual review of biochemistry,2013(82):237-266.
[16] MAKAROVA K S,WOLF Y I,ALKHNBASHI O S,et al.An updated evolutionary classification of CRISPR-Cas systems[J].Nature reviews microbiology,2015,13(11):722-736.
[17] 李聪,曹文广.CRISPR/Cas9介导的基因编辑技术研究进展[J].生物工程学报,2015,31(11):1531-1542.
[18] FRIEDLAND A E,TZUR Y B,ESVELT K M,et al.Heritable genome editing in C.elegans via a CRISPR-Cas9 system[J].Nature methods,2013,10(8):741-743.
[19] STERNBERG S H,REDDING S,JINEK M,et al.DNA interrogation by the CRISPR RNA-guided endonuclease Cas9[J].Nature,2014,507(7490):62-67.
[20] MCCAFFREY J,SIBERT J,ZHANG B,et al.CRISPR-CAS9 D10A nickase target-specific fluorescent labeling of double strand DNA for whole genome mapping and structural variation analysis[J].Nucleic acids research,2016,44(2):11.
[21] CHEN N,HU Z,YANG Y,et al.Inactive Cas9 blocks vitreous-induced expression of Mdm2 and proliferation and survival of retinal pigment epithelial cells[J].Experimental eye research,2019,186:1-7.
[22] BROCKEN D J W,TARK-DAME M,DAME R T.dCas9:a versatile tool for epigenome editing[J].Current issues in molecular biology,2018(26):15-32.
[23] KOMOR A C,KIM Y B,PACKER M S,et al.Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J].Nature,2016,533(7603):420-424.
[24] GAUDELLI N M,KOMOR A C,REES H A,et al.Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J].Nature,2017,551(7681):464-471.
[25] WALTON R T,CHRISTIE K A,WHITTAKER M N,et al.Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants[J].Science,2020,368(6488):290-296.
[26] DONG D,REN K,QIU X,et al.The crystal structure of Cpf1 in complex with CRISPR RNA[J].Nature,2016,532(7600):522-526.
[27] WU H,HE J S,ZHANG F,et al.Contamination-free visual detection of CaMV35S promoter amplicon using CRISPR/Cas12a coupled with a designed reaction vessel:rapid,specific and sensitive[J].Analytica chimica acta,2020,1096:130-137.
[28] DICARLO J E,NORVILLE J E,MALI P,et al.Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J].Nucleic acids research,2013,41(7):4336-4343.
[29] TSAI C S,KONG I I,LESMANA A,et al.Rapid and marker-free refactoring of xylose-fermenting yeast strains with Cas9/CRISPR[J].Biotechnology and bioengineering,2015,112(11):2406-2411.
[30] 朱敏阳.产甘油假丝酵母CRISPR-Cas9基因编辑系统的构建[D].无锡:江南大学,2019.
[31] JIANG Y,QIAN F,YANG J,et al.CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J].Nature communications,2017(8):15179.
[32] 夏婷婷.I-B-Svi型CRISPR-Cas系统在谷氨酸棒状杆菌中的基因编辑[D].合肥:安徽大学,2019.
[33] JIANG W,BRUEGGEMAN A J,HORKEN K M,et al.Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii[J].Eukaryotic cell,2014,13(11):1465-1469.
[34] NADUTHODI M I S,BARBOSA M J,VAN DER OOST J.Progress of CRISPR-Cas based genome editing in photosynthetic microbes[J].Biotechnology journal,2018,13(9):1-9.
[35] DAVIS K M,PATTANAYAK V,THOMPSON D B,ZURIS J A,LIU D R.Small molecule-triggered cas9 protein with improved genome-editing specificity[J].Nature chemical biology,2015,11(5):316-318.
[36] ZISCHEWSKI J,FISCHER R,BORTESI L.Detection of on-target and off-target mutations generated by CRISPR/Cas9 and other sequence-specific nucleases[J].Biotechnology advances,2016,35(1):95-104.
[37] 程妙文,罗玮,杜瑶.CRISPR-CAS系统介导的新一代基因靶向修饰技术及其在工业微生物中的应用[J].微生物学报,2017,57(11):1621-1633.
[38] 张驰,顾徽瑜,陆玲.曲霉高效基因编辑技术研究进展[J].菌物学报,2018,37(10):1349-1356.