摘 要: 【背景】以往双歧杆菌质粒分类学研究较少,双歧杆菌属质粒系统分类方法缺失。【目的】建立双歧杆菌属天然质粒系统分类和鉴定方法,促进质粒在双歧杆菌生物学研究中的理解和应用。【方法】利用质粒复制起始蛋白进化树和基因组共线性分析方法,对目前所有已测序的双歧杆菌属天然质粒进行系统分类研究。【结果】双歧杆菌所有已知天然质粒可以划分为6个不同类型的质粒家族和3个独特的复合质粒,家族III和家族VI质粒可进一步划分为不同的亚型类群。家族III质粒是双歧杆菌属天然质粒的主要类型和优势家族。家族VI质粒成员亚型分类最丰富。在质粒家族水平,两种方法的分类结果完全一致。【结论】本文全面、准确地揭示了所有分析质粒之间的系统分类关系,建立了双歧杆菌属天然质粒系统的分类标准、方法和体系,可为今后双歧杆菌天然质粒分类和鉴定提供重要的理论参考和分类依据。
关键词: 双歧杆菌属; 质粒分类; 复制起始蛋白; 基因组共线性; 复制子;
Abstract: [Background] In the past, there were few studies on the taxonomy of bifidobacteria plasmids, and the phylogenetic classification method of Bifidobacterium plasmid was missing. [Objective] In order to establish a method for phylogenetic classification and identification of natural plasmid in Bifidobacterium, and increase understanding and application of plasmids in the research of bifidobacteria biology, [Methods] we conducted to phylogenetic classification of all sequenced plasmids in Bifidobacterium by the analysis of phylogenetic relationship of replication initiation protein and genome colinearity. [Results] The results showed that all known natural plasmids of Bifidobacterium were divided into six different types of plasmid families and three unique complex plasmids. Plasmids of type III and VI family were further divided into different subtype groups. Plasmids of the type III and VI family can be further divided into different subtype groups. Plasmids of the type III family were the main type and dominant family of natural plasmids in Bifidobacterium. Plasmid subtypes of type VI family were most abundantly classified. At the plasmid family level, the classification results of the two methods were completely identical. [Conclusion] These results comprehensively and accurately revealed the phylogenetic classification relationship between all the plasmids that were analyzed, and established the phylogenetic classification standard and method of natural plasmid in Bifidobacterium, which provided important theoretical references and classification basis for the future classification and identification of natural plasmids in Bifidobacterium.
Keyword: Bifidobacterium; Plasmid classification; Replication initiation protein; Genome colinearity; Replicon;
双歧杆菌常发现于人和动物的消化道、生殖道和口腔等环境中,是人和动物体内重要的共生菌。双歧杆菌可以通过抑制致病菌生长、增强免疫应答、抑制肿瘤血管生成和改善营养等方式,在维持和调节体内微生态平衡、促进宿主健康方面发挥着广泛且积极的生理作用[1,2,3]。双歧杆菌被证实可在几种实体瘤中选择性定位和增殖,可用于癌症基因治疗中高度特异的基因递送载体[4,5]。研究表明,双歧杆菌与人和动物饮食、健康和疾病紧密相关[6,7]。目前,双歧杆菌已在食品和医药行业广泛应用,产品种类众多,具有重要的经济价值和社会意义。
目前,双歧杆菌属已有70个菌(物)种和10个亚种被明确分离和鉴定(http://www.bacterio. net/bifidobacterium.html)。基于美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)的RefSeq数据库(截止2019-10-10),双歧杆菌属共有38个质粒基因组序列被收录,其中23个质粒来自长双歧杆菌(Bifidobacterium longum),15个质粒来自双歧杆菌属其他物种。这些数据表明,双歧杆菌属已知天然质粒数量稀少,资源比较匮乏,多数物种可能不携带天然质粒,质粒分布存在明显的物种依赖性。此外,以往双歧杆菌质粒研究主要涉及质粒分离、鉴定和载体构建[8],而关于双歧杆菌质粒分类的研究很少,至今在双歧杆菌“属”水平上开展的质粒系统分类学研究仍未见报道。

质粒分类一直是细菌质粒研究的基础与核心问题。由于质粒遗传多样性丰富、变异率高,至今没有一种通用且准确的方法可以将所有天然质粒进行分类[9]。目前,使用最广、最常见的分类方法仍是早期研究建立的质粒不相容性分类法[10]。该方法分类和鉴定质粒时,需要进行繁琐的不相容性功能验证实验,费时费力、效率低下。同时,质粒不相容性与复制、分配、维持多个质粒功能相关,分子机制复杂多样,至今没有被完全阐明,这进一步降低了该方法的有效性和准确性。因此,该方法已不能满足后基因组时代大量新测序质粒的分类和鉴定需求。近年来,以质粒复制子[11,12]或松弛酶[13]作为分子标记的质粒系统分类方法已有报道。前者理论上适用于所有天然质粒,但实际中该方法只在有害菌(肠杆菌科[12]、鲍曼不动杆菌[14]、肠球菌[15]、葡萄球菌[16])中得到了发展和应用,而在有益菌质粒中未见报道;后者由于靶基因限制,只适用于可转移质粒的分类,因此应用范围受限[13]。
双歧杆菌和乳杆菌是益生菌研究和应用的典型代表,但它们的亲缘关系(双歧杆菌属于放线菌门,乳杆菌属于厚壁菌门)和染色体(G+C)mol%含量(双歧杆菌约60%,乳杆菌约40%)相差甚远,因此,天然质粒的多样性和分类很可能也显着不同。之前,基于质粒复制起始蛋白(replication initiation protein,Rep)和/或质粒全基因组序列,我们建立了两种不同的天然质粒系统分类方法,并成功应用于乳杆菌属天然质粒分类[17,18,19]。但该方法能否适用于亲缘关系较远和高(G+C)mol%含量的双歧杆菌或其他细菌质粒分类还不得而知。本文以所有测序的双歧杆菌天然质粒作为研究对象,通过质粒Rep蛋白进化树分析和质粒基因组共线性分析,探索建立双歧杆菌天然质粒系统分类方法、分类标准和分类体系,为今后双歧杆菌天然质粒分类、鉴定和应用提供重要的理论参考和分类依据。同时,本研究可进一步评价质粒Rep蛋白和质粒基因组共线性分类法对高(G+C)mol%含量细菌天然质粒的分类效果,以期为今后建立一种通用、高效的细菌天然质粒分类方法提供更加充实的理论基础和实验依据。
1 、材料与方法
1.1 、质粒基因组数据
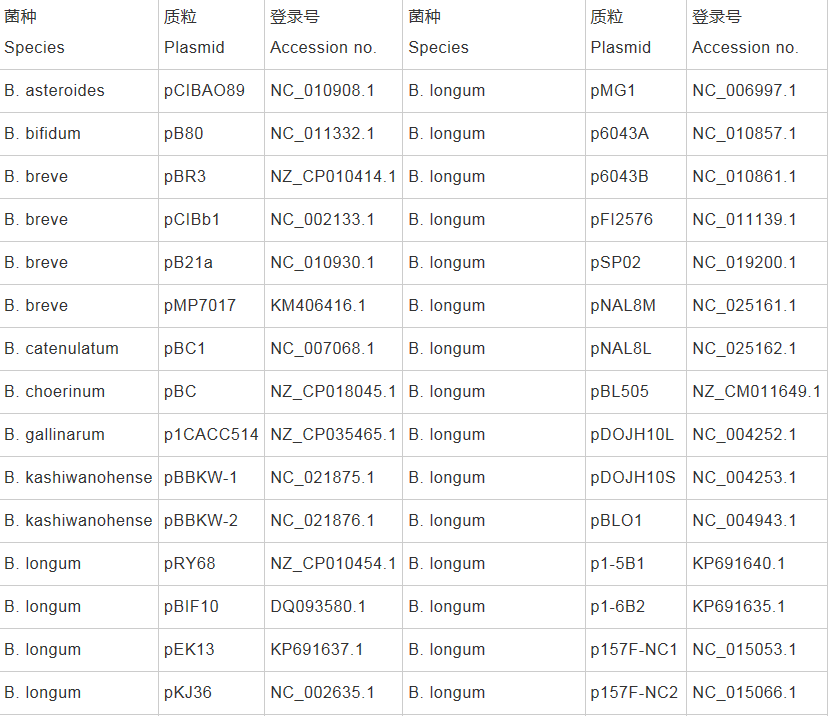
所有质粒基因组序列数据来自NCBI的Nucleotide和RefSeq数据库,序列详细信息见表1。
表1 Nucleotide和RefSeq数据库收录的双歧杆菌属质粒基因组序列
1.2 、方法
1.2.1、 质粒基因组序列的收集、整理和初步分析
检索Nucleotide和RefSeq数据库中所有收录的双歧杆菌质粒基因组序列,筛查获得完整基因组质粒及其Rep蛋白编码情况。对全部Rep蛋白序列进行BLAST序列比对分析,鉴定其保守结构域,初步确定蛋白家族。利用DNASTAR 7.1.0 EditSeq软件对核苷酸或氨基酸序列进行存储、编辑等处理。
1.2.2、 核苷酸和氨基酸多序列比对和重复序列分析
利用MEGA 7.0[20]软件中Muscle法进行核苷酸或氨基酸多序列比对,比对参数为默认设置。DNA重复序列分析利用DNASTAR 7.1.0 GeneQuest软件进行,最小序列长度设置为8 bp,间距设置为小于200 bp。
1.2.3、 质粒Rep蛋白系统进化树分析
利用MEGA 7.0软件中Neighbor-Joining (N-J)法进行质粒Rep蛋白系统进化树构建。系统进化树检验采用Bootstrap法,自展值设置为1 000,其他参数采用默认设置。
1.2.4、 质粒基因组共线性分析
利用Mauve 2.4.0[21]软件进行质粒基因组共线性分析,Match Seed Weight为13,其他参数均为默认设置。基因组共线性分析中,质粒基因组默认由小到大排列。
2 、结果与分析
2.1 、双歧杆菌天然质粒基本特征
经检索和筛查分析,目前Nucleotide数据库收录双歧杆菌质粒DNA序列103个(包括RefSeq数据库收录的38个双歧杆菌质粒DNA序列),其中41个质粒为完整基因组序列。统计分析发现,双歧杆菌41个完整质粒基因组总长387.77 kb,携带399个蛋白编码基因,大小范围为2.11-190.18 kb,平均(G+C)mol%含量为61.39%,大多数(27/41)质粒来自B. longum,大多数(60/70)物种尚未发现天然质粒(表2)。这些结果表明,双歧杆菌质粒普遍较小,大型质粒匮乏,平均(G+C)mol%含量接近染色体,质粒分布具有明显的物种依赖性,B. longum是双歧杆菌属中天然质粒多样性最丰富的物种。检索和BLAST分析发现,38个双歧杆菌质粒共编码42个Rep蛋白,其中35个质粒只含有1个Rep蛋白,3个复合质粒含有多个Rep蛋白;其余3个质粒没有编码已知的Rep蛋白;所有Rep蛋白根据氨基酸序列中的保守结构域可以初步划分为6个不同的蛋白家族,分别称为Rep_1、Rep_3、RepA_C、Rep*、Rep_trans和Viral_Rep (表2),这表明双歧杆菌大多数(38/41)质粒含有Rep蛋白,这些Rep蛋白序列具有一定的同源性和差异性,因此Rep蛋白可以为双歧杆菌质粒系统分类提供良好的分子标记和依据。
表2 双歧杆菌属质粒基因组和Rep蛋白基本特征
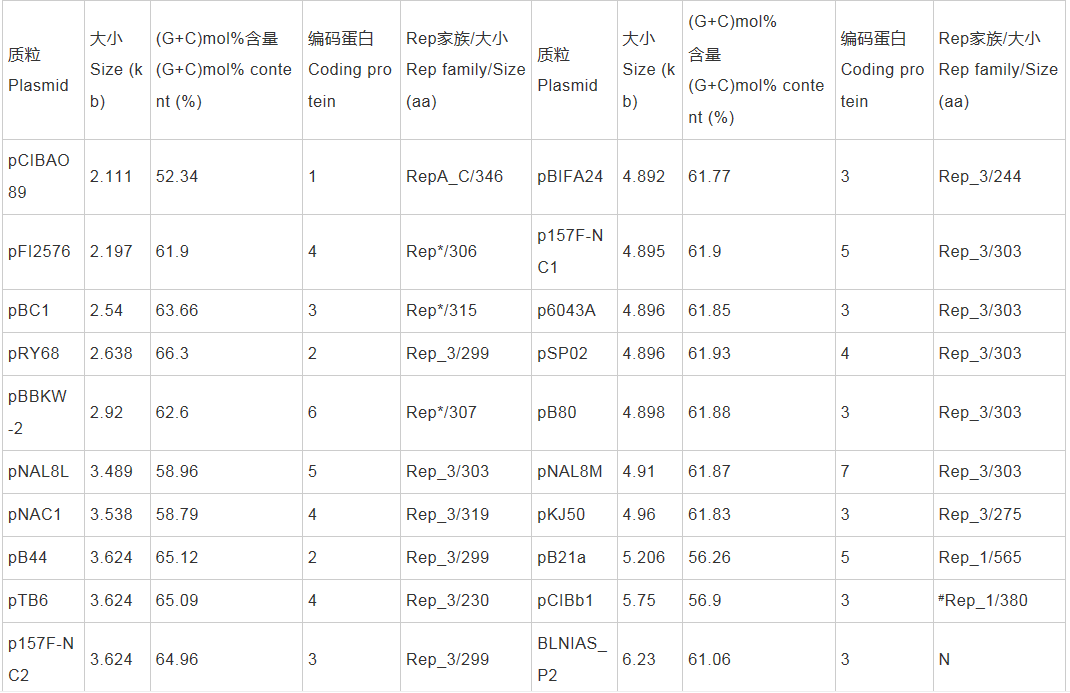
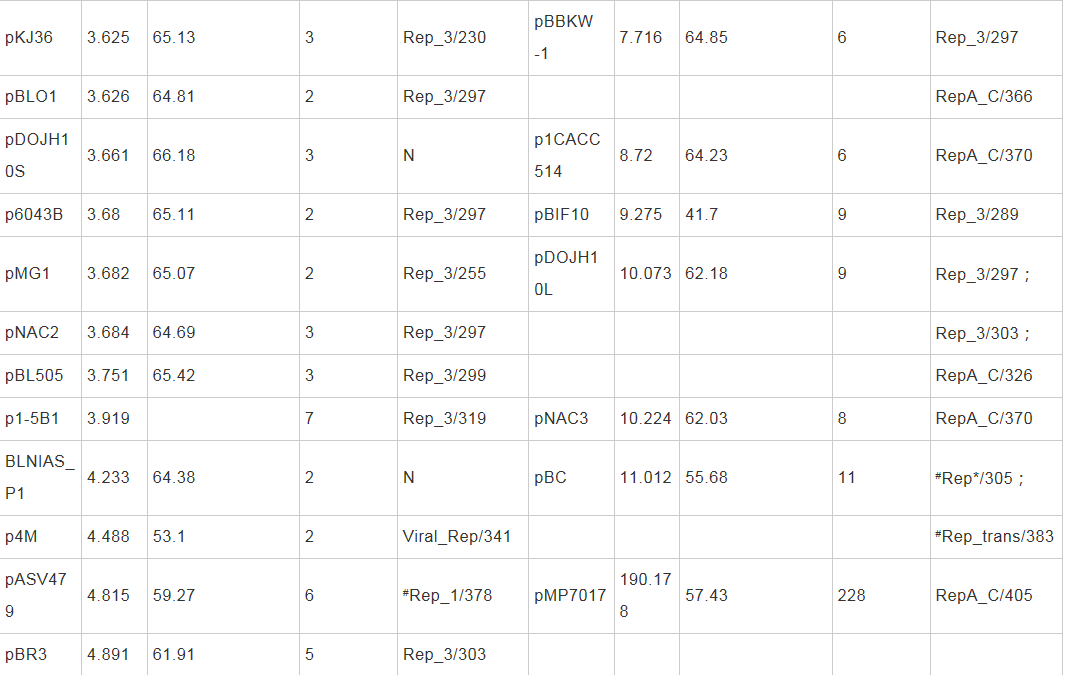
注:Rep_1:pfam01446;Rep_3:pfam01051;RepA_C:pfam04796;Rep*:pfam03090和pfam08708;Rep_trans:pfam02486;Viral_Rep:pfam02407;N:未编码已知Rep蛋白;#:本研究根据序列同源性推定的Rep蛋白。Note: Rep_1: pfam01446; Rep_3: pfam01051; RepA_C: pfam04796; Rep*: pfam03090 and pfam08708; Rep_trans: pfam02486; Viral_Rep: pfam02407; N: No known Rep has been encoded; #: Putative Rep by sequence similarity in this study.
2.2、 双歧杆菌质粒Rep蛋白进化树分类
基于Rep蛋白氨基酸序列的同源性,构建了双歧杆菌质粒Rep蛋白进化树,结果见图1。根据进化树拓扑结构和主要分支点置信度,双歧杆菌质粒所有Rep蛋白明显聚类为7个不同的蛋白家族,除了质粒pBIF10编码的Rep蛋白(AAZ79481.1),Rep蛋白进化树分类结果与前文Rep蛋白保守结构域分类结果一致。根据Rep蛋白与质粒的对应关系,35个编码单一Rep蛋白质粒根据Rep蛋白进化树可以明确划分为6个不同类型的质粒家族,其中质粒类型家族III是双歧杆菌天然质粒的主要类型和优势家族,根据进化树家族III分支的拓扑结构和主要分支点置信度,家族III质粒可以进一步明确划分为2个亚型类群(图1)。此外,尽管3个复合质粒不能划分到单一类型的质粒家族,但其可以明确划分到非单一类型的质粒家族。例如,复合质粒pDOJH10L含有Rep_3 (WP 011067901.1;WP 008783729.1)和RepA_C (WP 080504066.1)两类Rep蛋白,因此其可以同时归属于质粒家族III和家族VI (图1)。实际上,复合质粒很可能是由2个甚至更多不同质粒发生重组融合形成的,属于质粒进化的特殊阶段,因此,该方法对复合质粒的分类结果恰好体现了复合质粒基因组中不同复制子序列的进化起源,这进一步证明该方法对双歧杆菌质粒分类的有效性和准确性。总之,上述分析结果表明,双歧杆菌质粒Rep蛋白进化树分析比Rep蛋白保守结构域分析具有更高的分辨率和更好的分类效果,可以对双歧杆菌所有编码Rep蛋白天然质粒进行简单、有效和准确的系统分类,因此,Rep蛋白序列可以作为双歧杆菌所有编码Rep蛋白天然质粒系统分类理想的分类标准和分子标记。
图1 双歧杆菌属质粒Rep蛋白进化树
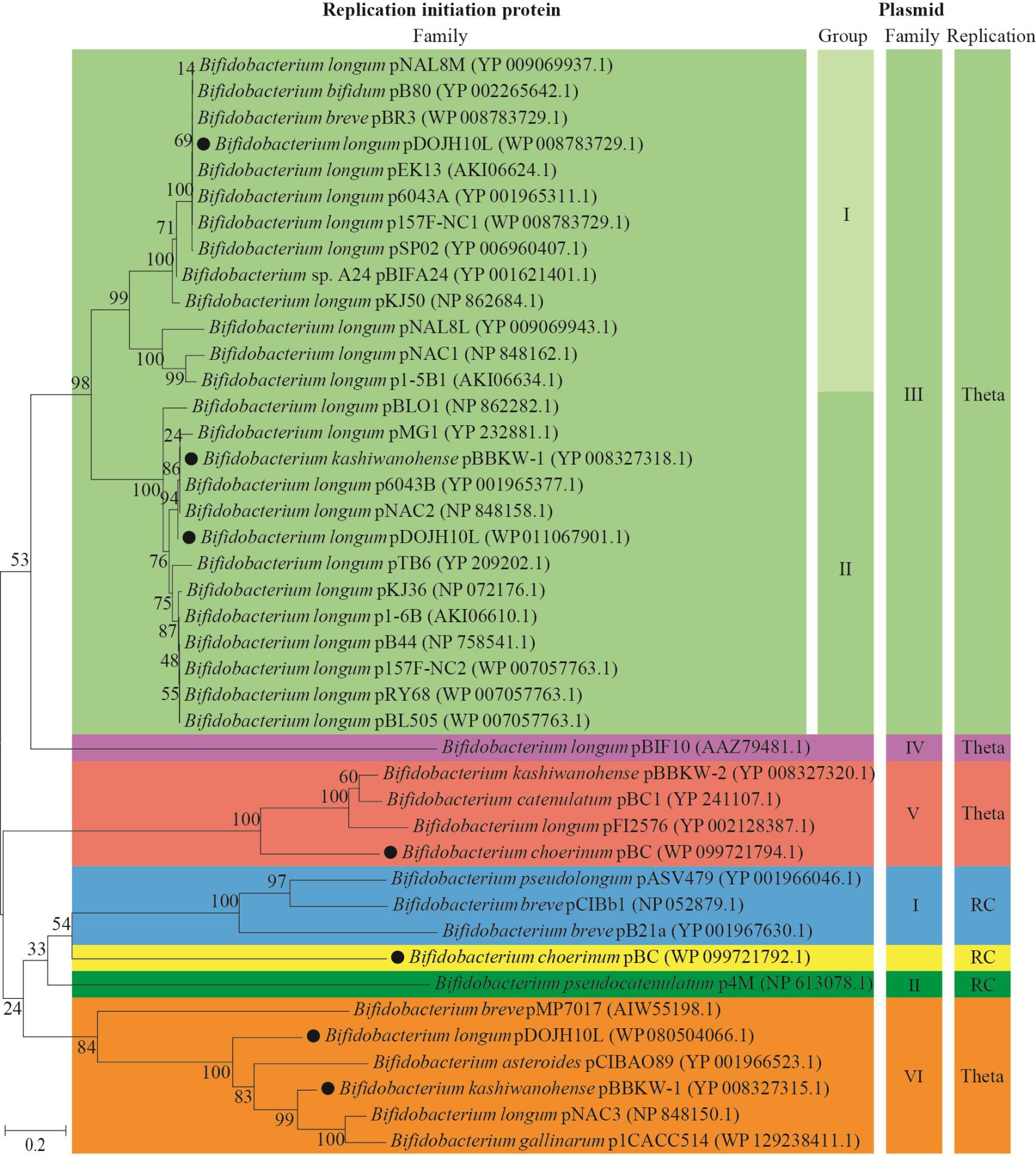
Figure 1 Phylogenetic tree of the plasmid Reps in Bifidobacterium
注:●:复合质粒Rep蛋白;RC:Rolling-circle;括号中字符为质粒编码Rep蛋白氨基酸序列登录号;分支点数字表示置信度;标尺表示遗传距离。Note: Black dot (●) indicates complex plasmid; RC indicates Rolling-Circle; The characters in parentheses are accession numbers of the plasmid-encoded Rep amino acid sequence; Branch point number indicates confidence; Scale indicates evolution distance.
2.3、 双歧杆菌质粒基因组共线性分类
由于复制子是天然质粒基因组唯一必需的遗传元件,因此,利用基因组共线性分析结果进行质粒系统分类时,同时将具有同源性复制子作为质粒家族分类的重要原则和标准。基于质粒基因组共线性和复制子序列分析,双歧杆菌属全部41个完整质粒系统分类结果如图2所示。
图2 双歧杆菌属质粒基因组共线性
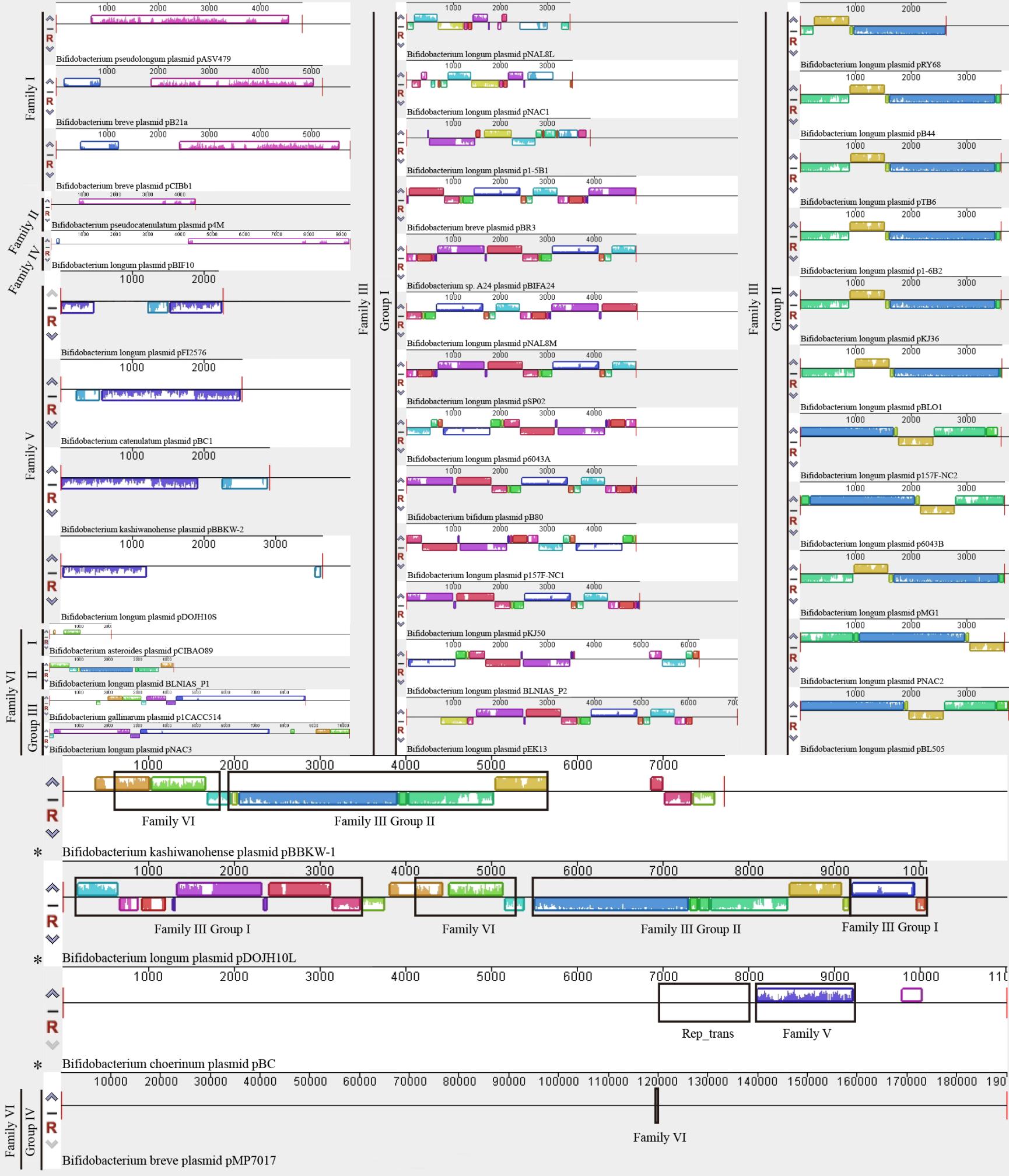
Figure 2 Genome colinearity of the Bifidobacterium plasmids
注:*:复合质粒;黑框:同源性区域。Note: Asterisk (*) indicates complex plasmid; Black rimmed indicates homology region.
2.3.1、 双歧杆菌属质粒家族I
经基因组共线性和复制子序列分析,发现质粒pASV479、pCIBb1、pB21a3含有高度同源的复制子和比较保守的基因组共线性,与其他双歧杆菌质粒没有显示同源性区域(图2)。它们的复制子均由同源的单链起点、双链起点和rep基因组成,Rep蛋白均含有保守结构域Rep_1 (pfam01446)。这些特征与已知滚环型复制pC194质粒家族[22]复制子高度相似。同时,O’Riordan等研究已证明,质粒pCIBb1采用滚环机制进行复制[23]。因此,基于基因组共线性和复制子序列特征,这3个质粒可以明确划分为同一个类型质粒家族,采用滚环机制复制,命名为双歧杆菌属质粒家族I。
2.3.2 、双歧杆菌属质粒家族II
基因组共线性分析显示,质粒p4M只与质粒pBIF10在复制子以外区域显示了一定的同源性,与其他双歧杆菌质粒没有显示任何同源性(图2)。复制子序列分析显示,质粒p4M的Rep蛋白含有保守结构域Viral_Rep (pfam02407),与GenBank数据库中众多来自动、植物单链DNA环状病毒的Rep蛋白显示高度一致性。目前,关于质粒p4M只有一篇文献报道[24],该研究表明,质粒p4M编码的Rep蛋白与多种动物环形病毒编码的Rep蛋白同属于一个滚环型复制的类质粒Rep蛋白家族,该家族蛋白很可能由跨物种的重组事件产生。基于基因组共线性和复制子序列特征,质粒p4M可以划分为一个独立类型的质粒家族,可能采用环形病毒相似的滚环机制复制,命名为双歧杆菌属质粒家族II。
2.3.3、 双歧杆菌属质粒家族III
经基因组共线性和复制子序列分析,发现25个质粒含有高度相似的复制子,除复制子区域外,这些质粒基因组共线性明显分为2个高度保守的亚型类群(图2)。复制子序列分析显示,这些质粒复制子均由rep基因及其上游的重复子序列组成,Rep蛋白均含有保守结构域Rep_3 (pfam01051),重复子均由3×10 bp或者4×10 bp和(3-5.5)×22 bp两组串联的正向重复序列组成。这些特征与已知Theta型复制pUCL287质粒家族[25]高度相似。因此,基于基因组共线性和复制子特征,这些质粒可以明确划分为同一个类型的质粒家族,可能采用Theta机制复制,命名为双歧杆菌属质粒家族III。根据基因组共线性的明显差异,可以进一步划分为2个亚型类群。另外,基因组共线性分析发现,尽管数据库中质粒BLNIAS_P2没有注释rep基因,但实际上该质粒编码了家族III质粒高度同源的重复子和rep基因,只是其rep基因编码区都发生了移码突变,不能产生完整的Rep蛋白。此外,尽管家族III所有质粒都含有一个典型的Theta型复制子序列,但质粒pNAL8L[26]、pNAC1[27]、pKJ36[28]、pMG1[29]、pSP02[30]、pKJ50[28,31]的DNA杂交实验结果显示,这些质粒复制过程中都存在单链中间体,而单链中间体是滚环复制质粒的典型特征[32]。因此,这些质粒真实的复制类型和机制仍然存疑,这些质粒的相关报道也都提及和论述了这一现象。这提示,家族III质粒可能存在一种未知的质粒复制机制。
2.3.4 、双歧杆菌属质粒家族IV
基因组共线性分析显示,质粒pBIF10只与家族II质粒p4M在复制子以外区域显示了一定的同源性,而与其他双歧杆菌质粒几乎没有显示同源性(图2)。复制子序列分析显示,质粒pBIF10复制子由rep基因及其上游的重复子序列组成,Rep蛋白含有保守结构域Rep_3 (pfam01051),重复子由3.5×20 bp一组串联的正向重复序列组成。这些结果表明,尽管质粒pBIF10的复制子结构和Rep蛋白结构域与家族III质粒相似,但该质粒Rep蛋白序列(图1)、重复子结构和基因组共线性(图2)均与家族III质粒明显不同,因此质粒pBIF10可以划分为一个独立类型的质粒家族,推测采用Theta机制复制[33],命名为双歧杆菌属质粒家族IV。
2.3.5 、双歧杆菌属质粒家族V
基因组共线性和复制子序列分析显示,pFI2576等4个质粒含有高度同源的复制子和高度保守的基因组区域,与其他双歧杆菌质粒没有显示同源性区域(图2)。它们的复制子由rep基因及其下游的复制起点序列组成,Rep蛋白均含有保守结构域pfam03090和pfam08708。复制子特征与已知的Theta型复制ColE3质粒家族[34]高度相似。同时,质粒pFI2576[35]、pBBKW-2[8]和pDOJH10S[36]都已经被实验证实采用Theta机制进行复制。此外,基因组共线性分析发现,质粒pDOJH10S实际上编码了rep基因,只是该基因发生了突变,形成了假基因。基于上述基因组共线性和复制子特征,这4个质粒可以明确划分为新的质粒家族,采用Theta机制复制,命名为双歧杆菌属质粒家族V。
2.3.6 、双歧杆菌属质粒家族VI
基因组共线性和复制子序列分析显示,pCIBAO89等5个质粒虽然基因组大小和共线性差异显着,但它们具有一个同源的复制子区域(图2)。该复制子由rep基因及其上游的重复子序列组成,Rep蛋白均含有保守结构域RepA_C (pfam04796),重复子序列、结构和间隔表现出较低的保守性。目前这5个质粒的复制机制没有得到实验验证,但根据质粒pCIBAO89和pNAC3的报道推测,它们可能采用Theta机制进行复制[27,37]。尽管这5个质粒的基因组大小和共线性差异显着,但基因组中同源性复制子的存在表明,它们很可能来自同一个祖先质粒,之后发生了分歧进化。因此,基于同源性复制子特征,这5个质粒可以划分为同一个类型的质粒家族,可能采用Theta机制复制,命名为双歧杆菌属质粒家族VI;同时,基于基因组共线性的明显差异,可以进一步划分为4个不同的亚型类群(图2)。另外,该家族不但包括双歧杆菌中最小的质粒pCIBAO89和唯一的巨型质粒pMP7017,并且该家族质粒亚型数量也是双歧杆菌中最多的,因此质粒家族VI很可能是双歧杆菌天然质粒中基因水平转移等重组进化事件发生最频繁的质粒家族。此外,与质粒BLNIAS_P2和pDOJH10S情况一样,基因组共线性分析发现,质粒BLNIAS_P1同样含有一个rep假基因。
2.3.7、 双歧杆菌属复合质粒
基因组共线性和复制子序列分析显示,复合质粒pBBKW-1同时含有质粒家族III和质粒家族VI类型的复制子,复合质粒pBC含有质粒家族V同源性复制子之外,还编码了一个独特的Rep_trans家族Rep蛋白,复合质粒pDOJH10L甚至同时含有3个完整的复制子(图2)。这些结果表明,这些复合质粒很可能在近期由2个不同类型质粒发生重组进化事件融合形成,这与质粒pBBKW-1和pDOJH10L的报道结果[8,36]一致。实际上,复合质粒只是天然质粒进化过程的一个特殊阶段,并非生存常态。鉴于复合质粒含有2个以上不同家族质粒的复制子区域,不能划分到单一类型的质粒家族,因此它们可以作为独特的质粒类型进行单独分类。
2.4 、双歧杆菌属天然质粒系统分类结果和特征
综合质粒Rep蛋白系统进化树和基因组共线性分析结果,双歧杆菌属所有41个天然质粒可以划分为6个不同类型和起源的质粒家族和3个独特的复合质粒,家族III和家族VI质粒可进一步划分为不同的亚型类群(图1和图2)。家族I、II质粒可能采用滚环机制复制,家族III-VI质粒可能采用Theta机制复制。家族III质粒成员数量最多,是双歧杆菌属天然质粒的主要类型和优势家族。家族VI质粒成员大小和亚型多样性最丰富,是双歧杆菌属天然质粒系统进化关系最复杂的质粒家族。在质粒家族水平两种方法分类结果完全一致,在亚家族水平基因组共线性分类法可以进行更加细致的亚型分类(例如家族VI质粒的亚型分类,图2)。两种独立方法的分类结果互为证明和验证,因此这些结果准确、有效地揭示了所有分析质粒之间的系统进化关系和对应的分类学地位,成功地建立了双歧杆菌属天然质粒系统分类标准、方法和体系,对今后双歧杆菌天然质粒分类和鉴定具有重要的参考价值和指导意义。
3 、结论与讨论
本文通过Rep蛋白进化树和基因组共线性两种分类方法,对目前已测序的双歧杆菌属天然质粒进行了系统分类研究。研究结果显示,质粒Rep蛋白进化树分类法可将双歧杆菌所有38个编码Rep蛋白天然质粒划分为6个不同类型的质粒家族和3个独特的复合质粒,家族III质粒可进一步划分为2个亚型类群(图1);质粒基因组共线性分类法可将双歧杆菌所有41个天然质粒划分为6个不同类型的质粒家族和3个独特的复合质粒,家族III和家族VI质粒可进一步划分为2个和4个亚型类群(图2)。除了3个rep基因突变质粒,两种方法分类结果在质粒家族水平完全一致,这有力地证明了分类结果和分类方法的有效性、准确性和可靠性。比较两种方法的分类结果可知,基因组共线性分类法的通用性和分辨率(亚型分类更细致)明显优于Rep蛋白进化树分类法。这些结果证明,基因组共线性分类法可以对双歧杆菌所有天然质粒进行全面、细致和可靠的系统分类,因此是双歧杆菌天然质粒一种比较理想的系统分类方法。同时,质粒基因组共线性分类法使用时需要同时进行质粒复制子序列特征分析和比较,这一定程度上增加了分析时间和操作难度,尤其对于初学者和复制机制不够清晰的质粒类型。比较而言,质粒Rep蛋白进化树分类法更加简单、快速和实用,更适合用于质粒类型的初步鉴定。总之,质粒Rep蛋白进化树和基因组共线性分类法各有优点和不足,应根据具体的研究目的、对象和范围,合理地选择一种或两种分析策略和分类方法,扬长避短,才可能获得最佳的质粒分类或鉴定结果。
随着DNA测序技术的快速发展,测序的质粒数量迅速增加,这对质粒分类和鉴定提出了更高的要求,同时也促使基于基因(组)系统进化关系的质粒系统分类成为可能。前文已经提到,早期建立的质粒不相容性表型分类法已经无法满足后基因组时代大量质粒的分类和鉴定需求。近年来,基于质粒保守序列的系统分类方法得到了较快的发展,其中复制子和松弛酶分类法最具有代表性[11,12,13]。遗传学研究显示,复制子是质粒中唯一普遍存在的遗传单元,但质粒复制子中并不存在普遍保守的基因,复制子遗传元件的组成和结构复杂且多样[13]。因此,尽管复制子是质粒系统分类中唯一具有普遍性和通用性的遗传标记,但至今质粒复制子分类法只在一些有害菌中得到了发展和应用,而在益生菌质粒分类研究中未见报道。本研究使用的Rep蛋白进化树分类法实际上是质粒复制子分类法的简化,其省去了复制子中其他序列的分析和鉴定,只保留了复制子中最常见的Rep蛋白,因此提高了分类方法的简便性和实用性。松弛酶是质粒水平转移必需的关键酶。一些研究基于松弛酶的系统发育关系对可移动质粒实现了简单、高效的系统分类,但该方法对于不可转移质粒无效,因而影响了该方法的广泛应用[13]。
另一方面,与单个保守基因或蛋白相比,理论上基于全基因组序列同源性的系统分类方法具有更好的准确性和可靠性,并且具有优良的全面性和通用性。以往研究显示,由于质粒可以跨物种频繁发生水平转移,突变和重组率高,不存在共有基因和性状,因此质粒分类一直是质粒研究领域的难点和挑战。目前质粒分类还没有一种通用、准确的方法,仍无法采用双名制命名和阶元分类系统。这直接造成了质粒分类、命名和鉴定的混乱,严重阻碍了质粒基础和应用研究的发展。利用质粒Rep蛋白进化树和基因组共线性分类法,我们前期对厚壁菌门低(G+C)mol%含量的乳杆菌属天然质粒进行了成功的系统分类[17,18,19]。本研究结果显示,这两种分类法同样适用于放线菌门高(G+C)mol%含量双歧杆菌属天然质粒的系统分类,进一步验证和拓展了这些分类方法的有效性和通用性。本研究与之前乳杆菌属质粒研究结果一致表明,质粒基因组共线性分类法比Rep蛋白进化树分类法具有更高的准确性和可靠性,并且表现出良好的全面性和通用性。因此,该方法克服了以往复制子分类法(包括Rep蛋白进化树分类法)和松弛酶分类法的不足和瓶颈,理论上适用于所有细菌天然质粒的系统分类。从这个角度而言,质粒基因组共线性分类法的建立和发展,不仅为今后乳杆菌和双歧杆菌天然质粒的系统分类鉴定提供了新的分类方法,而且为建立一种通用、准确的细菌天然质粒分类方法奠定了重要的研究基础和依据。因此,该方法可能对今后所有细菌天然质粒的系统分类、分类阶元界定、系统进化、双名制命名和分类鉴定研究具有普遍的指导意义和潜在的应用价值。同时,质粒基因组共线性分类法主要利用生物信息学软件Mauve在电脑中进行操作,实现了较高的通量(一次性分析,双歧杆菌41个质粒,乳杆菌家族5质粒116个[19])和自动化分析,基本解决了高通量测序中大量质粒分类和鉴定的难题,满足了后基因组时代天然质粒分类的高通量和自动化的需求。然而,质粒基因组共线性分类法仍存在一些不足,在使用过程中,其分析时间和分类效率仍有待于进一步优化、改进和提高。
参考文献
[1] Russell DA, Ross RP, Fitzgerald GF, Stanton C. Metabolic activities and probiotic potential of bifidobacteria[J]. International Journal of Food Microbiology, 2011, 149(1): 88-105
[2] Arboleya S, Watkins C, Stanton C, Ross RP. Gut bifidobacteria populations in human health and aging[J]. Frontiers in Microbiology, 2016, 7: 1204
[3] Ku S, Park MS, Ji GE, You HJ. Review on Bifidobacterium bifidum BGN4: functionality and nutraceutical applications as a probiotic microorganism[J]. International Journal of Molecular Sciences, 2016, 17(9): 1544
[4] Fu GF, Li X, Hou YY, Fan YR, Liu WH, Xu GX. Bifidobacterium longum as an oral delivery system of endostatin for gene therapy on solid liver cancer[J]. Cancer Gene Therapy, 2005, 12(2): 133-140
[5] Tang W, He YF, Zhou SC, Ma YP, Liu GL. A novel Bifidobacterium infantis-mediated TK/GCV suicide gene therapy system exhibits antitumor activity in a rat model of bladder cancer[J]. Journal of Experimental & Clinical Cancer Research, 2009, 28(1): 155
[6] Ruiz L, Delgado S, Ruas-Madiedo P, Sánchez B, Margolles A. Bifidobacteria and their molecular communication with the immune system[J]. Frontiers in Microbiology, 2017, 8: 2345
[7] Mahony J, Lugli GA, Van sinderen D, Ventura M. Impact of gut-associated bifidobacteria and their phages on health: two sides of the same coin?[J]. Applied Microbiology and Biotechnology, 2018, 102(5): 2091-2099
[8] Lee JH, O’Sullivan DJ. Sequence analysis of two cryptic plasmids from Bifidobacterium longum DJO10A and construction of a shuttle cloning vector[J]. Applied and Environmental Microbiology, 2006, 72(1): 527-535
[9] Orlek A, Stoesser N, Anjum MF, Doumith M, Ellington MJ, Peto T, Crook D, Woodford N, Sarah Walker A, Phan H, et al. Plasmid classification in an era of whole-genome sequencing: application in studies of antibiotic resistance epidemiology[J]. Frontiers in Microbiology, 2017, 8: 182
[10] Hedges RW, Datta N. Plasmids determining I pili constitute a compatibility complex[J]. Journal of General Microbiology, 1973, 77(1): 19-25
[11] Carattoli A, Bertini A, Villa L, Falbo V, Hopkins KL, Threlfall EJ. Identification of plasmids by PCR-based replicon typing[J]. Journal of Microbiological Methods, 2005, 63(3): 219-228
[12] Carloni E, Andreoni F, Omiccioli E, Villa L, Magnani M, Carattoli A. Comparative analysis of the standard PCR-based replicon typing (PBRT) with the commercial PBRT-KIT[J]. Plasmid, 2017, 90: 10-14
[13] Fernandez-Lopez R, Redondo S, Garcillan-Barcia MP, de la Cruz F. Towards a taxonomy of conjugative plasmids[J]. Current Opinion in Microbiology, 2017, 38: 106-113
[14] Bertini A, Poirel L, Mugnier PD, Villa L, Nordmann P, Carattoli A. Characterization and PCR-based replicon typing of resistance plasmids in Acinetobacter baumannii[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(10): 4168-4177
[15] Jensen LB, Garcia-Migura L, Valenzuela AJS, L?hr M, Hasman H, Aarestrup FM. A classification system for plasmids from enterococci and other Gram-positive bacteria[J]. Journal of Microbiological Methods, 2010, 80(1): 25-43
[16] Lozano C, García-Migura L, Aspiroz C, Zarazaga M, Torres C, Aarestrup FM. Expansion of a plasmid classification system for Gram-positive bacteria and determination of the diversity of plasmids in Staphylococcus aureus strains of human, animal, and food origins[J]. Applied and Environmental Microbiology, 2012, 78(16): 5948-5955
[17] Sun DQ, Li HF, Song DW, Yang J. Classification of natural plasmids in Lactobacillus plantarum[J]. Food Science, 2017, 38(12): 69-74 (in Chinese)
[18] Sun DQ, Li HF, Yang J, Song DW. Classification of natural plasmids in Lactobacillus brevis[J]. Food Science, 2018, 39(10): 173-178 (in Chinese)
[19] Sun DQ, Song DW, Li HF. Research on the Genomics of Natural Plasmids of Lactobacillus[M]. Beijing: China Light Industry Press, 2018: 187-189 (in Chinese)
[20] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874
[21] Darling ACE, Mau B, Blattner FR, Perna NT. Mauve: multiple alignment of conserved genomic sequence with rearrangements[J]. Genome Research, 2004, 14(7): 1394-1403
[22] Seery LT, Nolan NC, Sharp PM, Devine KM. Comparative analysis of the pC194 group of rolling circle plasmids[J]. Plasmid, 1993, 30(3): 185-196
[23] O'Riordan K, Fitzgerald GF. Molecular characterisation of a 5.75-kb cryptic plasmid from Bifidobacterium breve NCFB 2258 and determination of mode of replication[J]. FEMS Microbiology Letters, 1999, 174(2): 285-294
[24] Gibbs MJ, Smeianov VV, Steele JL, Upcroft P, Efimov BA. Two families of Rep-like genes that probably originated by interspecies recombination are represented in viral, plasmid, bacterial, and parasitic protozoan genomes[J]. Molecular Biology and Evolution, 2006, 23(6): 1097-1100
[25] Benachour A, Frère J, Novel G. pUCL287 plasmid from Tetragenococcus halophila (Pediococcus halophilus) ATCC 33315 represents a new theta-type replicon family of lactic acid bacteria[J]. FEMS Microbiology Letters, 1995, 128(2): 167-175
[26] Guglielmetti S, Karp M, Mora D, Tamagnini I, Parini C. Molecular characterization of Bifidobacterium longum biovar longum NAL8 plasmids and construction of a novel replicon screening system[J]. Applied Microbiology and Biotechnology, 2007, 74(5): 1053-1061
[27] Corneau N, ?mond ?, LaPointe G. Molecular characterization of three plasmids from Bifidobacterium longum[J]. Plasmid, 2004, 51(2): 87-100
[28] Park MS, Lee KH, Ji GE. Isolation and characterization of two plasmids from Bifidobacterium longum[J]. Letters in Applied Microbiology, 1997, 25(1): 5-7
[29] Park MS, Moon HW, Ji GE. Molecular characterization of plasmid from Bifidobacterium longum[J]. Journal of Microbiology and Biotechnology, 2003, 13(3): 457-462
[30] ?lvarez-Martín P, ?ycka-Krzesińska J, Bardowski J, Mayo B. Sequence analysis of plasmid pSP02 from Bifidobacterium longum M62 and construction of pSP02-derived cloning vectors[J]. Plasmid, 2013, 69(2): 119-126
[31] Park MS, Shin DW, Lee KH, Ji GE. Sequence analysis of plasmid pKJ50 from Bifidobacterium longum[J]. Microbiology, 1999, 145(3): 585-592
[32] Ruiz-Masó JA, Machón C, Bordanaba-Ruiseco L, Espinosa M, Coll M, del Solar G. Plasmid rolling-circle replication[J]. Microbiology Spectrum, 2015, 3(1): PLAS-0035-2014
[33] Ma YP, Xie TT, Hu QW, Qiu ZY, Song FZ. Sequencing analysis and characterization of the plasmid pBIF10 isolated from Bifidobacterium longum[J]. Canadian Journal of Microbiology, 2015, 61(2): 124-130
[34] Aoki K, Shinohara M, Itoh T. Distinct functions of the two specificity determinants in replication initiation of plasmids ColE2-P9 and ColE3-CA38[J]. Journal of Bacteriology, 2007, 189(6): 2392-2400
[35] Moon GS, Wegmann U, Gunning AP, Gasson MJ, Narbad A. Isolation and characterization of a theta-type cryptic plasmid from Bifidobacterium longum FI10564[J]. Journal of Microbiology and Biotechnology, 2009, 19(4): 403-408
[36] Takahata M, Toh H, Nakano A, Takagi M, Murakami M, Ishii Y, Takizawa T, Tanabe S, Morita H. Complete sequence analysis of two cryptic plasmids from Bifidobacterium kashiwanohense JCM 15439 (type strain) isolated from healthy infant feces[J]. Animal Science Journal, 2014, 85(2): 158-163
[37] Cronin M, Knobel M, O'Connell-Motherway M, Fitzgerald GF, van Sinderen D. Molecular dissection of a bifidobacterial replicon[J]. Applied and Environmental Microbiology, 2007, 73(24): 7858-7866