摘 要: 寨卡病毒(ZIKV)是黄病毒科黄病毒属的一种正链RNA病毒,可引起新生儿小头症和其他神经系统相关疾病。ZIKV反向遗传学技术是从ZIKV互补DNA(cDNA)获得感染性病毒的技术,该技术可在DNA水平上对病毒的基因组进行改造,为研究ZIKV提供了重要的技术平台。本文就ZIKV反向遗传学系统的构建方法及其在ZIKV致病机制、疫苗研发、药物筛选、肿瘤治疗等研究中的应用进行综述。
关键词: 寨卡病毒; 反向遗传学; 感染性克隆;
Abstract: Zika virus(ZIKV)is a positive-strand RNA virus belonging to the genus Flavivirus(the family Flaviviridae),which can cause congenital microcephaly and other severe neurological diseases. The ZIKV reverse genetics is a technology to obtain infective virus from complementary DNA(cDNA)of ZIKV. This technology modifies virus genome at the DNA level,providing an important technical platform for ZIKV research. This article reviews the construction strategies of ZIKV reverse genetics technique and its application in the research of ZIKV pathogenic mechanism,vaccine development,drug screening and tumor treatment.
Keyword: Zika virus; reverse genetics; infective clone;
寨卡病毒(Zika virus,ZIKV)是一种人类致病性蚊媒黄病毒。1947年,科学家首次从乌干达寨卡森林的恒河猴体内分离出该病毒[1]。自发现以来,ZIKV仅在非洲、亚洲及南太平洋岛屿有小规模暴发,并未引起人们注意。2015年,ZIKV在南美暴发,造成数百万人感染[2]。自此,ZIKV才引起世界范围的广泛关注。大部分ZIKV感染者无临床症状,仅约20%感染者表现出低热、斑丘疹、肌痛等轻微的临床症状[3],少数出现严重的神经和免疫系统并发症[4]。孕妇感染ZIKV可导致部分胎儿出现小头症[5,6],ZIKV感染还与格林-巴利综合征(GuillianBarre syndrome,GBS)的发生有关[7,8]。ZIKV可通过蚊叮咬进行传播,伊蚊属是其主要媒介载体[9]。此外,ZIKV还可通过垂直传播、性传播和输血传播这些非媒介方式进行传播[10,11,12,13]。
ZIKV属于黄病毒科黄病毒属,是一种包膜病毒[14],其基因组为单股正链RNA,长约11kb,5'和3'端两侧为非编码区(untranslated region,UTR)。病毒编码区在宿主细胞的胞质中翻译成一条多肽链,进而被宿主或病毒自身的蛋白酶加工成3个结构蛋白(C、pr M/M、E)和7个非结构蛋白(NS1、NS2A、NS2B、NS3、NS4A、NS4B、NS5)[15]。结构蛋白与病毒核酸等构成病毒粒子,非结构蛋白在病毒基因组复制、多肽加工、拮抗宿主免疫反应等方面起重要作用[16]。
反向遗传学技术从遗传物质着手研究生物的表型变化,该技术在病毒分子生物学研究中的应用,称为病毒反向遗传学技术(reverse genetics of virus),又称“病毒拯救”(the rescue of virus)。ZIKV是正链RNA病毒,反向遗传学技术最初就是在正链RNA病毒的研究中得到应用的[17]。正链RNA病毒本身可作为m RNA翻译出病毒蛋白,因此转染ZIKV互补DNA(complementary DNA,c DNA)的RNA体外转录产物或直接转染ZIKV c DNA于易感细胞,均可组装出感染性ZIKV。在病毒产生的环节中,c DNA的存在使操作病毒基因组成为可能,通过在病毒c DNA中引入突变对病毒进行改造,可对病毒基因组结构、基因产物及基因功能进行研究。探寻ZIKV暴发的原因、解析其感染及致病机制、构建ZIKV疫苗等均离不开病毒反向遗传学技术。本文就ZIKV反向遗传学技术、面临的问题及解决办法、病毒拯救的方法及其应用进行了综述,为ZIKV相关研究提供参考。

1 、ZIKV反向遗传系统的建立
建立ZIKV反向遗传学系统主要包括构建ZIKV全长c DNA克隆、体外转录出感染性RNA、转染感染性RNA或感染性c DNA于易感细胞和病毒鉴定[18]4个步骤(图1)。
1.1、 ZIKV c DNA克隆的构建
构建全长c DNA克隆是反向遗传学技术的第一步,也是最重要的一步。提取ZIKV基因组RNA后,使用逆转录方法获得其c DNA。在ZIKV c DNA的5'端添加启动子序列,用于转录出病毒RNA;在其3'端添加丁型肝炎核酶序列(HDVr),通过核酶自剪接去除转录出的非病毒基因组序列。以这种“启动子-ZIKV c DNA-HDVr核酶”结构为模板就能在体外或体内制备出感染性ZIKV。ZIKV全长c DNA的特殊之处在于其在细菌内的不稳定性,这种不稳定是基因组pr M-E-NS1片段中有毒的原核启动子造成的[19,20,21]。如何克服这个问题,获得稳定的“启动子-ZIKV c DNA-HDVr核酶”结构是构建ZIKV全长c DNA面临的主要问题,研究人员采取了多种方法构建“启动子-ZIKV c DNA-HDVr核酶”结构,现分述如下。
1.1.1、 使用低拷贝质粒载体或细菌人工染色体构建ZIKV全长c DNA克隆,增加其稳定性
含有ZIKV pr M-E-NS1基因片段的高拷贝质粒载体不稳定,复制过程中对宿主菌的毒性可导致目的基因的异常缺失或突变[22]。使用低拷贝质粒载体,可维持隐性启动子在较低的表达水平,从而提高ZIKV c DNA在载体中的稳定性。Annamalai等[23]使用低拷贝质粒p BR322,构建出了序列正确的ZIKV全长c DNA克隆。大多数质粒载体理论上可容纳<10 kb的片段,目的基因片段过长可能限制质粒复制,影响其稳定性,导致目的基因碱基丢失或突变[24]。细菌人工染色体(bacterial artificial chromosome,BAC)能容纳高达350kb的外源基因,多用于大片段c DNA文库的构建,可代替质粒用作感染性克隆的载体[25]。Márquez-Jurado等[26]将ZIKV毒株RGN的基因克隆到BAC中,成功构建了含有ZIKV全长c DNA的克隆p BAC-ZIKV-RGN。此外,?vila-Pérez等[27]将ZIKV基因组克隆到人工染色体p Belo BAC11中,组装出感染性c DNA克隆。BAC常以环状存在于细菌体内,拷贝数低,转化效率高,易于分离纯化,在一定程度上可降低ZIKV感染性克隆对宿主的毒性并提高其稳定性。
1.1.2、 在pr M-E-NS1编码区插入内含子或引入突变,消除有毒的原核启动子
ZIKV基因组中E基因和NS1基因编码序列中存在有毒的原核启动子,导致其难以在宿主中稳定地传代复制[28]。真核生物的内含子序列通常富含终止密码子,在病毒基因组编码区中插入短的真核内含子序列可有效终止病毒c DNA毒性肽的翻译,从而提高感染性c DNA克隆的稳定性。Liu等将真核生物的内含子插入ZIKV基因E和基因NS1区域内,获得了含有内含子的ZIKV c DNA克隆,并在转染细胞的上清液中检测到感染性ZIKV。此外,细菌启动子的活性可能影响病毒c DNA在细菌中的复制,通过在启动子序列中引入点突变,可降低细菌启动子的活性,从而提高病毒c DNA稳定性。Chen等通过引入同义突变降低细菌启动子的活性,构建出能产生具有遗传稳定性的ZIKV毒株ZG01的全长c DNA克隆。插入真核内含子或引入点突变虽降低了病毒c DNA克隆对宿主菌的毒性并提高了其稳定性,但其对病毒基因组进行的改造可能会对病毒基因结构与功能关系的研究造成干扰。
图1 寨卡病毒(ZIKV)反向遗传系统的建立示意图
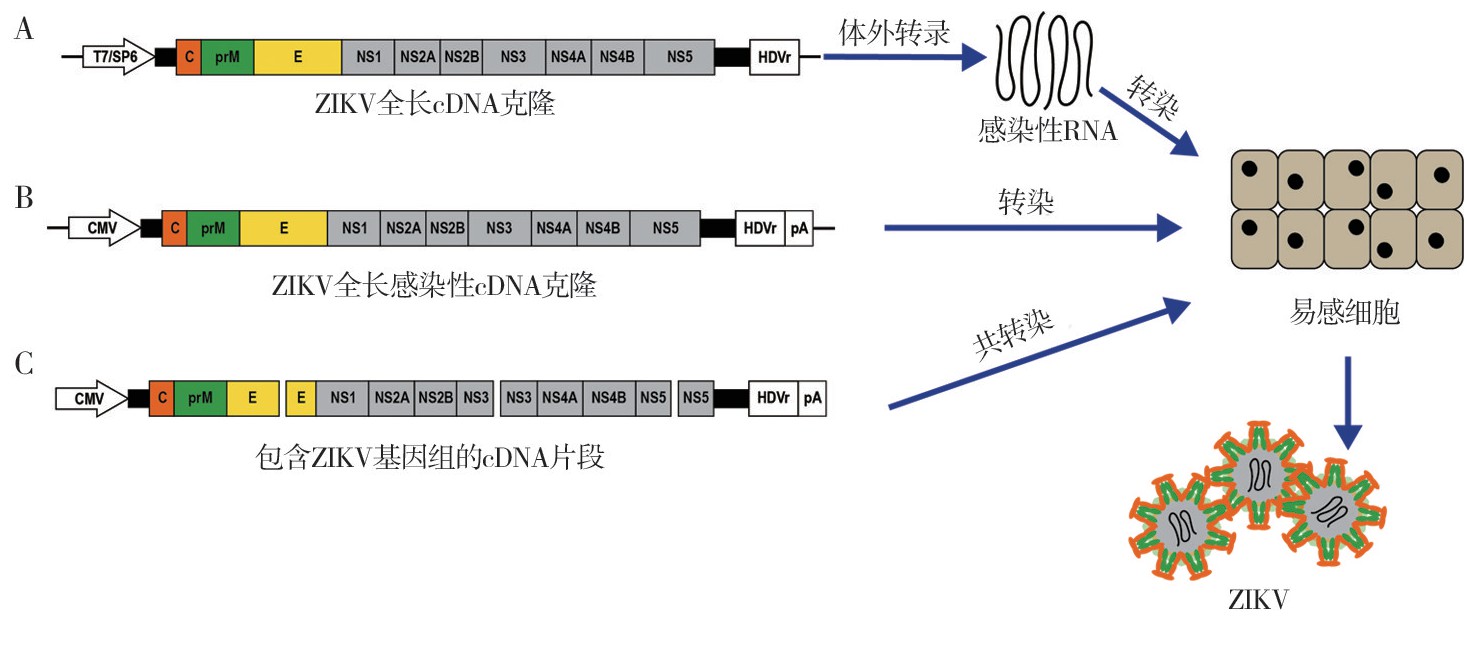
注:A.转染ZIKV全长cDNA克隆的RNA转录产物于易感细胞;B.转染ZIKV全长感染性cDNA克隆于易感细胞;C.共转染包含ZIKV基因组的cDNA片段于易感细胞
1.1.3、 体外连接构建ZIKV全长c DNA,避开细菌内的扩增步骤
获得包含ZIKV全长的各个亚克隆或c DNA片段以后,将病毒基因组片段在体外定向连接成全长ZIKV c DNA,以此c DNA为模板,在体外转录出病毒RNA。Deng等[33]构建了覆盖完整ZIKV基因组的4个c DNA亚克隆,利用体外连接方法组装出ZIKV的全长c DNA克隆。从全长c DNA转录的RNA转染易感细胞后获得了感染性ZIKV。2017年,Weger-Lucarelli等[34]将ZIKV株PRVABC59的病毒基因组从不稳定的NS1基因区域分隔开,分两段克隆到质粒载体p ACYC177中。体外连接2个c DNA亚克隆,将连接产物转录出的RNA转染Vero细胞,成功拯救出了感染性ZIKV。环状聚合酶延伸克隆(circular polymerase extension cloning,CPEC)基于聚合酶延伸机制将多个c DNA片段按顺序组装,体外组装成全长感染性ZIKV c DNA克隆。2017年,Marquez-Jurado等使用CPEC方法构建了巴西ZIKV株RGN的全长感染性c DNA克隆。该方法反应产物为环状,可直接转染Vero细胞[35]。体外连接方法构建全长c DNA克隆,可避免使用宿主菌扩增全长c DNA,也就避免了ZIKV c DNA在复制过程中对宿主的毒性及其不稳定性。体外连接的缺点是病毒基因组或c DNA片段中需要含有数量和位置合适的限制性酶切位点,连接效率低。与使用宿主菌扩增相比,难以获得大量的全长c DNA克隆,需要高效的转录系统支持[36,37]。
1.2 、转染易感细胞
转染易感细胞有2种方法:先在体外通过转录获得病毒RNA,然后将RNA转染易感细胞,或直接转染病毒c DNA于易感细胞[18]。常用的转染方法主要有脂质体转染法和电穿孔法。脂质体转染法操作方便,转染率高。但脂质体对细胞有一定的毒性,故转染时间一般不超过24 h[38]。电穿孔法转染周期更短,无潜在细胞毒性,因此使用范围更广。同时,使用电转保护剂,可减少电场对细胞的杀伤作用,并提高转染效率[39]。转染后根据细胞的贴壁情况、形态变化,适时更换新鲜的细胞培养基,以减少脂质体或其他转染试剂对细胞的毒害作用。一般情况下,培养3~4 d后可收集细胞培养上清液进行鉴定或者进一步扩大培养。
1.2.1 、转染全长c DNA克隆的RNA转录产物
在病毒c DNA的5'端添加噬菌体来源的T7或SP6启动子序列,3'端添加的丁型肝炎核酶(hepatitis delta virus ribozyme,HDVr)序列,体外转录出病毒RNA后转染易感细胞(图1A)。在细胞内,通过核酶自剪接去除体外转录产物中的非病毒序列。这样加工后的病毒RNA即可作为模板,一方面翻译出病毒增殖所需的蛋白,另一方面复制病毒基因组,最终在细胞内组装出感染性病毒[40,41]。
1.2.2、 直接转染病毒c DNA克隆
与原核RNA聚合酶T7或SP6启动子驱动的转录拯救系统相比,在病毒c DNA的5'端添加人巨细胞病毒(cytomegalovirus,CMV)启动子序列,3'端添加HDVr序列和RNA聚合酶Ⅱ终止子多聚腺苷酸化信号(polyadenylation signal,p A)序列构成真核RNA聚合酶Ⅱ转录系统,构建出的感染性全长c DNA克隆,可直接转染易感细胞或易感动物,在细胞内RNA聚合酶Ⅱ作用下驱动CMV启动子进行RNA转录及蛋白翻译,获得感染性病毒(图1B),该方法不需要体外转录病毒RNA[42]。有些ZIKV株难以获得稳定的全长c DNA克隆,可将包含整个病毒基因组的多个c DNA片段共转染于易感细胞(图1C),需在第一个c DNA片段的5'端添加CMV启动子,最后一个c DNA片段3'端添加HDVr和p A[43]。这些片段在细胞内经同源重组组装成感染性全长c DNA,从而进一步获得感染性病毒。Atieh等[44]使用该方法,分别基于ZIKV亚洲毒株PF和非洲毒株DAK构建了2个ZIKV反向遗传学系统。该方法不依赖宿主菌,可在短时间内获得野生型和含有不同标记的ZIKV嵌合体,常用于正链RNA病毒的拯救[45]。
1.3、 拯救病毒的鉴定
通过对反向遗传学技术获得的ZIKV的序列、复制能力、毒力变化及遗传稳定性等多方面进行分析,以评价反向遗传学系统的可靠性。具体包括:(1)使用限制性内切酶酶切感染性克隆质粒与理论值比较;(2)观察和比较拯救病毒与野生型病毒产生噬斑的大小及形态差异;(3)通过引入的报告基因或标记基因评价病毒在细胞内的增殖能力;(4)通过动物模型评价病毒在体内的毒力变化;(5)通过测序及生物信息学方法分析病毒的遗传稳定性等[46,47]。
2 、反向遗传学技术在ZIKV研究中的应用
通过使用反向遗传学技术,可在ZIKV基因组中引入突变,用于ZIKV致病机制的研究,也可用于抗ZIKV药物筛选平台、ZIKV疫苗和溶瘤病毒等构建。
2.1、 用于ZIKV复制机制研究
Schwarz等[30]通过在ZIKV c DNA的NS1基因区域插入合成内含子,降低了感染性c DNA克隆在细菌中的毒性,构建出ZIKV毒株MR766的感染性c DNA克隆;转染破坏了非结构蛋白NS5聚合酶活性位点的感染性c DNA克隆,无法获得感染性ZIKV。显示非结构蛋白NS1与病毒毒力相关,非结构蛋白NS5聚合酶活性位点与病毒复制相关。Márquez-Jurado等[26]从BAC感染性克隆中拯救出在体内和体外均具有完全感染性的ZIKV,发现感染性克隆的非结构蛋白NS2A中第175位氨基酸残基的突变(丙氨酸变为缬氨酸)影响了病毒RNA的合成,导致病毒毒性降低,显示非结构蛋白NS2A可能与病毒复制和毒力水平相关。此外,Yang等[48]发现对于拯救的ZIKV株FSS13025,在非结构蛋白NS1第265位引入氨基酸残基突变(赖氨酸变为谷氨酸)增强了ZIKV在Vero细胞内的复制。将该突变引入另一ZIKV毒株PRVABC5发现,NS1中该氨基酸残基突变的增强病毒复制效应与ZIKV毒株无关,该位点的这种突变普遍可增强ZIKV的复制能力。Yuan等[47]基于野生型ZIKV毒株CAM/2010构建了7个氨基酸突变感染性克隆,发现与野生型毒株相比,S139N突变体有更强的复制能力和神经毒力,经动物模型实验和生物信息学溯源分析,显示该突变与新生儿小头症密切相关。
2.2 、用于构建ZIKV疫苗
除了预防蚊叮咬外,接种疫苗可能是预防ZIKV感染最经济有效的手段,但目前尚无获批的预防ZIKV感染的疫苗[49,50]。建立ZIKV反向遗传学技术操作平台可作为高效的疫苗技术储备,对研究ZIKV和研发ZIKV疫苗有重要意义。Yang等[48]在ZIKV毒株PRVABC59的感染性c DNA克隆上引入了3个氨基酸残基突变,构建了一个能高效生产寨卡灭活疫苗的c DNA克隆平台,生产的纯化灭活疫苗在动物实验中具有较好的免疫效果,目前已进入临床试验。Shan等[51]在ZIKV感染性克隆的基础上,通过删除基因组3'-UTR中的10个核苷酸构建了一种寨卡减毒疫苗(10-del ZIKV),该疫苗具有很好的免疫原性和保护性。Li等[52]将ZIKV pr M/E基因克隆到乙脑病毒疫苗株SA-14-14-2感染性克隆上,构建出乙脑/寨卡嵌合病毒感染性克隆,成功拯救出对小鼠有较强神经毒力的乙脑/寨卡嵌合病毒,为研发ZIKV嵌合疫苗奠定了基础。
2.3 、用于建立抗病毒药物筛选平台
建立抗ZIKV药物筛选系统可为预防和治疗ZIKV感染提供支持[53]。利用反向遗传学技术构建的含有报告基因或标记基因的ZIKV复制子系统可用于抗ZIKV药物的筛选[54,55]。Li等[56]使用反向遗传学技术构建了一个包含海肾荧光素酶(Rluc)报告基因和嘌罗霉素N-乙酰基转移酶(Pac)选择标记基因的双顺反子ZIKV复制子(ZIKV-Pac-Rluc-Rep),通过嘌罗霉素的选择,建立了稳定表达高水平ZIKV复制子的克隆细胞系,开发出基于ZIKV复制子的高通量筛选(high throughput screening,HTS)分析技术,用于筛选抗ZIKV药物。刘娜等[46]使用ZIKV的感染性c DNA克隆,将Rluc插入ZIKV基因组中,构建了能表达荧光素酶的ZIKV复制子Rluc-ZIKV-Rep,通过检测荧光信号的变化反映药物对病毒增殖能力的影响,可用于抗ZIKV药物的筛选。
2.4 、用于构建溶瘤病毒
相比西尼罗病毒不加区别地感染杀伤肿瘤细胞和正常神经细胞[57],ZIKV可跨过血脑屏障,特异感染和杀伤神经前体细胞和神经干细胞[58]。ZIKV对成胶质细胞瘤干细胞(glioblastoma stem cells,GSC)有特异性杀伤能力,而对成人的正常神经细胞无明显毒性。为尽可能在降低ZIKV毒力的同时,保留其溶瘤能力[59],Chen等[60]通过反向遗传学技术,对病毒基因组进行靶向改造,得到了减毒的寨卡毒株ZIKV-LAV,在小鼠神经胶质瘤模型中,可特异性杀死肿瘤内的GSC,显着抑制了小鼠脑内肿瘤的生长,延长了小鼠的存活时间,同时具有很好的安全性。改造后的ZIKV作为治疗人成胶质细胞瘤的新型病毒疗法候选药物,在脑部肿瘤治疗方面,有重要的临床应用价值。
3 、结语
2020年6月,研究人员发现一种新型非洲系ZIKV毒株正在巴西传播,这很可能会引发另一场寨卡疫情。若要阻止病毒的进一步蔓延,并对ZIKV感染者进行有效治疗,很多关于ZIKV致病机制的问题亟待解决。反向遗传学技术为研究ZIKV的致病机制提供了新的思路和方法,为ZIKV疫苗和脑部肿瘤治疗药物的研发提供了技术平台。然而,对于ZIKV这类基因组较大且含有毒性启动子的病毒,现有构建c DNA克隆的方法都有其局限性。随着反向遗传学技术不断发展,研究人员可综合考虑多种方法,扬长避短,优化已有的方法,探索更多新型方法用于ZIKV或其他病毒的拯救。此外,多种新型寨卡嵌合病毒的出现,使得我们不得不关注它们潜在的生物安全风险。科研工作者应合理利用反向遗传学技术,在安全可控的范围内进一步推动相关研究。
参考文献
[1]Dick GW,Kitchen SF,Haddow AJ.Zika virus.I.isolations and serological specificity[J].Trans R Soc Trop Med Hyg,1952,46(5):509-520.
[2]Faria NR,Quick J,Claro IM,et al.Establishment and cryptic transmission of Zika virus in Brazil and the Americas[J].Nature,2017,546(7658):406-410.
[3] Chang C,Ortiz K,Ansari A,et al.The Zika outbreak of the21st century[J].J Autoimmun,2016,68:1-13.
[4] Fabienne K,Maurane R,Ludovic R,et al.Zika virus infection as a cause of congenital brain abnormalities and guillain-barrésyndrome:systematic review[J].PLoS Med,2017,14(1).DOI:10.1371/journal.pmed.1002203.
[5]Mlakar J,Korva M,Tul N,et al.Zika virus associated with microcephaly[J].N Engl J Med,2016,374(10):951-958.
[6]Miner JJ,Cao B,Govero J,et al.Zika virus infection during pregnancy in mice causes placental damage and ftal demise[J].Cell,2016,165(5):1081-1091.
[7]Lynch RM,Mantus G,Encinales L,et al.Augmented Zika and Dengue neutralizing antibodies are associated with guillainbarre syndrome[J].J Infect Dis,2019,219(1):26-30.
[8]Mécharles S,Herrmann C,Poullain P,et al.Acute myelitis due to Zika virus infection[J].Lancet,2016,387(10026):1481.
[9] Shan C,Xie X,Ren P,et al.A rapid Zika diagnostic assay to measure neutralizing antibodies in patients[J].E Bio Medicine,2017,17:157-162.
[10]Yockey LJ,Varela L,Rakib T,et al.Vaginal exposure to Zika virus during pregnancy leads to fetal brain infection[J].Cell,2016,166(5):1247-1256.e4.
[11]D'Ortenzio E,Matheron S,Yazdanpanah Y,et al.Evidence of sexual transmission of Zika virus[J].N Engl J Med,2016,374(22):2195-2198.
[12]Motta IJ,Spencer BR,Cordeiro da Silva SG,et al.Evidence for transmission of Zika virus by platelet transfusion[J].N Engl JMed,2016,375(11):1101-1103.
[13]Simpson DI.Zika virus infection in man[J].Trans R Soc Trop Med Hyg,1964,58:335-338.
[14]Musso D,Gubler DJ.Zika Virus[J].Clin Microbiol Rev,2016,29(3):487-524.
[15]Baronti C,Piorkowski G,Charrel RN,et al.Complete coding sequence of zika virus from a French polynesia outbreak in 2013[J].Genome Announc,2014,2(3):1-2.
[16]Hasan SS,Sevvana M,Kuhn RJ,et al.Structural biology of Zika virus and other flaviviruses[J].Nat Struct Mol Biol,2018,25(1):13-20.
[17]Taniguchi T,Palmieri M,Weissmann C.QB DNA-containing hybrid plasmids giving rise to QB phage formation in the bacterial host[J].Nature,1978,274(5668):223-228.
[18]?vila-Pérez G,Nogales A,Martín V,et al.Reverse genetic approaches for the generation of recombinant Zika virus[J].Viruses,2018,10(11):597.
[19]Gadea G,Bos S,Krejbich-Trotot P,et al.A robust method for the rapid generation of recombinant Zika virus expressing the GFP reporter gene[J].Virology,2016,497:157-162.
[20]Atieh T,Baronti C,de Lamballerie X,et al.Simple reverse genetics systems for Asian and African Zika viruses[J].Sci Rep,2016,6:39384.
[21] Bos S,Viranaicken W,Turpin J,et al.The structural proteins of epidemic and historical strains of Zika virus differ in their ability to initiate viral infection in human host cells[J].Virology,2018,516:265-273.
[22]Shi PY,Tilgner M,Lo MK,et al.Infectious cDNA clone of the epidemic west nile virus from New York City[J].J Virol,2002,76(12):5847-5856.
[23] Annamalai AS,Pattnaik A,Sahoo BR,et al.Zika virus encoding nonglycosylated envelope protein is attenuated and defective in neuroinvasion[J].J Virol,2017,91(23).DOI:10.1128/JVI.01348-17.
[24] 房恩岳,王玲,李玉华.登革病毒反向遗传学技术的研究进展[J].中国生物制品学杂志,2020,33(3):328-333.
[25]Monaco AP,Larin Z.YACs,BACs,PACs and MACs:artificial chromosomes as research tools[J].Trends Biotechnol,1994,12(7):280-286
[26]Márquez-Jurado S,Nogales A,?vila-Pérez G,et al.An alanineto-valine substitution in the residue 175 of Zika virus NS2A protein affects viral RNA synthesis and attenuates the virus in vivo[J].Viruses,2018,10(10):547.
[27]?vila-Pérez G,Park J-G,Nogales A,et al.Rescue of recombinant Zika virus from a bacterial artificial chromosome cDNAclone[J].J Vis Exp,2019,6(148).DOI:10.3791/59537.
[28]Pu SY,Wu RH,Yang CC,et al.Successful propagation of flavivirus infectious cDNAs by a novel method to reduce the cryptic bacterial promoter activity of virus genomes[J].J Virol,2011,85(6):2927-2941.
[29]Tsetsarkin KA,Kenney H,Chen R,et al.A full-length infectious cDNA clone of Zika virus from the 2015 Epidemic in Brazil as a genetic platform for studies of virus-host interactions and vaccine development[J].mBio,2016,7(4).DOI:10.1128/mBio.01114-16.
[30]Schwarz MC,Sourisseau M,Espino MM,et al.Rescue of the1947 Zika virus prototype strain with a cytomegalovirus promoterdriven cDNA clone[J].mSphere,2016,1(5).DOI:10.1128/mSphere.00246-16.
[31] Liu ZY,Yu JY,Huang XY,et al.Characterization of cis-acting RNA elements of Zika virus by using a self-splicing ribozyme-dependent infectious clone[J].J Virol,2017,91(21):doi:10.1128/JVI.00484-17.
[32] Chen Y,Liu T,Zhang Z,et al.Novel genetically stable infectious clone for a Zika virus clinical isolate and identification of RNA elements essential for virus production[J].Virus Res,2018,257:14-24.
[33]Deng CL,Zhang QY,Chen DD,et al.Recovery of the Zika virus through an in vitro ligation approach[J].J Gen Virol,2017,98(7):1739-1743.
[34]Weger-Lucarelli J,Duggal NK,Brault AC,et al.Rescue and characterization of recombinant virus from a new world Zika virus infectious clone[J].J Vis Exp,2017(124).DOI:10.3791/55857
[35]Quan J,Tian J.Circular polymerase extension cloning of complex gene libraries and pathways[J].PLoS One,2009,4(7):DOI:10.1371/journal.pone.0006441.
[36] Gibson DG.Enzymatic assembly of overlapping DNA fragments[J].Methods Enzymol,2011,498:349-361.
[37]Gibson DG,Young L,Chuang RY,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases[J].Nat Methods,2009,6(5):343-345.
[38]Kumar P,Nagarajan A,Uchil PD.DNA Transfection mediated by cationic lipid reagents[J].Cold Spring Harb Protoc,2019,2019(3):215-224.
[39]Shi J,Ma Y,Zhu J,et al.A Review on electroporation-based intracellular delivery[J].Molecules,2018,23(11):3044.
[40]Widman DG,Young E,Yount BL,et al.A reverse genetics platform that spans the Zika virus family tree[J].mBio,2017,8(2).DOI:10.1128/mBio.02014-16.
[41] Aubry F,Nougairède A,Gould EA,et al.Flavivirus reverse genetic systems,construction techniques and applications:a historical perspective[J].Antiviral Res,2015,114:67-85.
[42]Kümmerer BM.Establishment and application of flavivirus replicons[J].Adv Exp Med Biol,2018,1062:165-173.
[43] Usme-Ciro JA,Lopera JA,Enjuanes L,et al.Development of a novel DNA-launched dengue virus type 2 infectious clone assembled in a bacterial artificial chromosome[J].Virus Res,2014,180:12-22.
[44]Atieh T,Nougairède A,Klitting R,et al.New reverse genetics and transfection methods to rescue arboviruses in mosquito cells[J].Sci Rep,2017,7(1):13983.
[45]Atieh T,El Ayoubi MD,Aubry F,et al.Haiku:New paradigm for the reverse genetics of emerging RNA viruses[J].Plos One,2018,13(2).DOI:10.1371/journal.pone.0193069.
[46] 刘娜,胡媛媛,陈凯丽,等.基于寨卡病毒复制子的抗病毒药物筛选系统(英文)[J].生命科学研究,2020,24(2):95-101,117.
[47] Yuan L,Huang XY,Liu ZY,et al.A single mutation in the prM protein of Zika virus contributes to fetal microcephaly[J].Science,2017,358(6365):933-936.
[48] Yang Y,Shan C,Zou J,et al.A cDNA clone-launched platform for high-yield production of inactivated Zika vaccine[J].E BioMedicine,2017,17:145-156.
[49]Qu P,Zhang C,Li M,et al.A new class of broadly neutralizing antibodies that target the glycan loop of Zika virus envelope protein[J].Cell Discov,2020,6(5):1-14.
[50]Sharma V,Sharma M,Dhull D,et al.Zika virus:an emerging challenge to public health worldwide[J].Can J Microbiol,2020,66(2):87-98.
[51]Shan C,Muruato AE,Nunes BTD,et al.A live-attenuated Zika virus vaccine candidate induces sterilizing immunity in mouse models[J].Nat Med,2017,23(6):763-767.
[52]Li XF,Dong HL,Wang HJ,et al.Development of a chimeric Zika vaccine using a licensed live-attenuated flavivirus vaccine as backbone[J].Nat Commun,2018,9(1):673.
[53]Mutso M,Saul S,Rausalu K,et al.Reverse genetic system,genetically stable reporter viruses and packaged subgenomic replicon based on a Brazilian Zika virus isolate[J].J Gen Virol,2017,98(11):2712-2724.
[54]Fernandes RS,Freire MCLC,Bueno RV,et al.Reporter replicons for antiviral drug discovery against positive single-stranded RNA viruses[J].Viruses,2020,12(6):598.
[55] Xie X,Zou J,Shan C,et al.Zika virus replicons for drug discovery[J].E Bio Medicine,2016,12:156-160.
[56] Li JQ,Deng CL,Gu D,et al.Development of a replicon cell line-based high throughput antiviral assay for screening inhibitors of Zika virus[J].Antiviral Res,2018,150:148-154.
[57]Moore AE.Effects of viruses on tumors[J].Annu Rev Microbiol,1954,8:393-410.
[58]Noronha L,Zanluca C,Azevedo ML,et al.Zika virus damages the human placental barrier and presents marked fetal neurotropism[J].Mem Inst Oswaldo Cruz,2016,111(5):287-293.
[59] Zhu Z,Gorman MJ,McKenzie LD,et al.Zika virus has oncolytic activity against glioblastoma stem cells[J].J Exp Med,2017,214(10):2843-2857.
[60]Chen Q,Wu J,Ye Q,et al.Treatment of human glioblastoma with a live attenuated Zika virus vaccine candidate[J].mBio,2018,9(5).DOI:10.1128/mBio.01683-18.
麻疹病毒属隶属于副黏病毒科副黏病毒亚科,共6个病毒成员,包括犬瘟热病毒(Caninedistem-pervirus,CDV)、麻疹病毒(Morbillivirus)、海豹瘟病毒(Phocinedistempervirus,PDV)、牛瘟病毒(Rinderpestvirus,RPV)、鲸类动物麻疹病毒(Ceta-ceanmorbi...