摘 要: TET3(ten-eleven translocation 3)属于TET家族成员,是一种依赖于Fe2+和α-酮戊二酸(α-ketoglutarate,α-KG)的双加氧酶,可以把5-甲基胞嘧啶(5-m C)氧化为5-羟甲基胞嘧啶(5-hm C),介导DNA的去甲基化,以完成对基因表达的调控。研究显示TET3不仅在神经分化中上调表达,而且在配子发育中持续表达,并对早期胚胎的发育激活起到重要作用,揭示TET3介导的基因表达调控对哺乳动物的生殖发育具有一定影响。现对TET3的蛋白结构、在配子和胚胎发育中的调控作用以及TET3相关的生物学功能等进行综述,以促进TET3的功能研究及其在生命科学领域中的应用。
关键词: TET3; 5-甲基胞嘧啶; DNA甲基化; 配子发生; 胚胎发育;
Abstract: Ten-eleven translocation 3(TET3), one member of TET family, is a dioxygenase that depends on Fe2+and α-ketoglutarate(α-KG). TET3 can oxidize 5-methylcytosine(5-mC) to 5-hydroxymethylcytosine(5-hmC) and complete the regulation of gene expression. Studies showed that the expression of TET3 was not only up-regulated during neural differentiation, but also sustained during gametogenesis, and that TET3 played an important role in regulating the early embryonic development and activation. In other words, the TET3-mediated gene expression and regulation are involved in the mammalian reproduction and embryonic development. The structure of TET3 protein, the roles of TET3 in regulating gametogenesis and embryonic development, and other TET3-related biological functions, were reviewed in this paper, so as to promote the functional research of TET3 and the application in the field of life sciences.
Keyword: Ten-eleven translocation 3; 5-methylcytosine; DNA methylation; Gametogenesis; Embryonic development;
DNA甲基化修饰是表观遗传学修饰最主要的方式之一,主要通过DNA甲基转移酶(DNA methyltransferases,DNMTs)将甲基转运至胞嘧啶-磷酸-鸟嘌呤(Cp G)中胞嘧啶上的第五位碳原子上[1],形成5-甲基胞嘧啶(5-methylcytosine,5-m C),阻碍RNA聚合酶结合,进而限制基因转录。在哺乳动物细胞中,DNMTs包括DNMT1、DNMT2、DNMT3A、DNMT3B和DNMT3L 5种[2],它们可以分为从头甲基化转移酶和维持型甲基化转移酶两类。从头甲基化转移酶主要包括DNMT3A、DNMT3B和DNMT3L 3种,在早期胚胎中表达较高,但是在向成体细胞分化过程中表达很低[2],这类酶可以先将DNA的一条链进行甲基化,然后再对另一条链进行甲基化,最终完成双链DNA甲基化。维持型甲基化转移酶主要是DNMT1,其作用于DNA复制过程中,促使新合成的子链DNA按照母链的DNA甲基化位点进行甲基化,以维持分裂细胞基因组DNA的特定甲基化模式。DNMT1在成体细胞中持续表达,参与细胞增殖、器官发育和肿瘤发生等多方面调控[3]。研究表明这些DNMTs的功能有相互交叉作用,DNMT1也具有从头甲基化的活性,DNMT3A、DNMT3B和DNMT3L也具有维持DNA甲基化的作用[4]。研究发现DNMT2不能进行DNA甲基化,而是一种RNA甲基化转移酶,主要负责天冬氨酸t RNA的甲基化修饰,在胚胎期内脏、肌肉、卵巢和睾丸细胞中高水平表达[5]。DNA甲基化介导的表观遗传修饰作用,保证了胚胎和生殖发育的有序进行。

TET(ten-eleven translocation)家族介导的DNA去甲基化广泛存在于各种生命活动调控过程。TET1在胚胎发育中率先被激活,对多能干细胞的干性维持具有重要作用,胚胎干细胞中TET1表达下调可导致干细胞分化和多能性丧失[6]。TET2在造血系统中高表达,影响造血干细胞的增殖与分化,TET2表达缺失将导致功能血液细胞分化受阻,引起恶性血液肿瘤[7]。研究显示TET3在早期胚胎向神经分化过程中表达上调[8],促进神经发育。也有证据显示TET3在配子发育中持续表达[9],介导配子基因组甲基化调控,TET3表达缺失可以引起早期胚胎发育延迟,出生后小鼠生存率低。综述TET3在配子和胚胎发育中的调控作用,以期阐述TET3的生物学功能。
1 、TET家族介导DNA去甲基化作用
DNA去甲基化作用一般可以通过两种方式进行,一种是由于DNMTs活性降低或者入核受阻等原因[10],使得DNA复制后产生的新链DNA无法进行甲基化修饰,并且不能维持旧链的甲基化模式导致总体DNA甲基化水平下降,这种方式称为被动去甲基化;另一种去甲基化方式则是由TET家族(包括TET1、TET2和TET3)介导的5-m C氧化作用,这种方式主要以主动的方式进行(见图1)[11]。
TET家族成员可以将5-m C氧化生成5-羟甲基胞嘧啶(5-hydroxymethyluracil,5-hm C)、5-甲酰基胞嘧啶(5 formylcytosine,5-f C)和5-羧甲基胞嘧啶(5carboxylcytosine,5-ca C)[11],其中5-hm C在胞苷脱氨酶(activation-induced cytidinedeaminase,AID)作用下脱氨生成5-羟甲基尿嘧啶(5-hydroxymethyluracil,5-hm U),5-hm U被胸腺DNA糖化酶(thymine-DNA glycosylase,TDG)识别并切除,激活碱基切除修复(base excision repair,BER)途径,实现DNA主动去甲基化。此外,AID也可促进5-m C生成胸腺嘧啶[12],引起TDG切除完成主动去甲基化。5-f C和5-ca C可被TDG识别并切除,启动细胞内BER途径,将其替换为胞嘧啶,最终完成DNA的主动去甲基化。TET家族氧化5-m C生成的5-hm C也会使DNMT1的活性降低,随着DNA复制过程的进行,DNMT1不能维持DNA新链上的甲基化水平,导致被动去甲基化,而5-m C氧化生成的5-hm C、5-f C和5-ca C在DNA复制过程中不断被稀释,最终完成被动去甲基化[13]。而在细胞分裂过程中DNMT1是否会抑制TET家族去甲基化作用从而维持DNA的甲基化状态,目前尚无证据。
TET3在生殖发育中发挥重要调控作用,其在卵母细胞中高表达,介导受精卵中父系基因组和母系基因组的去甲基化调控。但研究发现在受精卵中TDG表达水平非常低,当DNA复制受阻时可以导致5-m C氧化产物的积累[14],提示在受精卵中TET3介导5-m C生成的产物可能并不是通过TDG清除的,而是依赖于DNA复制完成的。此外,TET3也受到一些因子的修饰调控,如乙酰氨基葡萄糖转移酶(O-linkedβ-N-acetyl glucosamine transferase,OGT)可以促使TET3向细胞核外转运[15],从而抑制其去甲基化调控作用;AID在初级卵母细胞中开始大量表达,在受精卵中可促进TET3的去甲基化作用[12]。
图1 TET家族介导DNA去甲基化过程
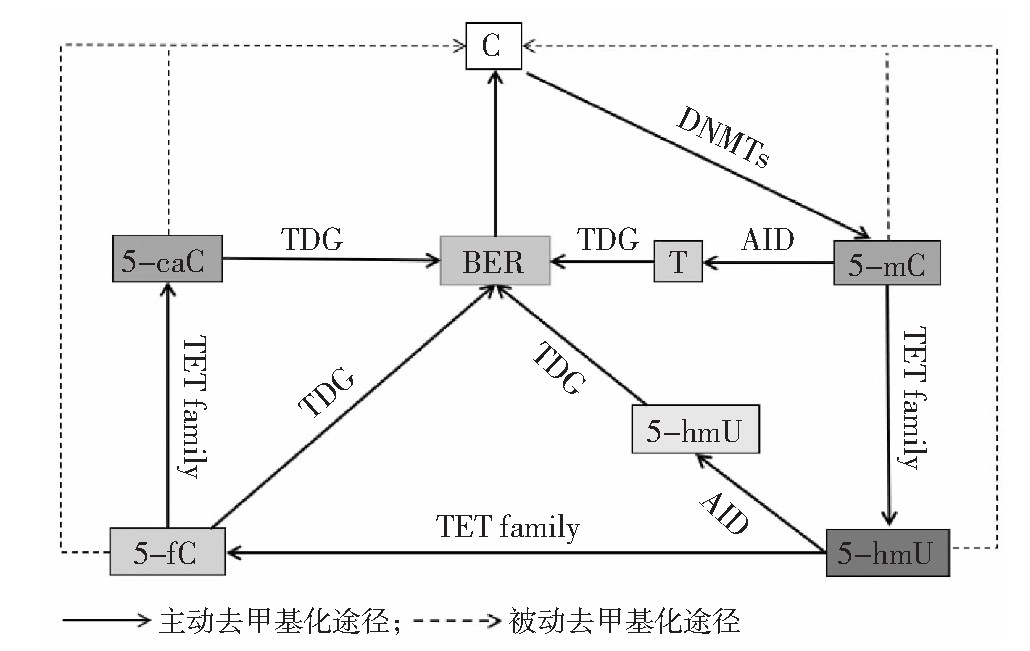
注:DNMTs DNA甲基转移酶;AID胞苷脱氨酶;TDG胸腺DNA糖化酶;BER碱基切除修复。
2、 TET3的结构特征
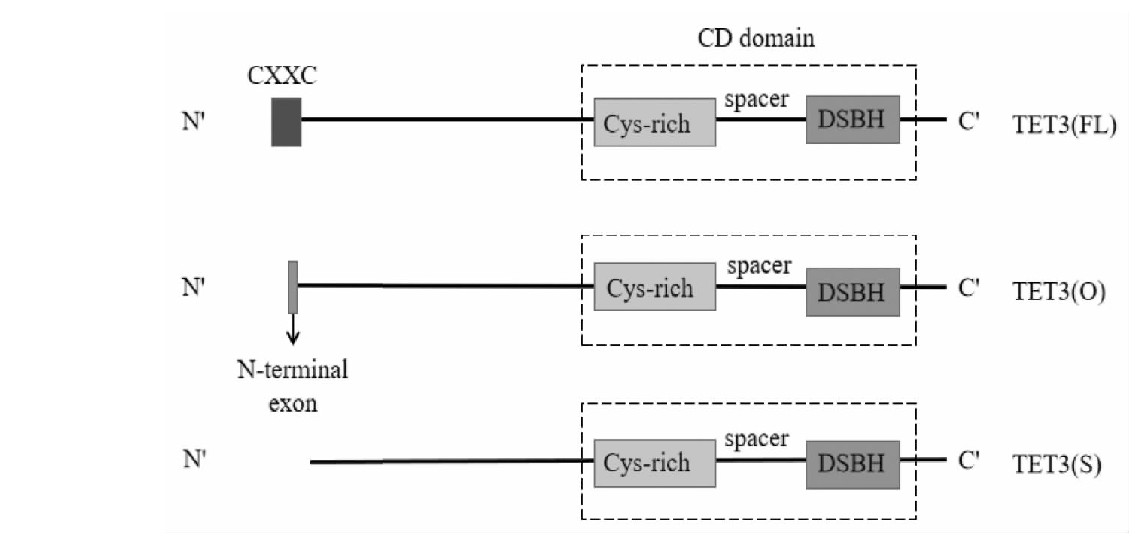
TET3蛋白主要存在3种亚型(见图2)[16],分别为TET3(FL)、TET3(O)和TET3(S)。其中TET3(FL)结构最复杂,由一个保守的C端催化域和一个N端调节域组成,C端的催化域由富含半胱氨酸(Cys-rich)、双链β螺旋依赖的双加氧酶区(DSBH)及间隔区组成,称为CD催化结构域,CD结构域可以与DNA上5-m C结合并氧化成为5-hm C;N端的调节域则由CXXC型锌指结构域组成,可以介导TET3(FL)优先识别和结合DNA中富含Cp G的区域[17]。TET3(O)是由位于TET3(FL)起始密码子上游约5 kb的另一种启动子转录表达的,N端缺失CXXC结构域,但替代的是含有11个氨基酸的小肽[16],这个小肽的作用机制尚不明确。TET3(S)和TET3(FL)是由同一个启动子转录翻译形成,但其N端的CXXC结构域序列被作为内含子选择性剪切而被去除;TET3 (S)的其他部分与TET3(FL)相同,与TET(O)相比少了11个氨基酸的小肽。研究发现TET3(S)和TET3(O)的氧化活性比TET3(FL)高,原因是CXXC结构域的优先识别能力使TET3(FL)在基因序列上的转移被限制,从而降低TET3(FL)对整体基因组的氧化活性[16]。TET3(S)和TET3(O)没有CXXC结构域,因此没有对特定基因组序列的识别能力。TET3(FL)和TET3(S)在胚胎干细胞向神经元分化过程中表达水平显着上调,而TET3(O)仅特异性表达于卵母细胞[18],提示TET3的3种亚型也具有组织和阶段特异性,分布在不同组织细胞内可能发挥不同的调控作用。
图2 TET3蛋白的亚型
3、 TET3在生殖发育中的调控作用
哺乳动物胚胎发育时,基因组经历了两次大规模的建立甲基化和去甲基化过程,TET家族在这个过程中也呈现动态变化(见图3)[19],表明TET家族介导的DNA甲基化调控在胚胎发育中具有重要作用。TET家族通过DNA甲基化调控实现了基因表达的时序性,促进胚胎发育和不同组织器官的分化。
3.1 、TET3在小鼠胚胎发育中的调控作用
TET家族在不同组织器官中的分工和调控作用存在很大差异。Gu等[20]通过条件性基因敲除将原始生殖干细胞(primordial germ cells,PGCs)中的TET3基因敲除,这类缺失TET3基因表达的雌性小鼠性成熟后和野生型雄性小鼠交配,后代中产生TET3杂合小鼠胚胎,研究显示这类胚胎可以正常发育至囊胚期,但在胚胎发育后期出现大量的死亡,只有少数小鼠出生,并且也存在很大比例的死亡。提示TET3在胚胎发育中发挥重要作用,猜测TET3表达不足造成早期胚胎基因组去甲基化水平不足[21],引起关键组织器官分化障碍,造成胎儿早期和出生后死亡。
TET1敲除的小鼠可以存活并具有生育能力[22],无形态和生育缺陷,但神经祖细胞增殖和神经发生相关的基因出现异常甲基化,小鼠表现出短期记忆和空间学习能力下降的现象。TET2敲除的小鼠同样具有正常形态和生育能力[7],但会引起造血干细胞或造血祖细胞的自我更新能力增强,使造血系统的发育出现异常,导致血液肿瘤的产生。
TET1/2均敲除的小鼠也可以存活并具有生育能力[23],但有一部分胚胎会在胚胎晚期及出生后死亡,存活的雌性小鼠卵巢变小、生殖能力降低,雄性却表现正常。研究显示在PGCs中,敲除TET1/2会导致减数分裂相关基因的缺陷,如联会复合体蛋白1(synaptonemal complex protein 1,Sycp1)等基因表达下降[24],从而导致雌鼠卵母细胞数量显着减少、生殖能力降低和卵巢体积显着变小。敲除TET1/2的PGCs印记基因会出现超甲基化,但敲除TET1/2雄性小鼠的后代中印记基因异常发生率较低[23],这可能与TET3在出生后动物的精子发育中发挥主要调控作用有关,提示在TET1/2缺失的情况下,TET3在生殖细胞发育过程中起到补偿作用,而在PGCs期TET3表达水平低,无法起到对TET1/2缺失的补偿作用。敲除TET1/3的小鼠胚胎发育至10.5 d (embryonic day10.5,E10.5)后均不能继续发育,与正常胚胎相比出现发育延迟、胚胎期细胞凋亡等[25],显示TET1和TET3在早期胚胎发育中具有重要作用。这些实验证实TET家族介导的去甲基化作用,促使小鼠胚胎正常发育和正常的配子发育功能,其中TET3在小鼠早期胚胎发育中起主要作用。
3.2 、TET家族在PGCs中的调控作用
在小鼠中,PGCs在胚胎发育至E6.25的上胚层中出现(见图3)[26],此后经历迁移和定植,最终分化为精子或卵子。PGCs通过甲基化修饰建立印记和向配子分化,其发育过程经历了两个阶段的全基因组去甲基化过程[27]。第一阶段发生在PGCs增殖和迁移期间的E7.25~E9.5,虽然这个阶段中TET1/2存在表达,而TET3的表达水平很低,但是研究表明这个时期5-m C去甲基化主要依赖于DNA复制被动去除[28]。第二阶段在E9.5~E13.5,该阶段TET1/2在PGCs中也存在表达,并在E10.5~E11.5达到峰值[26],而TET3表达仍然处于很低的水平,研究证明在这个时期中TET1/2介导了PGCs基因组的主动去甲基化,但研究发现敲除TET1/2后PGCs发育过程中整体基因组的去甲基化并未受到影响[28],表明这个阶段PGCs基因组去甲基化也可以通过DNA复制来完成。
3.3 、TET3在精子发育中的调控作用
精子发育是一个极其复杂的细胞分化和形态改变的过程。雄性小鼠性腺中的PGCs在E13.5后开始建立父方印记,雄性小鼠出生之后PGCs已分化为生殖母细胞(gonocyte)[29],然后从曲细精管中间迁移至曲细精管基底膜上分化成为A型精原细胞,随后精原细胞经过一系列分化生成精母细胞,再经过减数分裂后产生单倍体精子细胞,单倍体精子通过剧烈的形态变化形成成熟的精子(见图3)。精子发育是逐渐建立甲基化和关闭功能基因的过程,DNA甲基化修饰在精子发育过程中起到了非常重要的作用。研究显示小鼠精子发生过程中8个阶段的精原细胞5-hm C的表达呈动态变化[9],刚分化的精原细胞表达水平最高,然后逐渐降低,在所有类型的精原细胞里TET3的表达水平都显着高于TET1/2,说明在精原细胞分化过程中5-hm C主要由TET3氧化产生,随着精子发育的进行,精子基因组逐渐建立高甲基化的过程,TET家族和5-hm C表达水平都随之降低。5-hm C在精子细胞的一些功能基因和pi RNA (Piwi-interacting RNA)前体基因的启动子区域呈现的高水平分布[9],促进了这些功能基因和pi RNA的转录,对维持精子发育起到了重要的调控作用。而TET家族对精原细胞分化的调控作用尚未有更好的阐释,其中表达最高的TET3是否参与维持精子分化调控也没有相关数据。
生殖干细胞和胚胎干细胞基因组5-hm C表达水平相近[9]。有证据显示TET3在小鼠精原干细胞中高表达[30],而TET1/2则在胚胎干细胞中高表达[23],这也说明TET3对精原干细胞的5-hm C起主要的调控作用,而胚胎干细胞中5-hm C表达水平则是依靠TET1/2氧化产生,揭示了TET家族在发育阶段的特异性和细胞表达的差异性。
3.4 、TET家族在成熟精子中的调控作用
正常成熟精子的5-hm C含量只有血液的32.59%[31],这与正常成熟精子基因组的低转录活性相一致,5-hm C的低表达使得成熟精子基因组的转录活性显着低于血液。研究发现TET1和TET3在人圆形精子细胞至伸长型精子细胞中持续表达,并定位于细胞核中,患有弱精子症的男性精子中TET1和TET3表达水平显着低于正常男性,提示TET1和TET3的表达水平与精子活力和凋亡调控息息相关[32]。通过分析各年龄段成年男性成熟精子中5-m C和5-hm C的表达水平与精子质量的相关性,发现随着男性年龄增长精子中5-m C的年增长率为1.76%,而5-hm C的年增长率约为5%,一般而言精子整体DNA甲基化模式是稳定的,而老年男性精子中5-m C含量的增加导致精子活力变差[31]。同时,老年男性精子中5-hm C的增涨幅度比5-m C更大,意味着老年男性精子与青年男性精子相比,5-hm C在基因组中发生异常增加,从而可能导致成熟精子基因组中发生转录激活,这也说明成熟精子中5-hm C水平对于维持精子基因组甲基化模式的稳定和受精能力同样具有重要作用[33],但目前尚无证据证明这些变化是否对后代产生变异,而精子中5-hm C表达水平是否由TET3调控产生也缺乏相关数据证明。
图3 生命周期中TET3表达水平
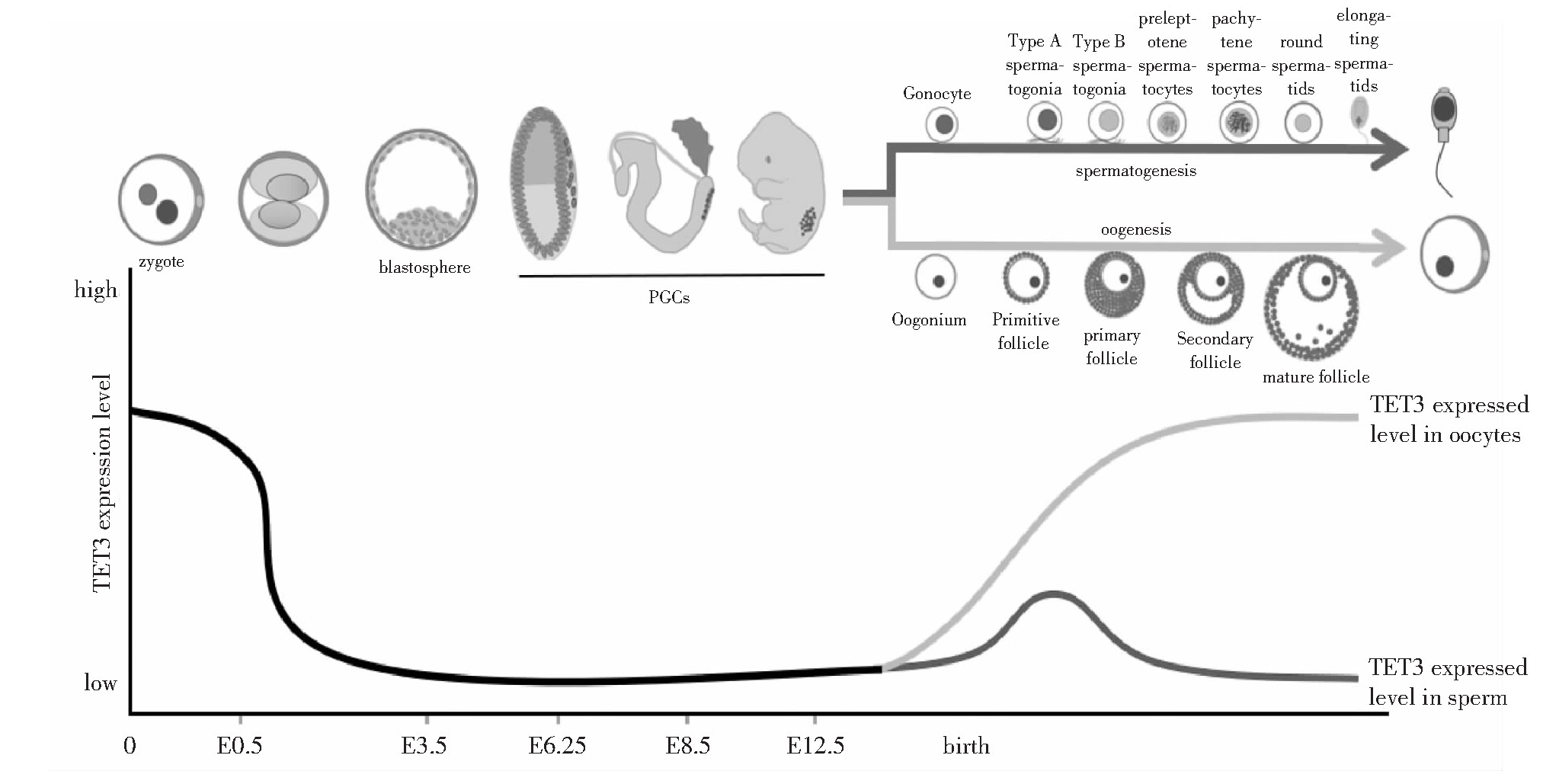
注:zygote受精卵;blastosphere囊胚;PGCs原始生殖干细胞;Gonocyte生殖母细胞;Type A spermatogonia A型精原细胞;Type B spermatogonia B型精原细胞;preleptotene spermatocytes细线期精母细胞;pachytene spermatocytes粗线期精母细胞;round spermatids圆形精细胞;elongating spermatids伸长型精细胞;Oogonium卵原细胞;Primitive follicle原始卵泡;primary follicle初级生长卵泡;Secondary follicle次级卵泡;mature follicle成熟卵泡;TET3 expressed level in oocytes卵子中TET3表达水平;TET3 expressed level in sperm精子中TET3表达水平;spermatogenesis精子发生;oogenesis卵子发生。
3.5 TET3在卵母细胞中的调控作用
与精子发育过程不同,卵母细胞发育过程建立的甲基化大约只有基因组的40%,大片的低甲基化区域与其高转录活性相对应[10]。卵母细胞内富含促进早期胚胎发育的功能蛋白的RNA,是胚胎发育的重要物质保证。研究显示在体细胞核移植的胚胎中普遍存在异常甲基化的现象[34],体细胞核移植后TET3在卵母细胞中过表达,可以启动DNA去甲基化,提高了体细胞核移植胚胎的发育率。TET3的表达及调控功能在卵母细胞中受到诸多因子调节。在初级卵母细胞中TET3被Cullin-RING泛素连接酶4 (Cullin-RING finger ligase-4,CRL4)复合物调控[35],维持初级卵母细胞发育相关基因如Sohlh1/2、Nobox和Figla等的表达。PGC7作为重要的母源因子在卵母细胞中高表达[36],其可以与TET3结合,抑制TET3的氧化活性,保护受精后母系基因组不受TET3介导的去甲基化作用,同时PGC7也可以促使印记基因(母系印记基因Peg1、Peg3、Peg5和父系印记基因H19和Rasgrf1)不被去甲基化。也有研究显示PGC7缺失可使卵母细胞的基因组过甲基化[37],达到与精子基因组甲基化相似的水平,导致成熟卵子质量降低。此外,延伸复合体3(elongator complex 3,ELP3)在卵母细胞中开始表达[38],在受精卵基因组激活后表达下降,并参与了受精卵中雄原核的去甲基化,但其是否与TET3相互作用尚不清楚。
4 、展望
随着表观遗传修饰研究方法的不断发展,已有研究揭示DNA甲基化在胚胎发育中的动态变化,而TET3在生殖发育过程中的表达提示TET3介导的DNA去甲基化在其中发挥了重要的调控作用,但其调控机制有待进一步明确。既往研究对TET1/2介导的DNA去甲基化调控在早期胚胎发育及肿瘤发生中的作用已经进行了很好的阐释,但对TET3的调控功能还存在很多未知。本文重点对TET3介导甲基化调控在配子和胚胎发育中的作用进行阐述,以期促进TET3的功能性研究,完善在生殖发育中TET3介导的表观遗传学调控机制研究。
参考文献
[1]Wikenius E,Moe V,Smith L,et al.DNA methylation changes in infants between 6 and 52 weeks[J].Sci Rep,2019,9(1):17587.
[2]Gowher H,Jeltsch A.Mammalian DNA methyltransferases:new discoveries and open questions[J].Biochem Soc Trans,2018,46(5):1191-1202.
[3]Lyko F.The DNA methyltransferase family:a versatile toolkit for epigenetic regulation[J].Nat Rev Genet,2018,19(2):81-92.
[4]宋乔,高世超,王培昌.DNA甲基化的分子机制及其研究进展[J].基因组学与应用生物学,2019,38(7):3317-3322.
[5]Zhang Y,Zhang X,Shi J,et al.Dnmt2 mediates intergenerational transmission of paternally acquired metabolic disorders through sperm small non-coding RNAs[J].Nat Cell Biol,2018,20(5):535-540.
[6]Ross SE,Bogdanovic O.TET enzymes,DNA demethylation and pluripotency[J].Biochem Soc Trans,2019,47(3):875-885.
[7]Zeng H,He H,Guo L,et al.Antibiotic treatment ameliorates Teneleven translocation 2 (TET2) loss-of-function associated hematological malignancies[J].Cancer Lett,2019,467:1-8.
[8]Wang L,Li MY,Qu C,et al.CRISPR-Cas9-mediated genome editing in one blastomere of two-cell embryos reveals a novel Tet3function in regulating neocortical development[J].Cell Res,2017,27(6):815-829.
[9]Gan H,Wen L,Liao S,et al.Dynamics of 5-hydroxymethylcytosine during mouse spermatogenesis[J].Nat Commun,2013,4:1995.
[10]卢绪坤,李元元,颉伟.哺乳动物早期胚胎发育中表观遗传信息的传递和重编程[J].中国细胞生物学学报,2019,41(5):822-833.
[11]Wu X,Li G,Xie R.Decoding the role of TET family dioxygenases in lineage specification[J].Epigenetics Chromatin,2018,11(1):58.
[12]Bochtler M,Kolano A,Xu GL.DNA demethylation pathways:Additional players and regulators[J].Bioessays,2017,39(1):1-13.
[13]Klungland A,Robertson AB.Oxidized C5-methyl cytosine bases in DNA:5-Hydroxymethylcytosine;5-formylcytosine;and 5-carboxycytosine[J].Free Radic Biol Med,2017,107:62-68.
[14]Santos F,Peat J,Burgess H,et al.Active demethylation in mouse zygotes involves cytosine deamination and base excision repair[J].Epigenetics&chromatin,2013,6(1):39.
[15]熊俊,朱冰.TET家族蛋白介导的DNA氧化的调控与其生物学功能[J].生命科学,2017,29(10):926-933.
[16]Jin SG,Zhang ZM,Dunwell TL,et al.Tet3 Reads 5-Carboxylcytosine through Its CXXC Domain and Is a Potential Guardian against Neurodegeneration[J].Cell Rep,2016,14(3):493-505.
[17]Xu Y,Xu C,Kato A,et al.Tet3 CXXC domain and dioxygenase activity cooperatively regulate key genes for Xenopus eye and neural development[J].Cell,2012,151(6):1200-1213.
[18]Liu N,Wang M,Deng W,et al.Intrinsic and extrinsic connections of Tet3 dioxygenase with CXXC zinc finger modules[J].PLo S One,2013,8(5):e62755.
[19]Zhu Q,Stoger R,Alberio R.A Lexicon of DNA Modifications:Their Roles in Embryo Development and the Germline[J].Front Cell Dev Biol,2018,6:24.
[20]Gu T,Guo F,Yang H,et al.The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes[J].Nature,2011,477(7366):606-610.
[21]Tsukada Y,Akiyama T,Nakayama KI.Maternal TET3 is dispensable for embryonic development but is required for neonatal growth[J].Scientific Reports,2015,5(1).
[22]Zhang RR,Cui QY,Murai K,et al.Tet1 regulates adult hippocampal neurogenesis and cognition[J].Cell Stem Cell,2013,13(2):237-245.
[23]Dawlaty MM,Breiling A,Le T,et al.Combined deficiency of Tet1and Tet2 causes epigenetic abnormalities but is compatible with postnatal development[J].Dev Cell,2013,24(3):310-323.
[24]Yamaguchi S,Hong K,Liu R,et al.Tet1 controls meiosis by regulating meiotic gene expression[J].Nature,2012,492(7429):443-447.
[25]Kang J,Lienhard M,Pastor W A,et al.Simultaneous deletion of the methylcytosine oxidases Tet1 and Tet3 increases transcriptome variability in early embryogenesis[J].Proc Natl Acad Sci U S A,2015,112(31):E4236-E4245.
[26]Hackett JA,Sengupta R,Zylicz JJ,et al.Germline DNAdemethylation dynamics and imprint erasure through 5-hydroxymethylcytosine[J].Science,2013,339(6118):448-452.
[27]Furuta A,Nakamura T.DNA hypomethylation circuit of mouse r DNA repeats in the germ cell lineage[J].Biochem Biophys Res Commun,2017,490(2):429-433.
[28]Wu X,Zhang Y.TET-mediated active DNA demethylation:mechanism,function and beyond[J].Nat Rev Genet,2017,18(9):517-534.
[29]Kanatsu-Shinohara M,Shinohara T.Spermatogonial stem cell selfrenewal and development[J].Annu Rev Cell Dev Biol,2013,29:163-187.
[30]Bai Y,Feng M,Liu S,et al.Differential gene expression in mouse spermatogonial stem cells and embryonic stem cells[J].Int J Mol Med,2016,38(2):423-432.
[31]Jenkins TG,Aston KI,Cairns BR,et al.Paternal aging and associated intrainpidual alterations of global sperm 5-methylcytosine and 5-hydroxymethylcytosine levels[J].Fertil Steril,2013,100(4):945-951.
[32]Ni K,Dansranjavin T,Rogenhofer N,et al.TET enzymes are successively expressed during human spermatogenesis and their expression level is pivotal for male fertility[J].Human Reproduction,2016,31(7):1411-1424.
[33]Urdinguio RG,Bayón GF,Dmitrijeva M,et al.Aberrant DNAmethylation patterns of spermatozoa in men with unexplained infertility[J].Human Reproduction,2015,30(5):1014-1028.
[34]Han C,Deng R,Mao T,et al.Overexpression of Tet3 in donor cells enhances goat somatic cell nuclear transfer efficiency[J].The FEBSJournal,2018,285(14):2708-2723.
[35]孙乐,丁之德.卵母细胞发育和受精中的重编程及对雄原核重编程的影响[J].国际生殖健康/计划生育杂志,2015,34(3):234-238.
[36]Zhao S,Xu J,Liu S,et al.Dppa3 in pluripotency maintenance of EScells and early embryogenesis[J].J Cell Biochem,2019,120(4):4794-4799.
[37]Li Y,Zhang Z,Chen J,et al.Stella safeguards the oocyte methylome by preventing de novo methylation mediated by DNMT1[J].Nature,2018,564(7734):136-140.
[38]Okada Y,Yamagata K,Hong K,et al.A role for the elongator complex in zygotic paternal genome demethylation[J].Nature,2010,463(7280):554-558.
DNA甲基化作为一种动态的和可逆的表观遗传修饰,是由甲基转移酶(DNA methyltransferases,DNMTs)介导的对胞嘧啶的第5位碳原子进行甲基修饰,形成5-甲基胞嘧啶(5-methylcytosine,5m C),主要发生在Cp G二核苷酸位点上。...
真核细胞的基因表达是高度复杂的过程,是遗传调控和表观遗传调控协同作用的结果.遗传调控以基因决定论为核心内容,表观遗传学则主要研究DNA序列以外的调控机制.已有研究结果证实,基因行为可通过表观遗传的行为进行调控(Reik,2007),并可遗传给...
表观遗传学研究已经成为生命科学研究关注的前沿, 取得了很大的进展。而DNA甲基化、组蛋白修饰以及非编码RNA作为重要的植物表观遗传修饰方式, 共同参与植物基因的表达调控, 调节着植物的生长发育。...