
真核细胞的基因表达是高度复杂的过程,是遗传调控和表观遗传调控协同作用的结果.遗传调控以“基因决定论”为核心内容,表观遗传学则主要研究DNA序列以外的调控机制.已有研究结果证实,基 因 行 为 可 通 过 表 观 遗 传 的 行 为 进 行 调 控(Reik,2007),并可遗传给后代.现今基于细胞生物学的表观遗传研究方向,旨在破译表观遗传信号,哪些分子干预转录促使细胞自我更新和分化(图1).
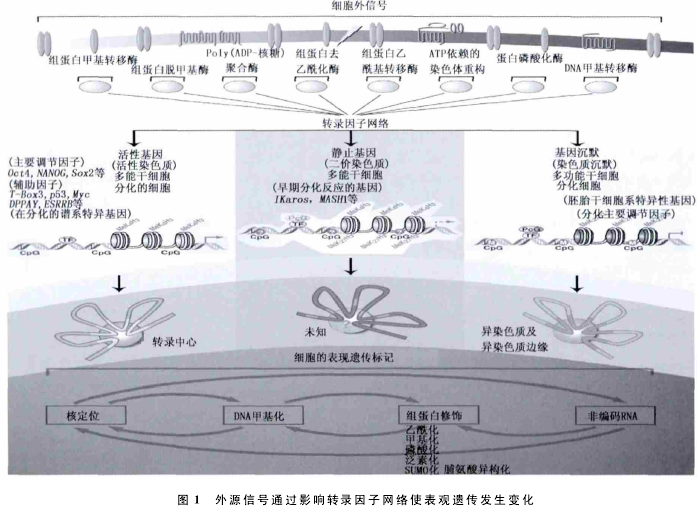
此外,解析甲基化的分子机制和不同细胞间表现形式的差异有助于关联细胞类型相关疾病的全基因组与差异甲基化区域(DMRS)之间的关系,这已成为遗传学的一个研究热点.作者就DNA甲基化对影响转录、分子调控、神经元发育、遗传等方面的最新研究成果进行综述.
1、转录与甲基化
表观基因组学的变化,包括DNA的化学修饰,可作为基因组的一层额外信息(Ray等,2013).甲基化,一种特殊的表观基因组学修饰形式,是一种发生在胞嘧啶核苷酸特别是鸟嘌呤前胞嘧啶(cyto-sine-phosphate-guauine,CpG)的共价修饰.这个过程是 催 化DNA甲 基 转 移 酶 (DNA methyltrans-ferases,DNMTs)转移甲基S-腺苷甲硫氨酸到目标胞嘧啶的碳5上.此外还发现这种转移酶家族的2个成员:DNMT1,主要功能是在DNA复制过程中维持DNA甲基化;DNMT3(包括DNMT3a和DN-MT3b),被认为是主要负责起始的CpG甲基化(蒙丽娜等,2013).在基因转录区域以外,基本都是甲基化的CpG.
CpG含量高的区域,被称为CpG岛,在哺乳动物启动子中发现其含量约40%(Nigro等,1989),而在非CG序列上则被称为non-CG(Dowen等,2012).研究结果表明,启动子CpG岛的甲基化状态可能是控制基因表达的一个重要因素,用大量的甲基化阻断基因转录,反之,则促进.此外,有证据表明,整个区域染色质的高甲基化可介导一整段基因的沉默,甚至延伸到相邻的非甲基化基因(Bert等,2012).
DNA甲基化的精确调控在正常细胞分化及基因组印迹的过程中(一对等位基因中部分基因的特殊表达现象)十分重要.在肿瘤细胞中,能频繁观察到异常模式的DNA甲基化.一般情况下,癌细胞拥有全基因组的低甲基化和肿瘤抑制相关因子启动子的超甲基化.由于全基因组的低甲基化导致基因组不稳定、基因印记损失,因而过度表达致癌基因(Trimarchi等,2011).与其相比,其余的基因组如肿瘤抑制基因的启动子通常会表现出超甲基化,表明肿瘤抑制基因的表观遗传沉默与基因突变导致功能丧失具有同样的效果,前者是后者的一种有效替代.
每种细胞个体的表观遗传都是通过早期进化结合CpG和蛋白修饰来协调或标记组织特异性基因的表达模式来形成的.在正常细胞中,大部分基因组是被DNA甲基化的,但与CpG岛启动的激活或使之变为双价的基因通常未被甲基化(Cedar等,2012),这 种 缺 乏 甲 基 化 的 机 理 尚 不 清 楚.虽 然CpG二核苷酸是DNA甲基转移酶(DNA methyl-transferase enzymes,DNAMTases)的首要目标,但DNAMTases从DNA一开始的甲基化到正常的发育和分化过程中基本上仍保持CpG二核苷酸的活性,这是否是由于活化了早已进化好的转录还是抑制了转录机制从而掩盖DNAMTases上的CpG位点,亦或是激活去甲基化的作用,具体机制仍有待探索(Clark等,2002).在癌细胞中,CpG岛启动子高度甲基化常伴随组蛋白抑制的标记基因的表达量增多(Baylin等,2011),这种通过“远程的表观遗传沉默”(LRES)的现象,表明肿瘤发生过程中表观遗传不仅限于单个基因的失活,也可以是整个基因组的失活.典型的LRES特点一般都是通过增加CpG岛的甲基化,抑制组蛋白修饰的H3K9二甲基化(H3K9me2)和H3K27三甲基化(H3K27me3),并结合失活的H3K9乙酰化(H3K9ac)一同来实现(Coolen等,2010).但最初公布在肿瘤中发现的全基因 组 低 甲 基 化 的 状 态,却 是 表 观 遗 传 的 特 例(Feinberg等,1988).长距离的低甲基化区域能覆盖近一半的癌症基因组(Cortessis等,2012),且在体细胞内也通常发现有部分甲基化的区域(Lister等,2013).传统意义上认为,癌症与LINE-1元素及其他重复序列的去甲基化有关(Kim等,2010),重复序列被发现与激活替代转录(Ross等,2010)和原癌基因过度表达有相关联系(Ley等,2010),且CpG启动子基因的去甲基化也被证明存在于多个癌症的单独基因里(Nishigaki等,2005).目前对特定基因的表观遗传激活已被证实是存在的,但专门针对在肿瘤细胞内全基因组表观遗传激活的范围和基因组的相关关系却鲜见报道.
2、双加氧酶与胸腺嘧啶DNA糖基化酶对甲基化调控的机制
甲基化一直被视作是一种稳定的DNA标记,研究结果发现,只有在如受精这样的全面重编程事件中它才会消失,可最近的研究结果表明,DNA甲基化并不是静态的,绝大部分的基因组都是以甲基化和去甲基化动态平衡的形式存在(Song等,2013;Shen等,2013).针对此不同的研究结果是否可以这样设想:这一效应是否可以被积极地逆转?能够证实这一假设正确性的办法就是找到并阻断其过程中的一个步骤来阻止这一周期性的变化.
最近有报道称,双加氧酶(ten-eleven transloca-tion,TET)蛋白可将甲基化的胞嘧啶(5-mC)氧化为5-羟 甲 基 胞 嘧 啶 (5-hydroxymethylcytosin,5-hmC),经研究证实,TET还可进一步氧化5-hmC为5-胞嘧啶甲酰(5-formylcytosine,5-fC)和5-胞嘧啶羧基(5-carboxytosine,5-caC),这些表观遗传学修饰也被称为DNA的第5和6种,第7和8种碱基(Ito等,2011).然而,发现这些修饰碱基主要存在于小鼠胚胎干细胞中的DNA重复区域(Shen等,2013).通过碱基切除修复和使用高纯度的单抗富集这些标记发现,胸腺嘧啶DNA糖基化酶(thy-mine DNA glycosylase,TDG)会用未修饰的胞嘧啶来代替5-fC和5-caC,并能选择性地剪切由TET蛋白催化5-mC氧化产生的5-fC及5-caC,继而启动下游DNA剪切修复(base-excision repair,BER)通路,将5-mC还原成未修饰的胞嘧啶,而在耗尽TDG的 细 胞 中5-fC与5-caC产 生 积 累 (He等,2011),说明甲基化酶TET和TDG蛋白运转了甲基化-去甲基化周期,并证实了甲基化是动态且广泛的.
Wang等(2013)通过分析去甲基化中间体,结果发现其去阻遏是一种激活机制,很多被认为是未甲基化的区域其实上是在不断地去甲基化.通过绘制5-hmC/5-fC/5-caC全基因组分布图谱,结果发现,在野生型和Tdg缺陷的小鼠胚胎干细胞(ESCs)中可检测到5-fC/5-caC在野生型小鼠的ESCs卫星重复序列上累积,相比之下,Tdg缺陷小鼠ESCs则在大量的近端和远端基因调控元件上显著累积了5-fC和5-caC(Shen等,2013).虽然目前对于这些去甲基化过程发生的程度及在基因组中的区域还不是很明确,但这些研究结果使研究人员进一步了解了DNA甲基化在细胞发育和功能方面所起的作用.
3、神经元的甲基化
大脑最前面的一部分称为额叶皮质,额叶皮质对于思考、行动能力的形成起着重要作用.大脑实现这一切可通过特殊细胞的交互如神经元和神经胶质.众所周知,这些细胞都有其独特的功能,但如何定义这些细胞本身?答案在于每个细胞如何通过内在的DNA表达信息.大脑基因组甲基化从婴儿到成年的整个过程中是以“non-CG”这种DNA甲基化的形式来呈现的,可以说是神经元专有的,且是神经元基因组的主要甲基化形式.另外,在研究诱导多能干细胞中发现其DNA甲基化形式也为“non-CG甲基化”(Lister等,2013).最初,研究者认为当干细胞分化为特殊的组织时这种甲基化形式就会消失,如肺脏细胞或脂肪细胞,但目前研究发现,这种甲基化形式并没有在大脑中消失,随着神经元细胞的分化仍 然 出 现 了 “non-CG甲 基 化”(Weng等,2013).随着对老鼠和人的神经元及神经胶质在出生后、青少年和成年阶段进行基因组测序,结果发现甲基化在神经元中逐渐积累,“non-CG甲基化”成为了神经元中的主要甲基化形式,表明成熟大脑神经元回路的建立是伴随着表观基因组学大规模重组的过程(Petanjek等,2012).这或许应对了最近的一些研究成果,如甲基化在精神分裂症、抑郁症、自杀和躁郁症疾病中扮演着潜在的角色,由于这些DNA甲基化的特点出现在大脑神经元发育的关键阶段,可影响神经网络的形成,反过来,此后可能引起与脑的功能和疾病有关的变化(Tan等,2012).
4、表观遗传记忆
正如之前所提到在生殖细胞的成熟过程中,绝大多数表观遗传修饰会被抹去,这种重置机制会删除相应的“环境记忆”,不过也会有一部分DNA将这种修饰保留下来.研究表观遗传这一重编程机制,不仅能加深研究者解析表观遗传学修饰在世代间传递的机制,还有助于对胚胎细胞发育的理解.
通过用Illumina测序平台生成高分辨率的甲基化图谱,标记未分化成熟的生殖细胞中DNA发生去甲基化的时机和位点,可观察到甲基化往往出现在不活跃的基因上,且在时间上显示大多数去甲基化事件比预想的要发生得更早.此外,令人兴奋的是研究人员发现了能避开去甲基化的DNA区域,这很可能就是将环境影响遗传给子代的机制(Seisen-berger等,2012).近年来有不少研究结果证实在哺乳动物中环境信息可传递到下一代,如高脂饮食的小鼠其后代的代谢也受到了影响,有过惊吓经历的雄鼠与雌鼠交配生下的小鼠比正常小鼠更胆怯,而同一只雄鼠通过人工授精所产下的小鼠则没有表现出异常行为等(Dietz等,2011).
Jiang等(2013)以斑马鱼为模型,结果发现,子代选择性地继承父本而抛弃母本的DNA甲基化图谱,研究人员共测量了斑马鱼卵子、精子、6个早期胚胎和精囊共9个时期全基因组的DNA甲基化序列,产生了约1200G的数据,结果发现,斑马鱼子代胚胎选择性地继承精子中的DNA甲基化,此研究结果被认为是对早期胚胎发育主要是由卵子决定这一传统观念的撼动.过去认为决定早期发育的信息几乎都在卵子中.研究结果证明,在斑马鱼中除了DNA可以从父母传递到子代外,精子的DNA甲基化图谱也可被遗传到子代中,用于指导胚胎早期发育(Jiang等,2013).
而形成精子的细胞与形成卵子的细胞相比,DNA去甲基化发生得更为频繁,意味着在表观遗传学修饰的遗传方面来自父方的作用可能更大.虽然目前还不清楚环境信息的遗传是如何发生的,但这些研究从机制上支持了上述现象.
5、小结
综上所述,目前对于表观遗传学机制的探索仍处于较初级的阶段.可以预见的是,应用高通量测序技术与单克隆抗体标识追踪技术来研究各种正常组织及癌症组织的DNA甲基化数据将不断涌现,利用这些不同组织的DNA甲基化数据,可以深层次地探讨表观遗传学在生物间的差异性及组织特异的表观遗传模式.虽然甲基化激活的分子通路及关键因子的发现也能在一定程度上解释细胞分化中的一些现象,但是否存在其他更多甚至更重要的因素参与其中及更系统全面的揭示其真实机理都有待于进一步的试验来推动.另外,结合其他表观遗传现象对人类基因组进行系统的研究势必成为医学上认识疾病基因表达的必然之路.利用这些与特定疾病相关的DNA甲基化标识对疾病进行检测,使之成为药物靶点也是极具热点的发展方向.
参 考 文 献
1蒙丽娜,罗婷,柯浩,等.DNA甲基转移酶抑制剂在动物体细胞核移植研究 中的应 用 [J].中 国 畜 牧 兽 医,2012,39(12):133~136.
2 Baylin S B,Jones P A.A decade of exploring the cancer epige-nome-biological and translational implications[J].Nature Re-views Cancer,2011,11(10):726~734.
3 Bert S A,Robinson M D,Strbenac D,et al.Regional activationof the cancer genome by long-range epigenetic remodeling[J].Cancer Cell,2012,23(1):9~22.
4 Cedar H,Bergman Y.Programming of DNA methylation pat-terns[J].Annual Review of Biochemistry,2012,81:97~117.
5 Clark S J,Melki J.DNA methylation and gene silencing in canc-er:Which is the guilty party[J].Oncogene,2002,21(35):5380~5387.
6 Coolen M W,Stirzaker C,Song J Z,et al.Consolidation of thecancer genome into domains of repressive chromatin by long-range epigenetic silencing(LRES)reduces transcriptional plastic-ity[J].Nature Cell Biology,2010,12(3):235~246.
7 Cortessis V K,Thomas D C,Levine A J,et al.Environmentalepigenetics:Prospects for studying epigenetic mediation of expo-sure-response relationships[J]. Human Genetics,2012,131(10):~1589.
8 Dietz D M,LaPlant Q,Watts E L,et al.Paternal transmissionof stress-induced pathologies[J].Biological Psychiatry,2011,70(5):408~414.
9 Dowen R H,Pelizzola M,Schmitz R J,et al.Widespread dy-namic DNA methylation in response to biotic stress[J].PNAS,2012,109(32):E2183~E2191.
10 Feinberg A P,Gehrke C W,Kuo K C,et al.Reduced genomic5-methylcytosine content in human colonic neoplasia[J].CancerResearch,1988,48(5):1159~1161.
11 He Y F,Li B Z,Li Z,et al.Tet-mediated formation of 5-car-boxylcytosine and its excision by TDG in mammalian DNA[J].Science,2011,333(6047):1303~1307.
12 Ito S,Shen L,Dai Q,et al.Tet proteins can convert 5-methyl-cytosine to 5-formylcytosine and 5-carboxylcytosine[J].Science,2011,333(6047):1300~1303.
13 Jiang L,Zhang J,Wang J J,et al.Sperm,but not oocyte,DNAmethylome is inherited by zebrafish early embryos[J]. Cell,2013,153(4):773~784.
14 Kim M S,Lee J,Sidransky D.DNA methylation markers incolorectal cancer[J].Cancer and Metastasis Reviews,2010,29(1):181~206.
15Ley T J,Ding L,Walter M J,et al.DNMT3A mutations inacute myeloid leukemia[J].NEJM,2010,363(25):2424~2433.
16Lister R,Mukamel E A,Nery J R,et al.Global epigenomicreconfiguration during mammalian brain development[J].Sci-ence,2013,341(6146):626~627.
17 Nigro J M,Baker S J,Preisinger A C,et al.Mutations in thep53gene occur in diverse human tumour types[J].Nature,1989,342(6250):705~708.
18 Nishigaki M,Aoyagi K,Danjoh I,et al.Discovery of aberrantexpression of R-RAS by cancer-linked DNA hypomethylation ingastric cancer using microarrays[J].Cancer Research,2005,65(6):~2124.
19Petanjek Z,Kostovi'cI.Epigenetic regulation of fetal brain devel-opment and neurocognitive outcome[J].PNAS,2012,109(28):11062~11063.
20 Ray M K,Wang Y,Borowsky M,et al.Identifying candidatencRNAs that direct changes in chromatin structure[J].Epige-netics & Chromatin,2013,6(1):69.
21 Reik W.Stability and flexibility of epigenetic gene regulation in mam-malian development[J].Nature,2007,447(7143):425~432.
22 Ross J P,Rand K N,Molloy P L.Hypomethylation of repeatedDNA sequences in cancer[J].Epigenomics,2010,2(2):245~269.
23Seisenberger S,Andrews S,Krueger F,et al.The dynamics ofgenome-wide DNA methylation reprogramming in mouse primor-dial germ cells[J].Molecular Cell,2012,48:849~862.
24Shen L,Wu H,Diep D,et al.Genome-wide analysis revealsTET-and TDG-dependent 5-methylcytosine oxidation dynamics[J].Cell,2013,153(3):692~706.
25Song C X,Szulwach K E,Dai Q,et al.Genome-wide profilingof 5-formylcytosine reveals its roles in epigenetic priming[J].Cell,2013,153:1~14.
26 Tan S L,Nishi M,Ohtsuka T,et al.Essential roles of the his-tone methyltransferase ESET in the epigenetic control of neuralprogenitor cells during development[J].Development,2012,139(20):3806~3816.
27 Trimarchi M P,Mouangsavanh M,Huang T H.Cancer epige-netics:A perspective on the role of DNA methylation in acquiredendocrine resistance[J].CJC,2011,30(11):749.
28 Wang T,Wu H,Li Y,et al.Subtelomeric hotspots of aberrant5-hydroxymethylcytosine-mediated epigenetic modifications dur-ing reprogramming to pluripotency[J].Nature Cell Biology,2013,15(6):700~711.
29 Weng Y L,An R,Shin J,et al.DNA modifications and neuro-logical disorders[J].Neurotherapeutics,2013,10(4):556~567.
胚胎干细胞(embryonicstemcells,ES细胞)具有无限增殖和多向分化的特性,在体外可分化为3个胚层的所有细胞[1],定向诱导人ES细胞分化为造血干/祖细胞,可以为造血干细胞移植提供新的细胞来源.目前诱导人ES细胞向造血细胞分化多采用ES细胞与基质细胞...