
细鳞鲑(Brachymystax lenok)隶属于鲑形目鲑科鲑亚科。在我国主要分布在东北地区黑龙江流域、绥芬河、图们江、鸭绿江, 新疆的额尔齐斯河, 河北省北部白河及滦河上游以及秦岭北麓渭河流域。
李思忠根据幽门盲囊、侧线鳞和最小性成熟年龄等形态学和生理学特征, 把分布在秦岭地区的细鳞鲑 定 名 为 细 鳞 鲑 的 一 个 新 亚 种 - 秦 岭 亚 种(Brachymystax lenok tsinlingensis Li), 为我国特有种。由于其肉质鲜美、营养价值高, 早在1938年就被人工引入到 湑水河中进行人工养殖。近年来环境污染加剧、不当的开发和过度捕捞等原因的影响,秦岭细鳞鲑资源量急剧减少, 呈点状分布在渭河上游部分支流中。1998年秦岭细鳞鲑被收录于《中国濒危动物红皮书-鱼类》中, 属濒危物种, 国家Ⅱ级保护水生野生动物。目前, 针对秦岭细鳞鲑的资源调查、生物学、胚胎发育和人工繁殖等方面的研究已有大量的报道, 然而对其遗传背景的研究较少, 仅见原居林等采用RAPD技术对黑河种群和 湑水河种群的遗传多样性进行了分析。为了逐步恢复和保护秦岭细鳞鲑的种质资源, 对其自然种群数量进行合理的补充, 了解人工繁育群体和野生群体的遗传背景是十分重要的。
线粒体DNA (mtDNA)由于结构简单、进化速度快和几乎不发生重组等特点, 使其成为研究物种起源、系统发生和种内遗传结构的有效遗传标记,其中一些基因被广泛应用在鱼类的种群遗传变异和系统发育分析。王培欣等利用线粒体D-loop区分析得出8个流域叉尾斗鱼种群遗传多样性很低且存在地理差异; 高天翔等利用线粒体Cytb基因分析得出松江鲈8个群体的遗传多样性较高; 张迪等利用线粒体COⅠ基因序列分析太湖新银鱼的遗传多样性, 得出有人工移植历史种群遗传多样性较高。本研究利用线粒体D-loop区和Cytb基因序列对秦岭细鳞鲑人工繁育群体和野生群体的遗传多样性进行比较分析, 以期为秦岭细鳞鲑放流效果的跟踪监测、种质资源遗传保护提供重要依据。
1 材料与方法
1.1 样品采集和 DNA 提取
收集自渭河上游支流马鹿河野生秦岭细鳞鲑为野生群体, 将收集自马鹿河和渭河另一支流西河的秦岭细鳞鲑人工驯化后, 繁殖的子一代为繁育群体,各采集 43 个尾鳍样品, 用无水乙醇固定带回实验室进行分析, 采用酚/氯仿法提取基因组 DNA。
1.2 PCR 扩增和序列测定
扩增 mtDNA 控制区序列的引物: 正向引物为:LRBT-25: 5′-AGAGCGCCGGTGTTGTAATC-3′; 反向 引 物 为 : LRBY-1195:5′-GCTAGCGGGACTTTCTAGGGT-3′。PCR 反应体系为 25 μL, 其中包括 1 UTaqDNA 聚合酶(TaKaRa), 1 μL dNTPs (2.5 mmol/L),5 μL 10?Taq buffer (TaKaRa, 含 Mg2+), 两条引物(10 mmol/L)各 1 μL, 3 μL DNA 模板, 其余双蒸水补足。PCR 反应程序为: 94℃预变性 3min; 94℃变性45s, 55℃退火 45s, 72℃延伸 45s, 共 30 个循环; 反应结束后在 72℃再延伸 8min。PCR 产物经琼脂糖凝胶电泳检测后送上海美吉生物工程公司纯化并测序, 测序引物为扩增引物。
扩增线粒体 Cytb 基因序列的引物: L14724(5′-GACTTGAAAAACCACCGTTG-3′); H15915 (5′-CTCCGATCTCCGGATTACAAGAC-3′)。PCR 反应体系为 25 μL, 其中包括 1 U TaqDNA 聚合酶(TaKaRa), 1 μL dNTPs (2.5 mmol/L), 5 μL 10×Taqbuffer (TaKaRa, 含 Mg2+), 两条引物(10 mmol/L)各1 μL, 3 μL DNA 模板, 其余双蒸水补足。PCR 反应程序为: 94℃预变性 5min; 94℃变性 45s, 50℃退火45s, 72℃延伸 45s, 共 35 个循环 ;反应结束后在72℃再延伸 10min。PCR 产物经琼脂糖凝胶电泳检测后送上海美吉生物工程公司纯化并测序, 测序引物为扩增引物。
1.3 数据分析
测序获得的序列通过 Chromas 1.45 软件获得原始序列数据; 利用 CLUSTAL X2 软件对所有序列进行 比 对 , 参 照 测 序 图 进 行 人 工 校 正 。 利 用PAUP*V4.0 软件进行同质性检验(Parition homoge-neity test)序列片段联合分析的可靠性; 用 DnaSP5.0 软件计算单倍型数、单倍型多样性和核苷酸多样性等; 运用 Arlequin 3.1软件中的分子变异(AMOVA)方法估算遗传变异在群体内和群体间的分布及遗传分化指数 Fst值及其 P 值(用排列测验法, 1000 次重排后的显着性检验), 根据 Nm=(1–Fst)/2Fst得到种群间的基因流值; 用 Mega 4.0 软件统计碱基组成, 并基于Kimura 2-papamter模型计算单倍型及群体间的遗传距离, 用邻接法(Neighbor-joining, NJ)构建系统进化树, 系统树中节点的自举置信水平应用自引导估计, 循环次数为 1000 次。Network 软件构建单倍型网络图, 用以检测单倍型之间的进化关系。
2 结果
2.1 序列分析
对野生群体和繁育群体的线粒体控制区序列进行比对排序后, 得到 730 bp 的同源序列。43 尾繁育群体所得序列共包含 20 个核苷酸变异位点, 其中简约信息位点 15 个, 单突变位点 5 个。所有序列间没有出现缺失与插入, 4 个转换位点, 1 个颠换位点, 平均转换与颠换数比值为 4.3。在野生群体中, 43 个个体共检测到 26 个变异位点, 其中简约信息位点 21个, 单突变位点 5 个, 平均转换与颠换数比值为3.3。在秦岭细鳞鲑养殖群体与野生群体中, 86 个个体共检测到 27 个变异位点, 其中简约信息位点 26个, 单突变位点 1 个, 平均转换与颠换数比值为3.4。碱基组成分析显示, A、T、C 和 G 碱基平均含量分别为 31.9%、31.6%、20.8%和 15.7%, 其中 A+T含量(63.5%)明显高于 G+C 含量(36.5%), 表现出明显的 AT 偏好和反 G 偏倚, 与其他脊椎动物线粒体控制区核苷酸的组成特点相一致。
获得 86 尾个体线粒体 Cytb 基因全序列共1141 bp。所得的序列共包含 25 个变异位点, 其中简约信息位点 24 个, 单突变位点 1 个。A、T、C 和 G碱基平均含量分别为 24.6%、28.2%、31.6%和 15.6%,其中 A+T 含量(52.8%)明显高于 G+C 含量(47.2%)。
43 尾野生群体所含序列共有 23 个变异位点, 其中简约信息位点 16 个, 单突变位点 7 个。43 尾繁育群体所含序列共有 10 个变异位点, 其中简约信息位点9 个, 单突变位点 1 个。
2.2 遗传多样性
对 86 尾个体的控制区片段和 Cytb 基因片段进行 同 质 性 检 验 , 结 果 显 示 两 者 之 间 是 一 致 的(P=0.13)。基于 1871 bp 线粒体基因联合片段分析秦岭细鳞鲑野生群体与繁育群体的遗传多样性信息见表 1, 由表 1 中可以看出 86 尾个体共检测出 34 个单倍型, 显示出较高的单倍型多样性和较低的核苷酸多样性(h=0.919±0.022; π=0.00321±0.00077)。繁育群体与野生群体相比较, 繁育群体的单倍型多样性和核苷酸多样性(h=0.917±0.035; π=0.00349±0.00083)都 高 于 野 生 群 体 (h=0.907±0.026; π=0.00287±0.00074)。在秦岭细鳞鲑繁育群体 43 尾个体中共检测出 24 个单倍型, 野生群体 43 尾个体中共检测出18 个单倍型, 其中两群体共享 8 个单倍型, 占总单倍型个数的 23.5%。【表1】

2.3 群体遗传分化
通过计算两群体间的遗传距离得出, 繁育群体和野生群体内的遗传距离分别为 0.004 和 0.003,繁育群体和野生群体之间的遗传距离为 0.003, 略低于繁育群体内的遗传距离, 与野生群体的遗传距离相近。分子变异(AMOVA)结果显示(表 2), 秦岭 细 鳞 鲑 群 体 内 的 存 在 较 高 的 遗 传 变 异 , 占98.37%, 而群体间的变异仅占 1.63%, 表明分子之间遗传变异主要来自于群体内的个体之间。两群体之间的 Fst=0.01631(P=0.1075), Nm=30.16, 也显示出秦岭细鳞鲑繁育群体和养殖群体之间的遗传分化不显着。【表2】

2.4 系统发育树
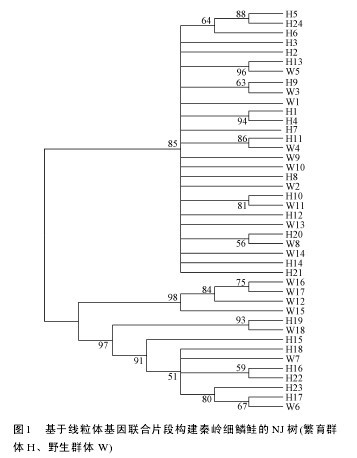
由秦岭细鳞鲑繁育群体24个单倍型和野生群体18个单倍型构建的系统发育树(图1)可以看出, 各野生群体和繁育群体的个体均为形成单系群, 两者之间互有交叉, 表明两群体的遗传相似性较高, 亲缘关系较近。由 network 软件形成秦岭细鳞鲑群体34个单倍型的简约网络图(图2)也显示出发散状态势,两群体未形成各自的单系群。单倍型 H3位于网络图的中心, 且享有该单倍型的个体数量在种群中所占的比例明显高于其他单倍型所占比例, 表明H3可能是原始单倍型。【图1-2】
3 讨论
3.1 群体的遗传多样性
物种遗传多样性的高低是评判物种能否长期存在的依据, 核苷酸多样性是衡量一个种群 mtDNA的遗传多样性的重要指标, 表示各种线粒体 DNA单倍型在群体中所占的比例。由实验结果可知,秦岭细鳞鲑 86 尾个体的单倍型多样性(h=0.919±0.022) 较高 , 但核苷酸多样性 (π=0.00321±0.00077)较低, 在银鲳、鲇鱼和松江鲈等鱼类中也存在这种现象, 这种单倍型多样性较高核苷酸多样性较低的现象表明秦岭细鳞鲑群体可能是由一个较小种群经历奠基者效应或瓶颈效应后迅速扩张, 随着个体数量的增多, 导致单倍型多样性提高, 但缺少足够的时间来积累核苷酸序列的变异, 这种推测与秦岭细鳞鲑的起源历程相一致。但本文中得到的秦岭细鳞鲑单倍型多样性较高的结论与夏颖哲等分析黄河地区细鳞鲑的单倍型多样性(0.622)的结果不符, 这可能由于夏颖哲等采集样本数量(n=10)较少影响了遗传多样性的正确评估。
在本研究中繁育群体的单倍型多样性和核苷酸遗传多样性(h=0.917±0.035; π=0.00349±0.00083)都高于野生群体(h=0.907±0.026; π=0.00287±0.00074),与原居林等对黑河种群和 湑水河种群的遗传多样性进行分析得出秦岭细鳞鲑繁殖群体后代遗传多样性降低的结论不相符, 这可能是由于本实验繁殖群体亲本来源分布较广, 且每年都会补充一定数量的亲本, 有效避免了近亲繁殖和瓶颈效应, 保持了秦岭细鳞鲑人工繁育群体的遗传多样性。而原居林等所采集样本为 20 世纪 30 年代引入到 湑水河中进行驯养的群体, 在长期的人工繁殖过程中, 由于基础繁育群体数量较少、近亲繁殖和遗传漂变等因素,使得养殖群体丧失一些多态等位基因造成遗传多样性水平降低。
3.2 群体的遗传结构分析
群体间的遗传距离和遗传分化指数是评价一个群体多态程度的重要指标。种群间遗传距离 D 值的范围是 0—0.05, 亚种间的遗传距离 D 值范围是0.02—0.2。本研究结果表明, 秦岭细鳞鲑 2 个种群内的遗传距离为(0.003—0.004), 繁育群体与野生群体之间的遗传距离为 0.003, 表明秦岭细鳞鲑繁育群体与野生群体之间的遗传变异程度低, 分化程度不明显。秦岭细鳞鲑群体单倍型之间的系统发育树和网络进化图分析结果表明, 繁育群体和野生群体之间无明显的遗传分化, 两者的遗传相似程度较高。根据 Wright 关于遗传分化程度的理论认为 Fst值在 0—0.05 表示低度遗传分化, 秦岭细鳞鲑繁育群体和野生群体之间的遗传分化指数(Fst) 为0.01631, 表明两群体之间存在低度遗传分化, 分子变异(AMOVA)的分析结果显示, 98.37%的遗传变异来源与群体内, 仅有 1.63%的变异来源与两群体之间。由基因流 Nm值来看, 基因流是指基因从一个群体迁移至另一个群体时产生的基因流动, 一般认为Nm>1, 表明群体间的基因流水平较高, 遗传分化较小。秦岭细鳞鲑野生群体与繁育群体之间的基因流Nm值达到 30.16, 远大于 1, 也同样表明了两群体之间的遗传分化较小, 这可能是由于两者之间共享单倍型较多有关。秦岭细鳞鲑资源量的减少可以通过人工增殖手段来恢复, 但如果其种群遗传多样性的降低却难以弥补。目前秦岭细鳞鲑的繁育群体主要用于增殖放流, 保持其较高的种群遗传多样性具有重要意义。
参考文献:
[1] Qin S Z, Wang S A. Studies on the subspecies ofBrachymystax lenok [J]. Salmon Fishery, 1989, 2(1): 52—61[秦树臻, 王所安. 细鳞鱼亚种问题的研究. 鲑鳟渔业,1989, 2(1): 52—61]
[2] Li S Z. Discussed the geographical distribution of Chinasalmonidae fish [J]. Chinese Journal of Zoology, 1984, (1):34—37 [李思忠. 中国鲑科鱼类地理分布的探讨. 动物学杂志, 1984, (1): 34—37]
[3] Li S Z. On a new subspecies of fresh-water trout,Brachymystax lenok tsinlingensis, from taipaishan, Shanxi,China [J]. Acta Zootaxonomica Sinica, 1966, 3(1): 92—94[李思忠. 陕西太白山细鳞鲑的一新亚种. 动物分类学报,1966, 3(1): 92—94]
0、引言制卵形鲳鲹属于鲈形目(Perciformes)、鲈亚目(Percoide)、鲹科(Carangidae),鲳鲹亚科(Trachinotinae)、鲳鲹属(Trachinotus),俗称黄腊鲳、短鳍鲳鲹、金鲳、红三、红沙等.主要分布于印度洋、印度尼西亚、澳洲、中国南海、东海、黄海、渤海、日本...
海藻场生态系统是典型近岸浅海生态系统之一,利用大型海藻吸收、固定,并转移海水中的碳、氮、磷等生源要素,减轻水域富营养化,同时形成多种海洋生物生长、栖息的局部稳定的小生境,达到增加生物多样性,修复渔业资源的目的[1-9]。近年来随着海洋环境污染...
近年来,为保护和合理开发海洋渔业资源,加快渔业调结构、转方式,遏制近岸海域生态环境不断恶化趋势,促进渔民就业、增收,河北省深入贯彻农业农村部的部署,坚持科学规划、示范带动、规范监管,大力推进人工鱼礁和海洋牧场建设,取得显着成效。...
泥鳅属于鲤形目、花鳅亚科、泥鳅属,广西各地的河川、沟渠、水田等都有分布。其肉质鲜美、营养丰富,是人们喜食的水产品之一,市场价格长年稳中有升。近年来,由于人为过度捕捞和生态环境破坏等原因,广西的泥鳅资源急剧下降。鉴于广西泥鳅资源濒危和经济价...
厚壳贻贝(Mytilus coruscus)隶属软体动物门(Mollusca)双壳纲(Bivalvia)贻贝属(Mytilus),俗称“淡菜”,是我国重要的经济贝类之一,主要分布于我国渤海、黄海、东海沿岸以及澎湖。...
受自然和人为因素的干扰, 淡水鱼类的产卵场及栖息地遭到严重破坏, 渔业资源日益衰退。近年来, 人工鱼巢被广泛应用于淡水流域及河口渔业资源及其生境的修复和管理, 为保护淡水流域生物多样性和制定渔业管理措施等方面提供了一条有效途径。...