摘 要: 蛋白质羰基化作为一种不可逆的翻译后修饰,与诸多疾病和衰老密切相关。有关蛋白质羰基化的各项研究受制于其低丰度、低电离效率及化学相对不稳定性而发展较慢。基于质谱的蛋白质组学分析技术的进步,使蛋白质羰基化的规模化研究成为可能,进而为蛋白质羰基化的相关调控通路研究提供了数据支撑。该综述介绍了蛋白质羰基化的概念、途径、检测方式,并重点介绍了蛋白组学技术应用于蛋白质羰基化分析的进展。
关键词: 氧化应激; 蛋白质羰基化; 蛋白质组学;
Abstract: Protein carbonylation,an irreversible post-translational modification,is closely related to many diseases and aging.The analysis of protein carbonylation is a challenge due to its low abundance,low ionization efficiency and relative chemical instability.Advances in mass spectrometry-based proteomics have made it possible to conduct large-scale studies on protein carbonylation,providing in turn the data support for the study of related regulatory pathways of protein carbonylation.The concept,pathways and analysis methods of protein carbonylation are presented in this review,and the advances in proteomic analysis of protein carbonylation are highlighted.
Keyword: oxidative stress; protein carbonylation; proteomics;
1、 氧化应激与蛋白质羰基化
绝大多数真核生物的生命活动都离不开氧气,氧气与高等生物体的能量代谢等过程密切相关[1,2]。氧气分子在代谢中间体、酶和辐射作用下产生活性氧(Reactive oxygen species,ROS),其在正常生理条件下对细胞代谢的调控发挥着至关重要的作用[3]。然而,当产生的ROS超过生命体内源性抗氧化防御的缓冲能力,即氧化剂和抗氧化剂之间的不平衡被打破时,将导致氧化还原信号与控制机制的破坏和/或分子损伤[4],产生氧化应激效应(Oxidative stress,OS),进而导致脂质、DNA/RNA和蛋白质等重要生命体活动承担者的功能受损。当持续氧化应激产生时,将对蛋白质、DNA和RNA等生物分子造成不可逆损伤,并造成蛋白酶体和溶酶体降解氧化损伤蛋白质的能力降低,使细胞活力降低,甚至造成细胞死亡[5,6]。氧化应激异常与衰老过程以及多种疾病的发生密切相关,包括动脉粥样硬化、恶性肿瘤、阿尔茨海默病和帕金森病等神经退行性疾病[7]。
蛋白质羰基化(Protein carbonylation,PCO)是指蛋白质在氧化应激状态下产生醛、酮或内酰胺等活性羰基官能团[8]。PCO是危害性最大的蛋白质翻译后修饰之一,可造成蛋白质结构的不稳定和功能的不可逆损伤[9]。有关蛋白质羰基化的研究始于20世纪80年代初,由美国国立卫生研究院(National Institutes of Health,NIH)的Stadtman实验室开创[10]。蛋白质羰基化被视为氧化应激的生物标志物,在细胞、组织和器官衰老中的作用受到特别关注[11]。Levine和Stadtman提出蛋白质羰基化水平随着年龄的增长而增加,以此支持细胞或生物体水平上衰老的自由基(氧化应激)理论[12,13,14,15],故蛋白质羰基化是与衰老相关的重要候选生物标志物。同时,蛋白质羰基化会调控蛋白功能,进而影响相关信号转导网络,造成疾病相关蛋白质功能的不可逆损伤[16]。因此,羰基化对于蛋白质功能的发挥具有至关重要的调节作用,实现蛋白质羰基化的表征,可以提供氧化应激相关蛋白的作用网络,为相关疾病的分子机制研究提供重要信息[17]。

2 、蛋白质羰基化途径
蛋白质羰基化存在超过35种形式[18],按照其产生方式主要分为以下4类[10](图1)。
图1 蛋白质羰基化的主要途径
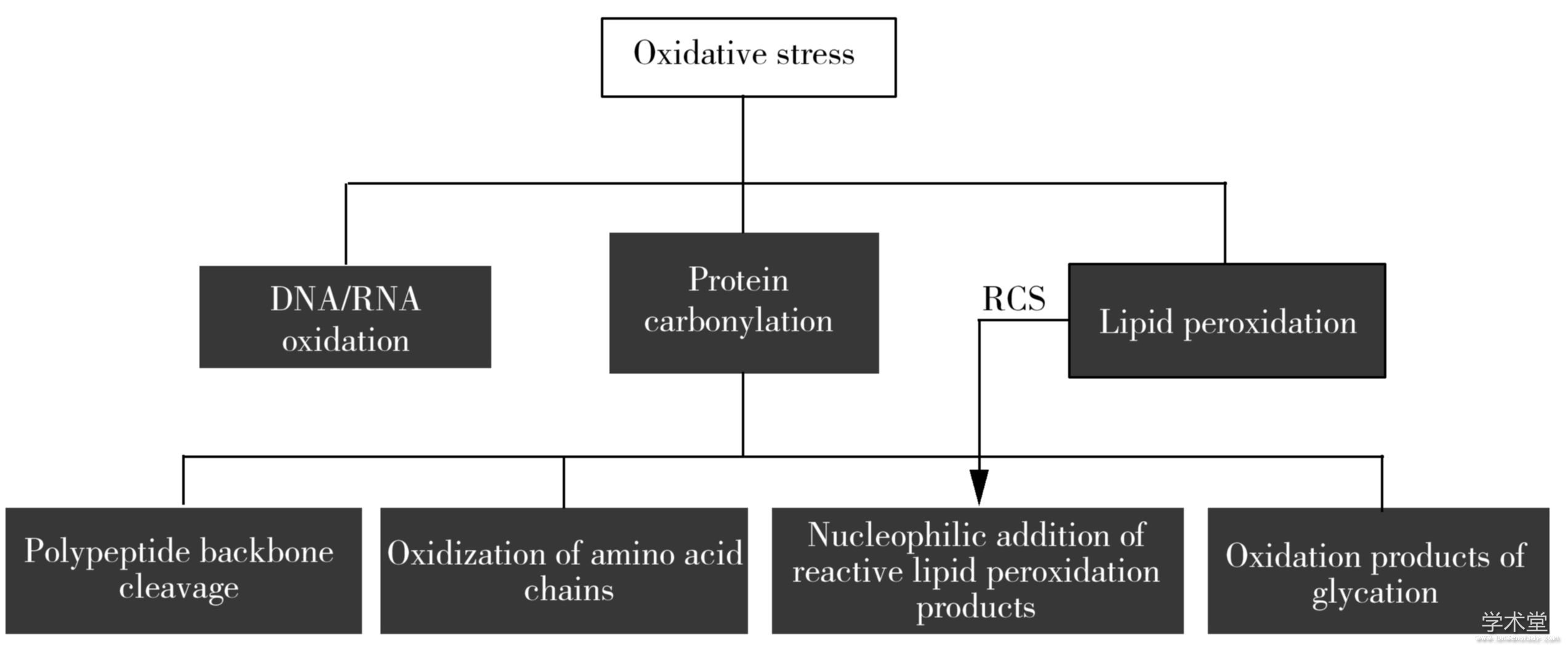
Fig.1 Main protein carbonylation pathways
2.1、 蛋白质、多肽主链骨架的裂解
蛋白质的多肽骨架中氨基酸的α-碳原子可被ROS攻击,形成烷氧基自由基,随后以α-酰胺化或二酰胺途径裂解。前者的键断裂发生在自由基起始位点的N-末端一侧,使来自蛋白质N-末端部分的多肽片段带有C-末端酰胺,而来自蛋白质C-末端部分的多肽N-末端具有N-α-酮酰基;后者的骨架裂解由α-碳起始位点的C-末端侧的键断裂引起,使来自蛋白质N-末端部分的新多肽的C-末端形成二酰胺,而来自蛋白质的C-末端部分的多肽N-末端产生异氰酸酯基团。
2.2、 蛋白质氨基酸侧链的氧化
ROS可通过直接攻击苏氨酸、赖氨酸、精氨酸、脯氨酸的侧链,引入活性羰基。以赖氨酸为例,羟基自由基在赖氨酸侧链的C6位置夺取氢,形成以碳为中心的自由基。过渡金属离子接受碳自由基的孤电子对后,产生赖氨酸亚胺,随后通过释放铵离子自发水解成氨基己二酸半醛(AAS)。类似地,精氨酸氧化成谷氨酸半醛(GGS),苏氨酸氧化成2-氨基-3-酮丁酸,脯氨酸氧化得到GGS和2-吡咯烷酮。
2.3、 脂质过氧化产物对蛋白活性位点的加成
ROS对脂质的进攻引起脂质过氧化(Lipid peroxidation),产生α,β-不饱和醛、二醛和酮醛等被称为活性羰基物质(Reactive carbonyl spices,RCS)的代谢产物(又称Lipid-derived electrophiles,LDEs,即脂质衍生亲电试剂),如丙烯醛(Acrolein,ACR)、丙二醛(MDA)、4-羟基-2-壬烯醛(4-Hydroxy-2-nonenal,HNE)等。这些活性羰基物质可以通过迈克尔加成或生成席夫碱的形式加合到亲核氨基酸(即半胱氨酸、组氨酸和赖氨酸)上引入活性羰基。研究这类共价修饰作用不仅能够发现亲电性化学信号的调控机制,而且对目前药物化学领域热门的靶向共价抑制剂开发有着重要参考价值。
2.4、 糖化的氧化产物
氨基酸和还原糖之间发生被称为美拉德反应的非酶促反应,在一系列化学重排之后,可以在蛋白质侧链引入活性羰基。除此之外,糖酵解中间体降解产生的小分子醛类,如乙二醛(MG)、甲基乙二醛(MGO)和3-脱氧葡糖醛酮(3-DG)也可以加合在蛋白质上从而引入活性羰基。
3、 蛋白质羰基化的富集检测方法
目前,蛋白质羰基化的鉴定和定量技术主要分为以下3类:(1)分光光度法和色谱法测定总蛋白质羰基含量;(2)生物化学和免疫学技术,如免疫印迹和ELISA(Enzyme-linked immunosorbent assay ,酶联免疫吸附测定法),提供有关修饰蛋白和羰基化水平的全局性信息;(3)基于质谱(MS)的羰基化检测技术,用于鉴定蛋白质的修饰位点以及蛋白质结合的羰基化合物的相对定量[19]。质谱以外的其他方法虽然能够高灵敏度检测氧化修饰蛋白质,却不能提供修饰蛋白质特性以及修饰本身的化学性质和位点等信息。基于质谱的蛋白质组学的进步,使蛋白质羰基化的规模化研究成为可能,进而为蛋白质羰基化的相关调控通路研究提供了数据支撑[19]。目前采用的蛋白质组学方法主要分为凝胶电泳与非凝胶电泳两大体系(图2)。
图2 蛋白质羰基化蛋白组学分析方法
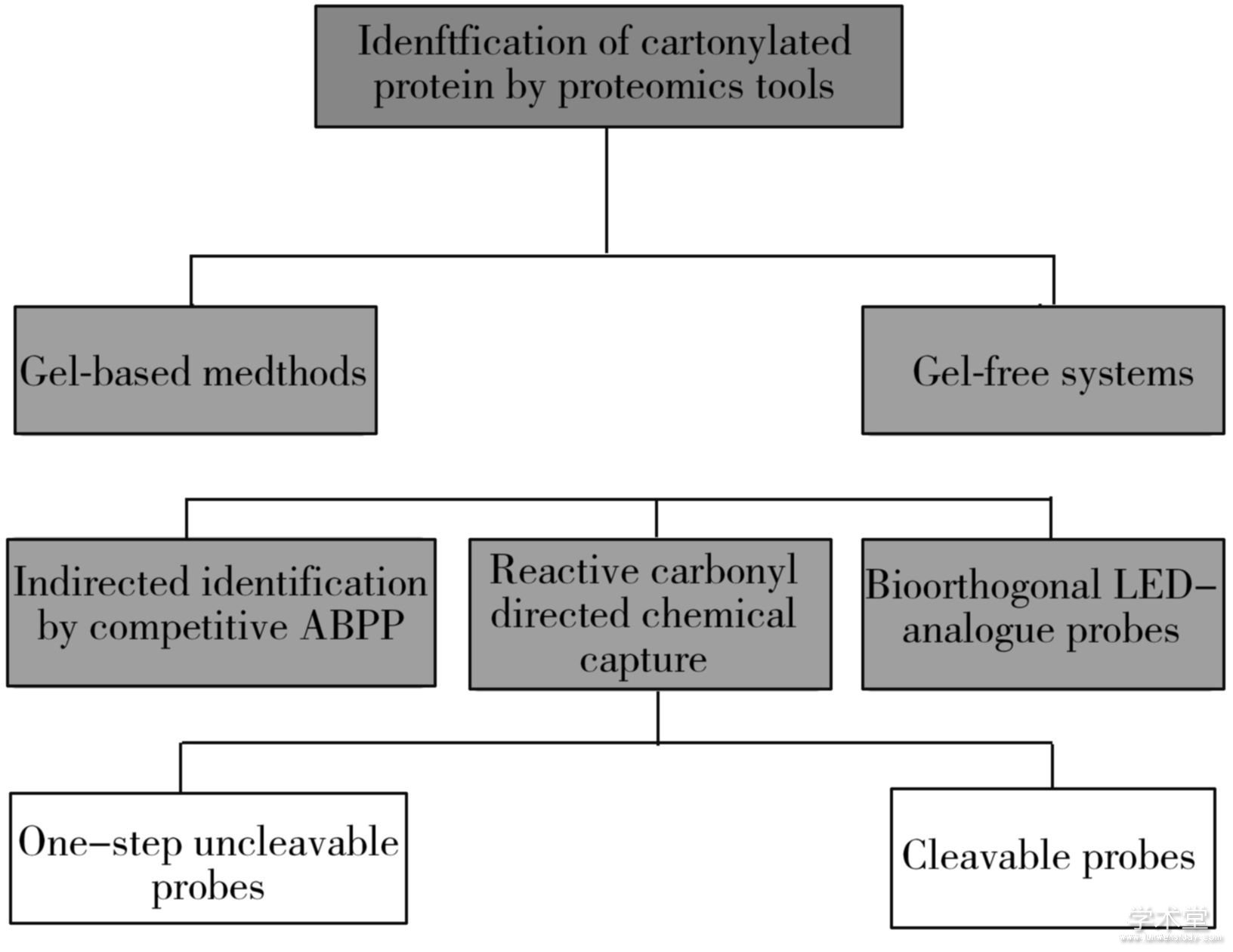
Fig.2 Proteomics analysis methods of carbonylated protein
3.1、 凝胶电泳体系
二维(2D)凝胶电泳已被广泛用于复杂样品中羰基化蛋白质的鉴定。由于相对丰度低、化学稳定性差、缺乏特定的物理化学性质(如吸收或荧光),羰基化蛋白质通常无法直接检测,其检测和定量需要使用特定的化学探针进行衍生化[15]。
2,4-二硝基苯肼(2,4-Dintrophenylhydrazide,DNPH)可与活性羰基反应,形成稳定的2,4-二硝基苯腙,其吸收光谱在365~375 nm处具有特征吸收峰。Levine等[20]首先使用DNPH,通过DNP(2,4-二硝基苯基)基团在360 nm处的吸光度来测定羰基化程度。这一思路被发展为“oxyblot”技术应用于蛋白质印迹法,即先用DNP衍生羰基化样品,再使用抗DNP抗体进行检测[21,22],还可以通过质谱法进行凝胶内消化后对羰基化修饰位点进行鉴定。
酰肼基团可以与活性羰基反应形成腙加合物,其含有的不稳定亚胺键,可被氰基硼氢化物特异性还原。此外,酰肼还可以引入多种官能团(如生物素、洋地黄毒苷和与酰肼偶联的各种荧光化合物等)后再与羰基反应,如可以用洋地黄毒苷-酰肼衍生化羰基并以抗洋地黄毒苷抗体进行检测,或用生物素-酰肼衍生化并用荧光素标记的亲和素检测。荧光素缩氨基硫脲和荧光素酰肼首先由Stadtman等[23]用于在一维凝胶中检测羰基;Regnier等[24]采用生物素化和亲和素-异硫氰酸荧光素(Biotinylation and avidin-fluorescein isothiocyanate,FITC)亲和染色法,建立了二维电泳中羰基化蛋白的检测方法,在用过氧化氢刺激后的酵母蛋白质组中鉴定出20种羰基化蛋白质。2D凝胶电泳可以在各种生物条件下用于差分氧化蛋白质的鉴定,但其灵敏度较低,不易检测低丰度的蛋白质,操作费力且不允许高通量检测[25]。
3.2、 非凝胶电泳体系
质谱法是蛋白质组学中用于鉴定蛋白质,尤其是PTM(Post-translational modification,翻译后修饰)表征的最通用技术。使用质谱法的无凝胶方法已经成为研究蛋白质羰基化的最有力技术。蛋白质羰基化的各项研究受制于其低丰度、低电离效率、可能产物的异质性以及与分析期间存在的其他化合物的反应性。因此在质谱分析之前,分离羰基化肽和蛋白质的富集策略通常是必需的[26]。
3.2.1、 基于竞争性反应策略对半胱氨酸羰基化修饰的间接鉴定
脂质过氧化产物会特异性修饰蛋白质的半胱氨酸等活性位点,进而影响蛋白质的功能。可以通过硫醇反应性化学探针标记未被修饰的半胱氨酸活性位点,结合蛋白质定量组学方法,间接实现相关羰基化蛋白质底物及作用位点的鉴定。Cravatt等[27]基于竞争性isoTOP-ABPP(Isotopic tandem orthogonal proteolysis-activity-based protein profiling,同位素串联正交蛋白水解-基于活性的蛋白谱分析)策略,使用LDE(引入活性羰基基团)或DMSO(Dimethylsulfoxide,二甲基亚砜)处理蛋白质组,再用硫醇反应性碘乙酰胺(Iodoacetamide,IAM)-炔探针标记,通过CuI催化的叠氮化物-炔烃环加成反应(点击化学)缀合上可切割的、同位素标记的生物素标签,以链霉亲和素珠富集,珠上酶解得到探针标记肽段,并用LC-MS/MS鉴定和定量。该策略能够通过半胱氨酸反应性碘乙酰胺-炔(IA)探针定量蛋白质组中LDE修饰的1000多个半胱氨酸位点。由于对半胱氨酸的LDE修饰可以竞争性地阻断其IA-炔烃标记,DMSO处理的(轻)与LDE处理的(重)信号之间的定量比率准确地反映了对半胱氨酸LDE修饰的程度。该方法基于LDE和碘代乙酰胺探针对半胱氨酸活性位点的竞争作用,有效实现了LDE相关底物的筛选,并获取了相应的底物位点信息。然而,该方法只能间接获取LDE对蛋白质中半胱氨酸残基的修饰位点信息,无法获取LDE与组氨酸和赖氨酸修饰位点信息。
3.2.2、 通过生物正交LDE-类似物探针对特定羰基化类型进行检测鉴定
与基于竞争作用的间接羰基化检测方法相比,实现修饰位点的直接鉴定具有更高的灵敏度,并能有效减少假阳性干扰。Marnett等[28]设计合成了带有叠氮或炔基的4-羟基-2-壬烯醛(HNE)类似物,并将其加入到RKO细胞(Human colon adenocarcinomacells,人结肠腺癌细胞)中,使RKO细胞中靶蛋白产生羰基化修饰,再加入相对应的接入炔基或叠氮基团的生物素,利用点击化学反应获得生物素化靶蛋白,最后用链霉亲和素珠亲和纯化。针对链霉亲和素结合非特异性蛋白质以及生物素标签影响质谱检测等问题,Porter等[29]提出了亲和纯化-光致分离策略,开发了一种可由光裂解的叠氮-生物素标签,可以实现链霉亲和素捕集的HNE-炔烃探针加合蛋白质或肽段的特异性释放,并用脂质衍生亲电试剂HNE和4-氧代-2-壬烯醛(4-oxo-2-Nonenal,ONE)的炔基类似物进行了一系列定量加合物分析实验[30]。在此基础上,Liebler等[31]开发了一种以位点为中心的定量化学蛋白质组学方法,其核心在于在光可切割叠氮基生物素试剂中接入轻重同位素标记,有效提高了鉴定位点的可信度,实现了对约400个HNE修饰位点的大规模、原位、位点特异性鉴定和定量。
生物正交法局限于只能鉴定特定类型的羰基化修饰,不能捕获蛋白质组中所有类型的羰基化修饰蛋白。合成特定LDE类似物探针成本较高、工作量大,此外,在LDE上接入炔基或叠氮化物可能会改变它们的物理和化学性质,反过来又会影响其与一些天然靶蛋白的相互作用。
3.2.3、 靶向活性羰基的化学捕集策略
(1)“一体化”生物素探针
利用某些化学基团可以快速动力学和高特异性结合活性羰基形成稳定的共价加合物的性质,将其与生物素连接为探针,再使用亲和素进行捕获,可以实现衍生化羰基化蛋白/肽段的高效富集。其中使用最广泛的是生物素酰肼(Biotin hydrazide,BHZ)探针[32]。
Yang等[33]将BHZ与样品中羰基化蛋白反应生成亚胺,并用氰基硼氢化钠特异性还原亚胺稳定产物,用链霉亲和素珠捕集生物素化蛋白,珠上洗涤、还原、烷基化封闭、酶解后,取酶解上清液进行LC-MS/MS检测。该研究在单次LC-MS/MS实验中能够鉴定老年小鼠中的至少100种羰基化蛋白质。
亲和素可在广泛的pH值范围内以非常高的亲和力结合生物素化多肽,使携带位点信息的肽段难以从珠上释放,因而无法得到所对应蛋白质中羰基化修饰的具体位点。无论是为了更深入地理解氧化应激导致活组织损伤和功能丧失的原因和机制,还是为了提高鉴定结果置信度,鉴定蛋白质序列中的氧化位点都是必要的。鉴于此,Regnier等[34]采用亲和能力较弱的固定化单体亲和素构建层析柱,柱上捕获的生物素化蛋白质使用2 mmol/L生物素或0.1 mol/L甘氨酸[18]洗脱下来,从而获得含有羰基化位点的肽段。Regnier等[35]在亲和富集后通过反相液相色谱(Reversed-phase liquid chromatography,RPLC)在C8柱上进一步分离分析羰基化蛋白质,并首次报道了基于蛋白质组学的人血浆中氧化蛋白的鉴定和表征[36]。
内源生物素化蛋白质,以及通过疏水和(或)静电相互作用与亲和素非特异性结合的蛋白质和肽段,是生物素探针法鉴定羰基化蛋白的主要干扰源[19]。为了有效鉴定特异性纯化的蛋白质组分并过滤掉非特异性背景蛋白质,Griffin等[37]对亲和纯化后的蛋白进行稳定同位素标记。该方法结合了蛋白质羰基的生物素酰肼标记、亲和素亲和层析、多重iTRAQ(Isobaric tag for relative absolute quantitation,同位素标记相对和绝对定量)试剂稳定同位素标记等方法,并尝试在线性离子阱质谱仪上使用脉冲碰撞解离(PQD)操作,定量富集得到200多种羰基化蛋白。与之类似的同位素标记方法已被广泛应用于人体血浆[38]、鼠血浆[39]、RCS修饰蛋白质[40,41,42]中羰基化的鉴定。
由于羟胺与活性羰基生成产物的肟键(Keq>108 M-1)比与酰肼反应生成产物的腙键(Keq约为104~106 M-1)稳定得多[43],ARP(Aldehyde-reactive probe,醛反应性探针)作为最开始被用于DNA修饰检测的生物素化羟胺衍生物,因不需要额外的还原步骤,而成为广泛应用的一种酰肼生物素替代品[44,45,46,47]。
基于酰肼或羟胺生物素化探针的富集方法,能够实现多种内源性羰基化蛋白质的高效捕获,并通过蛋白质组学技术实现多种类型内源性羰基化的直接鉴定。然而,由于缺乏有效的脱除手段,庞大的生物素基团限制了MS分析过程中羰基化肽序列的碎裂和鉴定效率,并且冗长的生物素侧链的片段化使质谱解析变得更加复杂。针对以上“一体式”探针面临修饰位点鉴定效率低的问题,实现羰基化修饰肽段可控释放成为研究的热点。
(2)“分体式”可控释放探针
Griffin等[48]提出一种基于固相酰肼(SPH)试剂开发的无标记策略,通过形成共价可逆的腙键,可在酰肼包被的玻璃珠上捕获HNE修饰肽,并在较高温度(60 ℃)和低pH值(10%三氟乙酸或甲酸)下洗脱释放。类似地,基于Affi-gel酰肼珠的共价色谱法在自下而上的蛋白质组学方法中得到很好的应用,譬如该酰肼珠富集法也被应用于糖基化修饰的研究[49]。Maier等[42]将用于相对定量的d0/d4-琥珀酸酐-肽胺标记方法和用于捕获脂质过氧化产物-肽缀合物的Affi-gel酰肼珠富集方法结合,实现了羰基化蛋白/肽段的富集和定量分析。Prokai等[40]通过还原烷基化对HNE-修饰的肽进行二甲基标记,使用固相酰肼化学捕集羰基化肽段,再由酸催化释放回收。这种以修饰为重点的定量有助于表征蛋白质功能的伴随变化,并且可提供有关分子信号传导机制的重要信息,加深对与氧化应激相关的细胞过程的理解,但是该方法酸释放的条件比较苛刻,且仍然面临释放不完全的问题。
GPR(Girard preagent:1-(2-hydrazino-2-oxoethyl) pyridinium chloride,1-(2-肼基-2-氧代乙基)吡啶鎓氯化物)是最初开发用于衍生和溶解类固醇的系列试剂的一部分[50],其酰肼基团易与羰基反应,而季铵基团则可向氧化蛋白质和肽添加正电荷。Regnier等[51]将GRP与羰基化蛋白质反应,还原、酶解后用SCX(Strong cation exchange,强阳离子交换)色谱法富集。接着,两种同位素形式的GPR试剂被用于量化和表征酵母提取物中蛋白质羰基化位点[52]。与此有异曲同工之妙的是,Lu等[53]开发了基于氟衍生化和FSPE(Fluorous solidphase extraction,氟固相萃取)的新方法,将标记的肽与固相氟特异性结合用于HNE修饰肽富集。该方法的优点是氟标签可以在MS分析期间增强HNE修饰肽的强度,从而有助于鉴定HNE-修饰的肽,并促进氟衍生化肽段的MS/MS片段化。
AminoxyTMT(Aminoxy tandem mass tag,羟胺串联质量标签)是串联质量标签(Tandem mass tag,TMT)同量异位标记试剂的衍生物,Griffin等[54]利用其羟胺基官能团共价标记蛋白质中的活性羰基并实现了富集。Lu等[55]提出了六重同量异位标记亲和纯化(SiLAP)策略,使用aminoxyTMT标记试剂和抗TMT抗体树脂同时实现HNE修饰蛋白的选择性富集和特异性位点的准确定量,量化了2 257种独特的HNE修饰肽和1 121种HNE修饰蛋白。
Wang等[56]合成了生物正交的含炔基酰肼探针(HZyne)和羟胺衍生探针(Ayyne),评估了它们捕获HNE修饰的蛋白质和残留位点以进行MS鉴定的能力,通过基于二甲基标记的RD-ABPP(Reductive dimethylation-ABPP,还原型二甲基标记-ABPP)策略,定量了4 000多个HNE修饰蛋白细胞裂解物;通过TOP-ABPP(Tandem orthogonal proteolysis-ABPP,串联正交蛋白水解-ABPP)策略鉴定了约400个高可信度的HNE加合位点。Yang等[57]使用前述HZyne探针,富集并分析了肽骨架碎裂形成的全局和位点特异性的亲电子蛋白质降解物,并鉴定到N-甲酰基多肽这一新的羰基化产物。值得注意的是,Yang提及了将定量化学蛋白质组学与开放式搜索工具相结合的潜在应用前景。Wang等[58]开发了一种苯胺衍生的探针,并结合ABPP策略分析铁死亡(ferroptosis)中的羰基化蛋白,鉴定到400多种内源性羰基化修饰蛋白质和20余个残基位点。Wang等[59]将苯胺探针用于鉴定丙烯醛(ACR)修饰位点,鉴定了2 300余种蛋白质和500余个由丙烯醛靶向的半胱氨酸位点。
这些基于可控释放思路开发的新型探针,极大地提高了羰基化蛋白及其位点的鉴定数量和质量,推动了蛋白质羰基化的研究进程,但此类方法操作步骤较为繁琐,肽段携带标签依然较大,因此尚需开发新的更好的方法解决此类问题。近来,Aye等在T-REX[60](Targetable reactive electrophiles and oxidants,靶向活性亲电物质和活性氧)基础上开发出G-REX[61]技术,即在细胞内异位表达HaloTag蛋白,该蛋白可特异性结合光笼装RES(Reactive electrophilic specie,活性亲电物质,即上文所述RCS)前体。在低能量光照射下,体内可控释放RES,如5 min内细胞最多可释放出5 μmol/L HNE-炔烃,大大减轻了此前研究体外大剂量暴露相关的毒性/脱靶效应,进而提高了所鉴定到羰基化位点的可靠性。可以预见G-REX在研究氧化应激,特别是RES相关的蛋白质羰基化方面的巨大潜力。目前G-REX诱导得到的HNE修饰蛋白依然使用链霉亲和素点击/生物素亲和富集的方法。如前文所述,这无疑限制了该技术的更有效应用。我们认为应该利用新的化学反应,开发新型可逆生物正交探针,其应满足:①高效特异地与羰基化蛋白结合,最好与活性羰基结合;②能够被有效地富集出来;③与羰基化蛋白的结合应该是可逆的,以方便地释放富集蛋白/肽段,获取羰基化位点信息;④如果可能,该探针应该与G-REX技术兼容。G-ERX技术与新型探针富集技术的配套使用,必将有力推动蛋白质羰基化研究的进程。
4 、结 语
化学生物学技术的快速发展为蛋白质羰基化的富集提供了更多的有力工具,而质谱技术的发展则为蛋白质羰基化提供了检测手段,两者的结合极大地推动了蛋白质羰基化的研究进展。然而,蛋白质羰基化具有高度动态性,并且存在数目繁多的修饰形式,实现不同类型羰基化的同时检测尚需要富集、检测技术的进一步完善,并且需要借助相关生物信息学技术的发展。随着相关技术的进一步发展,蛋白质羰基化的研究必将大为加速,进而为阐明氧化应激的机制提供技术支撑。
参考文献
[1] Davies K J A.Biochem.Soc.Symp.,1995,61:1-31.
[2] Davies K J A.Arch.Biochem.Biophys.,2016,595(1):28-32.
[3] Bachi A,Dalle-Donne I,Scaloni A.Chem.Rev.,2013,113(1):596-698.
[4] Fink G.Encyclopedia of Stress.2nd Ed.Amsterdam:Elsevier,2007:45-48.
[5] Griesser E,Vemula V,Raulien N,Wagner U,Reeg S,Grune T,Fedorova M.Redox Biol.,2017,11:438-455.
[6] Mullineaux P M,Baker N R.Plant Physiol.,2010,154(2):521-525.
[7] Dalle-Donne I,Scaloni A,Butterfield D A.Redox Proteomics:From Protein Modifications to Cellular Dysfunction and Diseases.United States of America:Wiley-Interscience,2006:123-168.
[8] Colombo G,Clerici M,Garavaglia M E,Giustarini D,Rossi R,Milzani A,Dalle-Donne I.J.Chromatogr.B,2016,1019:178-190.
[9] Bollineniab R C,Fedorovaab M,Hoffmann R.Analyst,2013,138:5081-5088.
[10] Ros J.Protein Carbonylation:Principles,Analysis,and Biological Implications.1st Edition.Hoboken,NJ:John Wiley & Sons,Inc.,2017:27-48.
[11] Dalle-Donne I,Rossi R,Giustarini D,Milzani A,Colombo R.Clin.Chim.Acta,2003,329(1/2):23-38.
[12] Stadtman E R.Science,1992,257:1220-1224.
[13] Stadtman E R,Berlett B S.Drug Metabol.Rev.,1998,30(2):225-243.
[14] Stadtman E R.Free Radical Res.,2006,40(12):1250-1258.
[15] Levine R L,Stadtman E R.Exp.Gerontol.,2001,36(9):1495-1502.
[16] Yan L J,Forster M J.J.Chromatogr.B,2011,879(17/18):1308-1315.
[17] Fritz K S,Petersen D R.Chem.Res.Toxicol.,2011,24(9):1411-1419.
[18] Madian A G,Regnier F E.J.Proteome Res.,2010,9(8):3766-3780.
[19] Fedorova M,Bollineni R C,Hoffmann R.Mass Spectrom.Rev.,2014,33:79-97.
[20] Levine R L,Garland D,Oliver C N,Amici A,Climent I,Lenz A-G,Ahn B-W,Shaltiel S,Stadtman E R.Methods Enzymol.,1990,186 :464-478.
[21] Goto S,Nakamura A,Radak Z,Nakamoto H,Takahashi R,Yasuda K,Sakurai Y,Ishii N.Mech.Age.Dev.,1999,(107):245-253.
[22] Shacter E,Williams J A,Lim M,Levine R L.Free Radical Biol.Med.,1994,17:429-437.
[23] Rhee B A S G,Stadtman E R.Anal.Biochem.,1987,161(2):245-257.
[24] Yoo B S,Regnier F E.Electrophoresis,2004,25:1334-1341.
[25] Tamarit E,Ros J.Mass Spectrom.Rev.,2014,33:21-48.
[26] Bollineni R C,Fedorova M,Hoffmann R.J.Proteomics,2011,74(11):2351-2359.
[27] Wang C,Weerapana E,Blewett M M,Cravatt B F.Nat.Methods,2014,11:79-85.
[28] Vila A,Tallman K A,Jacobs A T,Liebler D C,Porter N A,Marnett L J.Chem.Res.Toxicol.,2008,21(2):432-444.
[29] Kim H H,Tallman K A,Liebler D C,Porter N A.Mol.Cell.Proteomics,2009,8(9):2080-2089.
[30] Codreanu S G,Ullery J C,Zhu J,Tallman K A,Beavers W N,Porter N A,Marnett L J,Zhang B,Liebler D C.Mol.Cell.Proteomics,2014,13(3):849-859.
[31] Yang J,Tallman K A,Porter N A,Liebler D C.Anal.Chem.,2015,87(5):2535-2541.
[32] Chen Y,Qin W,Wang C.Curr.Opin.Chem.Biol.,2016,30:37-45.
[33] Soreghan B A,Yang F,Thomas S N,Hsu J,Yang A J.Pharm.Res.,2003,20:1713-1720.
[34] Mirzaei H,Regnier F.Anal.Chem.,2005,77(8):2386-2392.
[35] Mirzaei H,Regnier F.J.Chromatogr.A,2007,1141(1):22-31.
[36] Madian A G,Regnier F.J.Proteome Res.,2010,9(3):1330-1343.
[37] Meany D L,Xie H W,Thompson L D V,Arriaga E A,Griffin T J.Proteomics,2007,7:1150-1163.
[38] Madian A G,Diaz-Maldonado N,Gao Q,Regnier F E.J.Proteomics,2011,74(11):2395-2416.
[39] Madian A G,Myracle A D,Diaz-Maldonado N,Rochelle N S,Janle E M,Regnier F E.J.Proteome Res.,2011,10(9):3959-3972.
[40] Prokai N R L.J.Mass Spectrom.,2011,46:976-985.
[41] Han B,Stevens J F,Maier C S.Anal.Chem.,2007,79(9):3342-3354.
[42] Han B,Hare M,Wickramasekara S,Fang Y,Maier C S.J.Proteomics,2012,75(18):5724-5733.
[43] Dirksen A,Dawson P E.Bioconjugate Chem.,2008,19:2543-2548.
[44] Chavez J,Wu J Y ,Han B,Chung W G,Maier C S.Anal.Chem.,2006,78(19):6847-6854.
[45] Griesser E,Vemula V,Raulien N,Wagner U,Reeg S,Grune T,Fedorova M.Redox Biol.,2017,11:438-455.
[46] Bollineni R C,Fedorova M,Blüher M,Hoffmann R.J.Proteome Res.,2014,13(11):5081-5093.
[47] Chavez J D,Wu J Y,Bisson W,Maier C S.J.Proteomics,2011,74(11):2417-2429.
[48] Roe M R,Xie H W,Bandhakavi S,Griffin T J.Anal.Chem.,2007,79(10):3747-3756.
[49] You X,Yao Y T,Mao J W,Qin H Q,Liang X M,Wang L M,Ye M L.Anal.Chem.,2018,90(21):12714-12722.
[50] Bray B M.Analyst,1952,77:426-429.
[51] Mirzaei H,Regnier F.Anal.Chem.,2006,78(3):770-778.
[52] Mirzaei H,Regnier F.J.Chromatogr.A,2006,1134(1/2):122-133.
[53] Yuan W J,Zhang Y,Xiong Y,Tao T ,Wang Y,Yao J,Zhang L,Yan G Q,Bao H M,Lu H J.Anal.Chem.,2017,89(5):3093-3100.
[54] Afiuni-Zadeh S,Rogers J C,Snovida S I,Bomgarden R D,Griffin T J.BioTechniques,2016,60(4):186-196.
[55] Zhang S W,Fang C Y,Yuan W J ,Zhang Y,Yan G Q,Zhang L,Di Y,Cai Y,Lu H J.Anal.Chem.,2019,91(8):5235-5243.
[56] Chen Y,Cong Y,Quan B Y,Lan T,Chu X Y,Ye Z,Hou X M,Wang C.Redox Biol.,2017,12:712-718.
[57] Tian C P,Liu K K,Sun R,Fu L,Yang J.Anal.Chem.,2018,90(1):794-800.
[58] Chen Y,Liu Y,Lan T,Qin W,Zhu Y T,Qin K,Gao J J,Wang H B,Hou X M,Chen N,Friedmann J P A,Conrad M,Wang C.J.Am.Chem.Soc.,2018,140(13):4712-4720.
[59] Chen Y,Liu Y,Hou X M,Ye Z,Wang C.Chem.Res.Toxicol.,2019,32(3):467-473.
[60] Long M J C,Poganik J R,Aye Y.J.Am.Chem.Soc.,2016,138(11):3610-3622.
[61] Zhao Y,Long M J C,Wang Y,Zhang S,Aye Y.ACS Cent.Sci.,2018,4(2):246-259.
蛋白质组学研究的主要内容包括在蛋白质水平上大规模地分析组织细胞的蛋白质表达水平、翻译后修饰、蛋白质间的相互作用等,从而揭示蛋白质的功能,已在疫苗筛选、指导治疗、临床药物开发及预后判断等领域发挥了重要作用。...
蛋白质组是指一种细胞、组织乃至一种生物所表达的全部蛋白质。相较于传统的蛋白质研究,蛋白质组学可以在大规模水平上一次性鉴定成千上万种蛋白质的表达水平、修饰水平相互作用等,从而揭示蛋白质参与生命活动的作用。...
生物膜主要由磷脂双分子层和镶嵌或附着在其上的蛋白质构成。动物、植物和微生物通常都有一种或多种膜系统。细胞质膜 (质膜) 是最为常见的一种膜系统,负责把细胞质和其他内溶物与周围环境分隔开,并负责与环境交换物质和感受信号。...
自2003年中国人类蛋白质组组织(Chinahumanproteomeorganization,CNHUPO)成立至今,中国的蛋白质组学研究经历了十年多的发展,呈现出百家争鸣、百花齐放的局面.继中国科学家领衔人类肝脏蛋白质组计划(humanliverproteomeproject,HLPP)之后,2014...