摘要:目的:为阿尔茨海默病(AD)发病机制的阐释、早期预防与诊断以及治疗靶点的筛选提供参考。方法:从美国国立生物技术信息中心公共数据平台基因表达数据库中下载基因芯片数据集GSE28146,使用GEO2R在线分析工具筛选出AD相关差异表达基因(DEGs);使用DAVID 6.8生物信息学资源数据库进行基因本体(GO)分析和KEGG通路富集分析;使用STRING数据库和Cytoscape 3.2.1软件进行蛋白-蛋白相互作用(PPI)网络分析。结果与结论:筛选出AD相关DEGs共1 478个,其中上调913个、下调565个。GO分析结果显示,DEGs主要分布于细胞质、膜、细胞外隙中,主要通过转录的正/负调节、核因子κB活性的正调节、Rho蛋白信号转导的调节、蛋白质磷酸化的调节等生物学过程以及蛋白质结合、DNA结合、转录因子活性(序列特异性DNA结合)等分子功能来诱导AD的发生。KEGG通路富集分析结果显示,DEGs显着富集于癌症途径、肺结核、破骨细胞分化、Janus激酶/信号传导及转录激活因子信号通路、叉头转录因子信号通路、EB病毒感染等信号通路上。DEGs编码蛋白的PPI网络含节点蛋白共1 205个、边3 931条;其中的关键核心基因为SOCS3、NEDD4、CBLB,可能是AD发生发展的潜在靶点。
关键词:阿尔茨海默病; 差异表达基因; 生物信息学; 基因本体; KEGG通路富集; 蛋白-蛋白相互作用;
Differentially Expressed Genes Related to Alzheimer's Disease and Their Bioinformatics Analysis
XU Qian SU Yuanqi TAN Yi YANG Yuanjuan
School of Pharmacy, Chongqing Medical and Pharmaceutical College & Chongqing Engineering Research Center of Pharmaceutical Sciences Laboratory Animal Center,Chongqing Medical University
Abstract:OBJECTIVE:To provide reference for interpretation of pathogenesis,early prevention and diagnosis,and selection of therapeutic targets of Alzheimer's disease(AD). METHODS:The gene chip dataset GSE28146 was downloaded from the NCBI public data platform GEO,and the AD-related differentially expressed genes(DEGs) were identified by using GEO2 R online analysis tool. GO analysis and KEGG enrichment pathway analysis were performed by using DAVID 6.8 bioinformatics resource database. The protein-protein interaction(PPI) network analysis was performed by using STRING database and Cytoscape 3.2.1 software. RESULTS & CONCLUSIONS:A total of 1 478 AD-related DEGs were identified,consisting of 913 up-regulated genes and 565 down-regulated genes. GO function enrichment analysis showed that DEGs mainly distributed in cytoplasm,membrane,extracellular space, and induced AD via biological processes such as positive/negative regulation of transcription, positive regulation of NF-κB activity,regulation of Rho protein signaling transduction,protein phosphorylation;via protein binding,DNA binding,transcription factor activity(sequence specific DNA binding)and other molecular functions. KEGG pathway enrichment analysis showed that DEGs was enriched in cancer pathway, pulmonary tuberculosis, osteoclast differentiation, JAK/STAT signaling pathway,FoxO signaling pathway,EB virus infection and other signaling pathways. There are 1 205 nodes and 3 931 edges in the PPI network of DEGs coding protein. Among them,the key genes are SOCS3,NEDD4 and CBLB,which may be the potential target of AD development.
阿尔茨海默病(Alzheimer’s disease,AD)是一种慢性进行性的中枢神经系统退行性疾病,临床上主要表现为记忆障碍、进行性认知功能损害、人格和行为改变等神经精神障碍[1]。AD的发病机制较为复杂,呈现多样性和不确定性,以β-淀粉样蛋白(Aβ)瀑布理论、Tau蛋白学说、氧化应激、炎症机制、线粒体功能障碍和基因突变等假说较为常见[2]。AD是老年痴呆最常见的病因,据统计,60%~80%的痴呆由AD导致,且痴呆已经成为65岁以上人群的第五大死亡原因[3]。近年来,随着人口老龄化进程加剧,我国AD患者的数量迅速增加,现已超过700万人[4]。AD病程长、并发症多样且需要长期护理,给患者家庭和社会带来沉重的经济负担,已成为不容忽视的社会问题[4]。随着高通量测序技术及基因芯片技术的发展,从诸如基因组、转录组、蛋白质组和代谢组等不同层次研究疾病的发生、发展及预后已成为全世界学者共同关注的热点之一[5,6,7]。本研究通过对美国国立生物技术信息中心(NCBI)公共数据平台中的表达谱芯片原始数据进行挖掘,筛选出AD相关差异表达基因(DEGs),并对其进行基因本体(GO)分析、KEGG通路富集分析以及蛋白-蛋白相互作用(PPI)网络分析,以期为AD发病机制的阐释、早期预防与诊断以及治疗靶点的筛选提供理论参考。
1 资料与方法
1.1 资料来源
从NCBI公共数据平台基因表达数据库(GEO,http://www.ncbi.nlm.nih.gov/geo)下载AD相关基因芯片数据集GSE28146(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE28146)。该数据集基于GPL570平台采用[HG-U133_Plus_2]Human Genome U133 Plus2.0 Array阵列芯片(美国Affymetrix公司)检测的30例人海马组织获得,其中对照组8例、AD病例组22例。
1.2 方法
运用GEO2R在线分析工具(https://www.ncbi.nlm.nih.gov/geo/geo2r/?acc=GSE28146)对芯片原始数据进行DEGs的筛选,筛选条件为P<0.05,其中log2FC≥1.0为上调、log2FC<-1.0为下调(式中,“FC”表示AD病例组受试芯片荧光信号强度与对照组相比的差异倍数)[8]。采用GraphPad Prism 5在线软件(https://www.graphpad.com/scientific-software/prism)绘制DEGs火山图。
利用DAVID 6.8生物信息学资源数据库(https://david.ncifcrf.gov/)对DEGs进行GO分析和KEGG通路富集分析(以Fisher确切概率法计算P值,P<0.05为“显着富集”)[9]。将P值由小到大排序,分列出排序前10位的GO功能族和KEGG信号通路。
通过STRING数据库(https://www.string-db.org/)对筛选所得DEGs进行PPI网络分析,设置“可信度(Confidence)”为0.7;借助Cytoscape 3.2.1软件的“CytoHubba”插件对DEGs编码蛋白的相互作用进行可视化展示。其中,节点表示蛋白,边表示蛋白之间的相互联系,节点度值表示与某节点相连边的数量(其值大小与对应节点在网络中的重要程度成正比),将节点度值排序前3位的基因视为关键核心基因。
2 结果
2.1 DEGs的筛选结果
筛选出AD相关DEGs共1 478个,其中上调913个、下调565个。DEGs的火山图见图1。
图1 DEGs的火山图
2.2 DEGs的GO分析及KEGG通路富集分析结果
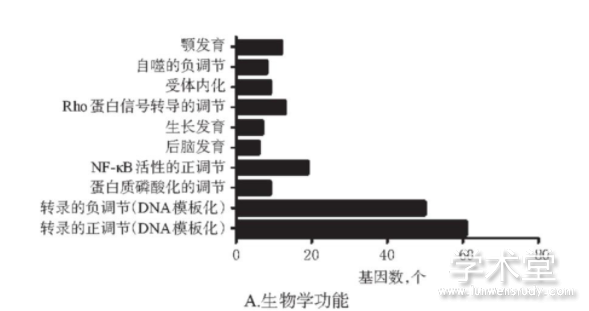
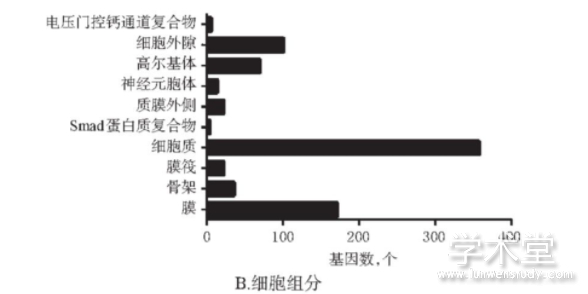
GO分析结果显示,DEGs主要涉及转录的正/负调节、核因子κB(NF-κB)活性的正调节、Rho蛋白信号转导的调节、颚发育、蛋白质磷酸化的调节、自噬的负调节等生物学功能;DEGs主要分布于细胞质、膜、细胞外隙、高尔基体等细胞组分;DEGs主要涉及蛋白质结合、DNA结合、转录因子活性(序列特异性DNA结合)、蛋白质同源二聚体活性等分子功能,详见图2。
KEGG通路富集分析结果显示,DEGs在癌症途径、肺结核、破骨细胞分化、Janus激酶/信号传导及转录激活因子(JAK/STAT)信号通路、叉头转录因子(FoxO)信号通路、EB(Epstein-Barr)病毒感染、转化生长因子β(TGF-β)等信号通路上显着富集,详见图3。
2.3 DEGs编码蛋白PPI网络分析结果
DEGs编码蛋白的PPI网络中,共包含节点蛋白1 205个、边3 931条,见图4。其中,节点度值排序前30位蛋白的PPI网络见图5(以基因表示),节点度值排序前3位的基因为SOCS3、NEDD4和CBLB,是网络中的关键核心基因。
3 讨论
近年来,随着生物芯片、高通量测序等现代生物技术的高速发展和生物信息学分析的日益成熟,通过大数据分析,挖掘在疾病发生发展中起主导作用的相关基因,可为疾病发病机制和治疗评价等研究提供新的思路。例如曹丹等[10]采用生物信息学方法分析肝癌相关DEGs,并构建编码蛋白PPI网络,发现TOP2A基因可能是肝癌相关的核心基因;冯晓飞等[11]利用生物信息学方法对骨肉瘤基因表达谱芯片进行分析,从分子水平上初步分析了骨肉瘤的潜在发病机制;许丁文等[12]应用生物信息学方法分析了丝甘蛋白聚糖对卵巢癌耐药性的影响及作用机制。
图2 DEGs的GO分析结果
图3 DEGs的KEGG通路富集分析结果
AD作为痴呆的主要原因,是目前全球医疗保健领域最严峻的挑战之一[13]。AD致病机制尚未明确,其发病是多基因、多途径、多步骤、多阶段相互作用和相互影响的复杂过程,尚缺乏有效的诊疗手段,对其早发现、早治疗成为了临床亟待解决的难题[1,2,3,4]。为此,本研究应用生物信息学方法,拟初步分析AD发生的潜在分子机制,以期为进一步揭示该症病因提供新的线索,同时为AD治疗新靶点的寻找提供理论依据。
本研究通过生物信息学方法从GEO数据库中搜索得到芯片数据集GSE28146(人脑海马组织,其中健康人脑海马样本8例,AD患者海马样本22例),并对其进行数据挖掘,包括GO分析、KEGG通路富集分析和PPI网络分析。利用GEO2R在线分析工具对该芯片数据进行分析,共筛选出DEGs 1 478个,其中上调913个、下调565个。GO分析结果显示,DEGs主要分布于细胞质、膜、细胞外隙中,主要通过转录的正/负调节、NF-κB活性的正调节、Rho蛋白信号转导的调节、蛋白质磷酸化的调节等生物学功能以及蛋白质结合、DNA结合、转录因子活性(序列特异性DNA结合)等分子功能来诱导AD的发生。KEGG通路富集分析结果显示,DEGs显着富集于癌症途径、肺结核、破骨细胞分化、JAK/STAT信号通路、FoxO信号通路、EB病毒感染等信号通路上。已有研究证实,蛋白质磷酸化[2]、转录调节[14]、NF-κB活性[15]、Rho蛋白信号转导[16]、线粒体自噬[17]等在AD的发生发展中具有重要作用,且TGF-β信号通路[18]、JAK/STAT信号通路[19]、FoxO信号通路[20]及EB病毒感染[21]等信号通路均参与了AD的发生发展,与本文结果基本一致。
图4 DEGs编码蛋白的PPI网络
图5 节点度值排序前30位DEGs的PPI网络
DEGs编码蛋白PPI网络共包含节点蛋白1 205个、边3 931条,其中的关键核心基因为SOCS3、NEDD4、CBLB。
SOCS3基因编码细胞因子信号转导抑制因子3(SOCS3),该蛋白在不同组织、细胞中均广泛表达,是JAK/STAT信号通路的主要负调控因子家族成员之一[22,23]。SOCS3基因位于人染色体17q25.3,由675个核苷酸组成,为抑癌基因之一[24]。Deng J等[25]研究表明,胃癌组织中SOCS3蛋白及其mRNA的表达均较癌旁正常组织明显下降,其表达下调及淋巴结转移是胃癌患者预后的独立危险因素;Jiang BG等[26]研究结果显示,肝癌组织中SOCS3基因DNA甲基化阳性率较高,可导致该基因表达下调,与肝癌细胞的分化、侵袭和转移密切相关,但目前尚未见该基因与AD发生的相关报道。本研究发现,SOCS3基因主要富集于调节蛋白质磷酸化功能族(GO0001932)。既往Tau蛋白学说指出,Tau蛋白的磷酸化在AD发生发展中具有重要意义[2]。由此推断,该基因可能与AD的Tau蛋白学说有关,但有待基础研究予以验证。
发育抑制蛋白4(NEDD4)是一种重要的HECT型泛素连接酶,由NEDD4基因编码,在多种神经退行性疾病相关组织中呈高表达[27]。NEDD4基因在机体发育和疾病进展过程中具有重要的生物学功能,并可结合多种底物、调节多种信号通路。有研究发现,NEDD4酶可通过对成纤维细胞生长因子受体1(FGFR1)的泛素化来调控神经和胚胎发育[28]。此外,在细胞增殖、分化、凋亡中起重要作用的第10号染色体缺失的磷酸酶及张力蛋白同源物基因(PTEN)为NEDD4酶的底物分子,后者可通过结合生长因子受体结合蛋白10(Grb10)来负调控胰岛素样生长因子1(IGF1)通路,最终发挥对细胞增殖、分化、迁移的调节作用[29,30]。同时有研究还发现,NEDD4泛素化降解丝裂原诱导因子6(Mig6)与肿瘤细胞迁移有关[31]。此外,NEDD4可调控获得性免疫,可通过降解泛素连接酶E3来促进T细胞的激活[32];还可通过刺激B细胞中的肿瘤坏死因子受体相关因子3(TRAF3)发生赖氨酸63(K63)形式的泛素化来调节CD40介导的蛋白激酶B(Akt)通路的激活,从而参与免疫球蛋白的转换[33]。上述研究表明,NEDD4酶可通过泛素化调控和获得性免疫调节来参与多种神经退行性疾病的发生发展,可作为研究AD发病机制的潜在靶点之一。本研究结果显示,NEDD4基因主要富集于对钙的反应功能族(GO0031623),但该基因如何调节钙反应功能族及此功能族在AD发生发展中的作用尚有待进一步研究。
CBLB基因编码的泛素连接酶E3(CBLB)属于环指(RING)型泛素连接酶,由氮端保守的酪氨酸激酶(TKB)结构域、RING结构域以及其他蛋白-蛋白结合模体组成。该酶中的TBK结构域可识别并结合底物蛋白酪氨酸激酶,RING结构域可募集泛素结合酶E2,从而启动底物泛素化降解,参与细胞内信号转导的负向调控[34]。同时,CBLB酶也可作为连接蛋白的招募信号分子,从而激活或抑制下游的信号通路分子,发挥促进细胞增殖、转移和抗凋亡等作用[35,36]。本研究结果发现,CBLB基因主要富集于信号转导功能族(GO0007165),在生物学功能上富集于蛋白质结合相关功能族。但笔者尚未发现该基因与AD发生的相关研究,其与AD的相关性有待确证。
综上所述,本研究基于生物信息学方法筛选出了AD相关DEGs,并分析了其GO功能以KEGG富集通路,最终筛选出了SOCS3、NEDD4、CBLB等3个关键核心基因,可作为AD发病机制研究的潜在靶点,在一定程度上为深入揭示AD的分子机制和治疗靶点提供了研究方向和理论依据。在后续研究中,本课题组将进一步针对我国AD人群进行分子生物学基础研究,对上述结论进行确证。
参考文献
[1] DOS SANTOS PICAN?O LC,OZELA PF,DE F?TIMA DE BRITO BRITO M,et al. Alzheimer’s disease:a review from the pathophysiology to diagnosis,new perspectives for pharmacological treatment[J]. Curr Med Chem,2018,25(26):3141-3159.
[2] SERRANO-POZO A,FROSCH MP,MASLIAH E,et al.Neuropathological alterations in Alzheimer disease[J].Cold Spring Harb Perspect Med,2011. DOI:10.1101/cshperspect.a006189.
[3] GBD 2016 Dementia Collaborators. Global,regional,and national burden of Alzheimer’s disease and other dementias,1990-2016:a systematic analysis for the global burden of disease study 2016[J]. Lancet Neurol,2019,18(1):88-106.
[4] JIA J,WEI C,CHEN S,et al. The cost of Alzheimer’s disease in China and re-estimation of costs worldwide[J].Alzheimers Dement,2018,14(4):483-491.
[5] ZHANG L,NIE Q,SU Y,et al. MicroRNA profile analysis on duck feather follicle and skin with high-throughput sequencing technology[J]. Gene,2013,519(1):77-81.
[6] YONGFENG H,FAN Y,JIE D,et al. Direct pathogen detection from swab samples using a new high-through put sequencing technology[J]. Clin Microbiol Infect,2011,17(2):241-244.
[7] HE X,LI X,GUO Y,et al. Newborn screening of genetic mutations in common deafness genes with bloodspot based gene chip array[J]. Am J Audiol,2018,27(1):57-66.
[8]张东亮,赵舒煊,向高,等.基于高通量芯片对强直性脊柱炎的生物信息学分析[J].第三军医大学学报,2019,41(1):63-70.
[9]郭明飞,高家荣,姜辉,等. 2型糖尿病模型大鼠肝脂代谢相关基因的筛选及生物信息学分析[J].中国生物化学与分子生物学报,2017,33(7):726-735.
[10]曹丹,余乐,王丽春.肝癌相关差异表达基因的生物信息学分析及蛋白互作网络构建[J].四川大学学报(医学版),2018,49(6):899-903.
[11]冯晓飞,马遥,赵舒煊,等.骨肉瘤基因表达谱芯片的生物信息学分析[J].华中科技大学学报(医学版),2019,48(3):281-286.
[12]许丁文,熊彦,严慧深,等.基于生物信息学分析丝甘蛋白聚糖对卵巢癌耐药性的影响及作用机制[J].中国药房,2019,30(1):46-51.
[13] SCHELTENS P,BLENNOW K,BRETELER MM,et al.Alzheimer’s disease[J]. Lancet,2016,388(10043):505-517.
[14] ZOU C,WANG J,HUANG X,et al. Analysis of transcription factor-and ncRNA-mediated potential pathogenic gene modules in Alzheimer’s disease[J]. Aging:Albany NY,2019,11(16):6109-6119.
[15] LIAN H,YANG L,COLE A,et al. NFκB-activated astroglial release of complement C3 compromises neuronal morphology and function associated with Alzheimer’s disease[J]. Neuron,2015,85(1):101-115.
[16] GAO Y,LI XI,LIU XH,et al. Determining association of rho kinase 1 gene polymorphisms with risk of Alzheimer’s disease:a multicenter pilot study[J]. Ann Transl Med,2018. DOI:10.21037/atm.2018.05.51.
[17] MOLOUDIZARGARI M,ASGHARI MH,GHOBADI E,et al. Autophagy,its mechanisms and regulation:implications in neurodegenerative diseases[J]. Ageing Res Rev,2017. DOI:10.1016/j.arr.2017.09.005.
[18] DINIZ LP,MATIAS I,SIQUEIRA M,et al. Astrocytes and the TGF-β1pathway in the healthy and diseased brain:a double-edged sword[J]. Mol Neurobiol,2019,56(7):4653-4679.
[19] LI W,LIU H,YU M,et al. Folic acid alters methylation profile of JAK-STAT and long-term depression signaling pathways in Alzheimer’s disease models[J]. Mol Neuroibol,2016,53(9):6548-6556.
[20] FERNANDEZ AM,HERVAS R,DOMINGUEZ-FRAILE M,et al. Blockade of the interaction of calcineurin with FOXO in astrocytes protects against amyloid-β-induced neuronal death[J]. J Alzheimers Dis,2016,52(4):1471-1478.
[21] SHIM SM,CHEON HS,JO C,et al. Elevated EpsteinBarr virus antibody level is associated with cognitive decline in the Korean elderly[J]. J Alzheimers Dis,2017,55(1):293-301.
[22] SOROKINA LN,MINEEV VN,LIM VV. Role of negative regulators of SOCS1,SOCS3,and SOCS5 gene transcription in the negative cell signaling regulation system in asthma[J]. Ter Arkh,2017,89(3):43-47.
[23] GUI T,HE BS,GAN Q,et al. Enhanced SOCS3 in osteoarthiritis may limit both proliferation and inflammation[J].Biotech Histochem,2017,92(2):107-114.
[24] CHIKUMA S,KANAMORI M,MISE-OMATA S,et al.Suppressors of cytokine signaling:potential immune checkpoint molecules for cancer immunotherapy[J]. Cancer Sci,2017,108(4):574-580.
[25] DENG J,JIAO X,LIU H,et al. Lymph node metastasis is mediated by suppressor of cytokine signaling-3 in gastric cancer[J]. Tumour Biol,2013,34(6):3627-3636.
[26] JIANG BG,WANG N,HUANG J,et al. Tumor SOCS3methylation status predicts the treatment response to TACE and prognosis in HCC patients[J]. Oncotarget,2017,8(17):28621-28627.
[27] KWAK YD,WANG B,LI JJ,et al. Upregulation of the E3ligase NEDD4-1 by oxidative stress degrades IGF-1 receptor protein in neurodegeneration[J]. J Neurosci,2012,32(32):10971-10981.
[28] PERSAUD A,ALBERTS P,HAYES M,et al. NEDD4-1binds and ubiquitylates activated FGFR1 to control its endocytosis and function[J]. EMBO J,2011,30(16):3259-3273.
[29] WANG X,TROTMAN LC,KOPPE T,et al. NEDD4-1 is a proto-oncogenic ubiquitin ligase for PTEN[J]. Cell,2007,128(1):129-139.
[30] VECCHIONE A,MARCHESE A,HENRY P,et al. The Grb10/NEDD4 complex regulates ligand-induced ubiquitination and stability of the insulin-like growth factorⅠreceptor[J]. Mol Cell Biol,2003,23(9):3363-3372.
[31] SUN M,CAI J,ANDERSON RA,et al. TypeⅠγphosphatidylinositol phosphate 5-kinaseⅠ5 controls the ubiquitination and degradation of the tumor suppressor mitogen-inducible gene 6[J]. J Biol Chem,2016,291(41):21461-21473.
[32] LIU Q,ZHOU H,LANGDON WY,et al. E3 ubiquitin ligase CBLB in innate and adaptive immunity[J]. Cell Cycle,2014,13(12):1875-1884.
[33] FANG DF,HE K,WANG N,et al. NEDD4 ubiquitinates TRAF3 to promote CD40-mediated Akt activation[J]. Nat Commun,2014. DOI:10.1038/ncomms5513.
[34] LIYASOVA MS,MA K,LIPKOWITZ S. Molecular pathways:CBL proteins in tumorigenesis and antitumor immunity-opportunities for cancer treatment[J]. Clin Cancer Res,2015,21(8):1789-1794.
[35] TSYGANKOV AY,TECKCHANDANI AM,FESHCHENKO EA,et al. Beyond the RING:CBL proteins as multivalent adapters[J]. Oncogene,2001,20(44):6382-6402.
[36] THIEN CB,LANGDON WY. CBL:many adaptations to regulate protein tyrosine kinases[J]. Nat Rev Mol Cell Biol,2001,2(4):294-307.