摘 要: 定向进化作为改善生物分子目的活性的有效策略之一,已广泛应用于食品、工业、医学等领域,该技术依赖于高质量突变体文库的建立与高效目的产物选择与筛选。近年来,随着CRISPR的飞速发展,相继开发了各类CRISPR衍生工具以满足不同需求。其中,一些新型CRISPR工具的出现为定向进化技术带来了更大的发展潜力,使人们可以在广泛宿主中实现对特定基因的原位进化。同时,借助人工或天然途径产生遗传多样性的方式给了人们更多的选择,可以根据研究需求采取更高效的进化策略。该文首先介绍各类CRISPR工具,随后总结CRISPR介导的突变与筛选平台,最后讨论CRISPR在定向进化技术中的发展趋势与机遇。
关键词 : 基因编辑; CRISPR/Cas;定向进化; CRISPR介导的定向进化;
Abstract: To gain the desirable activity of biomolecules, directed evolution is served as a competent technology depending on high quality mutant library, effective selection and screening, which has been widely used in food, industry and medical fields. CRISPR(clustered regularly interspaced short palindromic repeats) has developed rapidly in recent years, and various CRISPR derivatives have been developed to meet different demands. The emergence of some new CRISPR tools has given greater development potential for directed evolution, allowing people to evolve specific genes in situ in a wide range of hosts. At the same time, the way of generating genetic diversity through artificial or natural pathways has given people more choices, and more efficient evolution strategies can be adopted according to research needs. This article will first introduce the CRISPR tools, then summarize the CRISPR-mediated mutation and screening platforms, and finally discuss the development trends and opportunities of the CRISPR in the field of directed evolution.
Keyword: gene editing; CRISPR/Cas; directed evolution; CRISPR-mediated directed evolution;
1、 引言
随着定向进化技术的不断发展,利用传统的物理、化学和生物诱变技术[1,2]寻找所需的生物性状往往会带来过多有害突变,已无法满足研究或应用的需求。于是人们通过先对单个基因或生物途径建立突变文库,再筛选得到具有更高活性的酶[3]、目的产物产量更高的生物体[4]。但这些方法大多需要体外建立突变文库、体内进行筛选,反复迭代,这无疑增加了得到目的突变体的难度与成本。而CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)技术的出现和发展,使人们可以站在新的角度,利用新的工具开发定向进化技术,以解决目前所面临的问题。
借助CRISPR/Cas系统,可以实现对特定基因或区域进行编辑。根据小向导RNA(small guide RNA,sg RNA)引导的Cas蛋白或Cas融合蛋白的特性,可以使该区域基因产生双链断裂、单链缺刻和单碱基替换[5]等变化。再由修复机制、错配机制或化学催化机制引入突变,实现突变文库的构建。结合生物大分子如转录因子、核酸适配体等,CRISPR的功能可以扩展到分子检测领域。由此可见,CRISPR系统的性能与定向进化技术的需求有着很高的契合度。
另外,通过Pub Med数据库检索CRISPR与定向进化的论文数量发表情况可知,定向进化论文数从1975年开始,到2018年左右出现平台期,其年增长速率仅为2%~5%。而CRISPR在同期正呈蓬勃发展的趋势,年增长速率达14%~24%。因此,借助CRISPR为定向进化技术提供新的方法和策略,对于定向进化技术将是一个新的机遇。
本文首先介绍CRISPR技术的最新进展,包括不同核酸酶及诱导系统的介绍。其次,从定向进化的两个核心出发,总结利用CRISPR工具产生遗传多样性的策略以及借助CRISPR工具进行筛选与选择的方法。最后设想CRISPR介导的定向进化(CRISPR-Mediated Directed Evolution,CDE)可能的未来发展方向。
2、 不断丰富的CRISPR工具箱
CRISPR/Cas作为适应性免疫系统,存在于许多细菌和古细菌中,由规律的间隔成簇短回文重复序列组成。在CRISPR序列附近,可以找到编码Cas蛋白的基因短簇[6]。CRISPR系统主要分为两类,1类包含、和型CRISPR系统,2类包括、和型CRISPR系统[7]。其中,每个类型又具有其特殊的标签Cas蛋白:、和型的标签蛋白分别为Cas3、Cas10和Csf1[8];、和型的标签蛋白则分别为Cas9、Cas12和Cas13[9]。尽管许多CRISPR系统被相继开发出来,但Cas9和Cas12被人们研究得最为透彻,故本小节主要针对Cas9与Cas12系统进行介绍。
2.1、 CRISPR-Cas9及其衍生变体
Cas9属于2类型CRISPR系统,该系统最初来自于化脓性链球菌,是一种多结构域的DNA核酸内切酶,包含一个HNH样核酸酶结构域和一个Ruv C样核酸酶结构域,分别负责切割与向导RNA(guide RNA,g RNA)序列互补的DNA链(靶向链)和靶链互补链(非靶向链)[10]。自Jinek等[11]首次利用CRISPR-Cas9进行体外基因组编辑,到利用CRISPR-Cas9系统对哺乳动物[12,13]、植物[14,15]基因组进行编辑,人们可以根据需要设计出sg RNA[11],通过sg RNA与化脓性链球菌Cas9(Streptococcus pyogenes Cas9,Sp Cas9)组装并结合到靶向DNA 3′处的前间隔序列邻近基序(Protospacer Adjacent Motif,PAM),实现对目的位点DNA的切割(图1(a))。根据Cas9的结构,通过向HNH结构域引入H840A或者向Ruv C结构域引入D10A突变,可以得到Cas9缺刻酶(Cas9 nickase,n Cas9),仅切割DNA双链中的其中一条单链[16,17]。当两个突变同时被引入时,形成无活性的Cas9(Catalytically Deficient Cas9,d Cas9),这时d Cas9仅有靶向作用而无DNA链切割活性[18,19]。
但在Sp Cas9系统中,存在PAM序列限制、Sp Cas9蛋白较大和脱靶等问题,限制了其进一步的应用。早期人们通过开发其他细菌和古细菌的Cas9同源蛋白来改善这些问题。比Sp Cas9更小的来自金黄色葡萄球菌的Cas9(Sa Cas9)可以识别5′-NNGRRT-3′PAM[20],且当引入D10A和N580A突变时,Sa Cas9同样可显示出缺刻酶活性[21]。来自脑膜炎奈瑟氏球菌Cas9 (Nm Cas9)则可以识别5′-NNNRRT-3′或5′-NNNNGMTT-3′PAM且脱靶效应降低[22]。同样还有嗜热链球菌Cas9(St1Cas9)可以识别5′-NGGNG-3′或5′-NNAGAAW-3′[23],空肠弯曲杆菌C a s 9 (C j C a s 9)可以识别5′-NNNNACAC-3′或5′-NNNNRYAC-3′[24],犬链球菌(Sc Cas9)可以识别5′-NNG-3′[25]。
图1 主要的CRISPR-Cas工具分子特征与调控
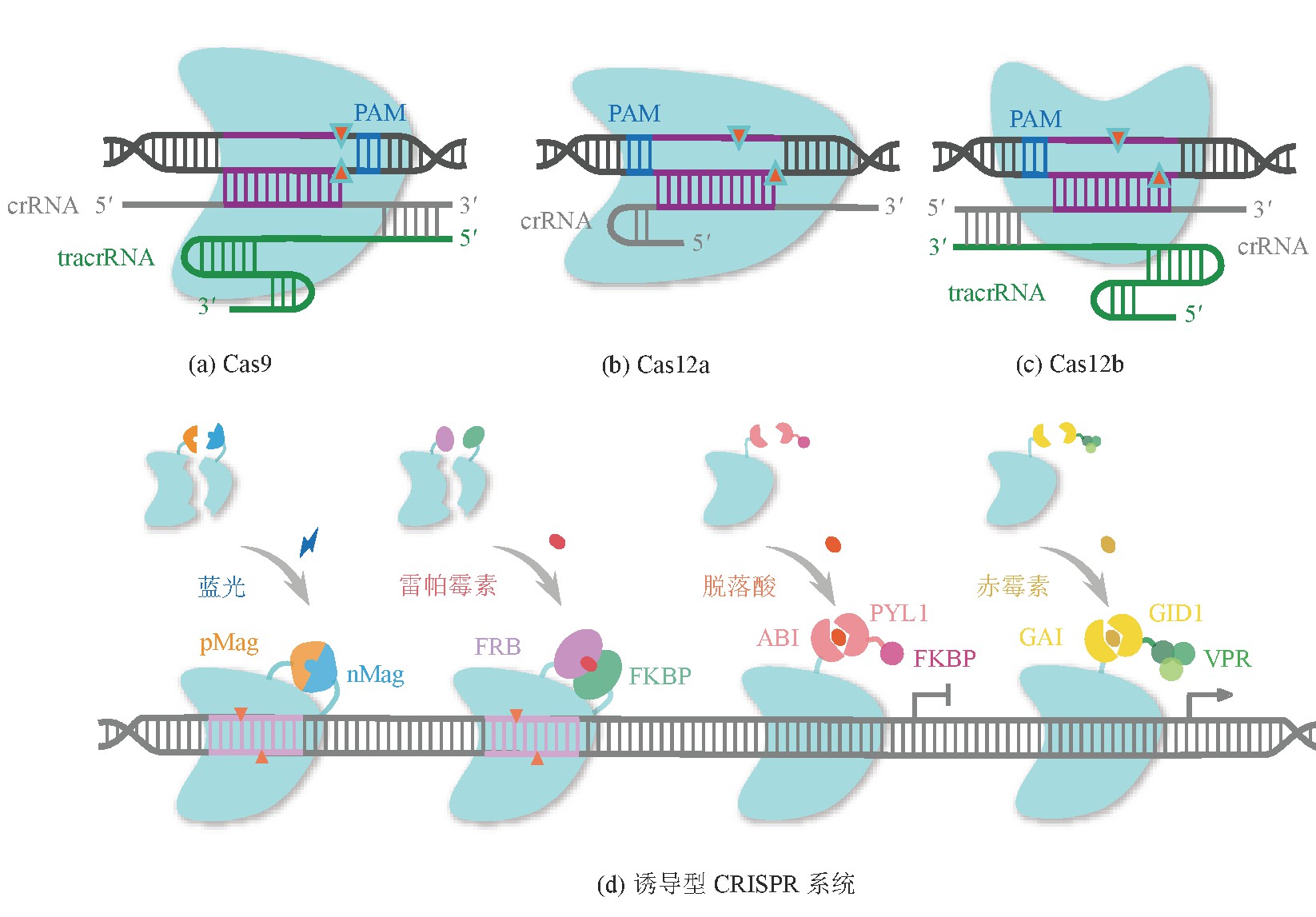
Fig.1 Main CRISPR-Cas tools’molecule characteristic and regulation
通过基于结构信息,借助细菌生存选择的定向进化策略与突变位点组合设计,Sp Cas9 VQR、EQR和VRER变体可以分别识别NGA、NGAG和NGCG PAM[26]。借助噬菌体辅助的连续进化,得到可以识别广泛PAM序列(NG、GAA和GAT)且特异性更高的Sp Cas9变体x Cas9[27]。另一种Sp Cas9变体(Sp Cas9-NG)则是通过结构设计获得识别NG PAM的能力[28]。Sp Cas9、x Cas9和Sp Cas9-NG的PAM均包含鸟嘌呤(G),但人们希望PAM能不受依赖G的限制。Miller等[29]借助噬菌体辅助进化,得到3个对非G PAM具有活性的Sp Cas9变体,分别可以识别NRRH、NRCH和NRTH PAM;利用结构导向工程策略同样得到识别非G PAM的Sp G(NGN PAM)和Sp RY(NRN>NYN PAM)[30]。这些新型Sp Cas9变体的开发使得CRISPR-Cas9系统几乎可以靶向基因组任意位置而不再受PAM序列的限制。
改善CRISPR系统特异性和表达泄露对宿主的影响,可以对Cas9蛋白进行组合诱变[31]以及将Cas9蛋白与诱导性二聚体结构域进行融合,如光介导的Mag蛋白二聚化[32]、雷帕霉素诱导的FRB与FKBP互作、脱落酸及赤霉素诱导的对转录的激活或抑制[33,34],以实现时间或空间上的表达(图1(d))。
2.2、 CRISPR-Cas12家族
Cas12a (Cpf1)属于2类型核酸内切酶,典型的包括氨基酸球菌Cas12a(As Cas12a)和毛螺菌ND2006 Cas12a(Lb Cas12a),虽然弗朗西斯菌Cas12a(Fn Cas12a)和食肉莫拉菌Cas12a(Mb Cas12a)也被报道,但活性较不稳定[35]。与Cas9不同,Cas12a仅需要CRISPR RNA (cr RNA),而无需反式激活cr RNA(transactivating CRISPR RNA,tracr RNA)作为引导,识别富含胸腺嘧啶(T)的PAM,并产生5′黏性末端的DNA双链断裂[36](图1(b))。Cas12a也被成功地进行改造以获得更广泛的PAM位点,其中携带S542R/K607R和K548V/N552突变的As Cpf1分别识别TYCV和TATV PAM,E174R/S542R/K548R变体(en As Cas12a)可以针对多种PAM,如TTYN、VTTV和TRTV等。与野生型As Cas12a相比,E174R/S542R变体与en As Cas12a在TTTV PAM上的编辑效率可以提高2倍[37,38]。Cas12b(C2c1)是一种含有Ruv C结构域,不含HNH结构域的核酸内切酶,它需要cr RNA和tracr RNA识别靶标DNA[9,39]。来源于嗜酸热脂环酸杆菌的Cas12b系统(Aa Cas12b)(图1(c)),其由于比Cas9和Cas12a更小,具有更宽的耐受温度,已成功用于哺乳动物和植物的基因组工程研究[40,41]。还有如Cas12e[42]、Cas12d[43]、Cas12g、Cas12i、Cas12c和Cas12h[44],均被发现并进行了表征。
3 、利用CRISPR工具实现的遗传多样性
CRISPR工具首先作为基因编辑系统,可以与DNA修复机制串联,既可以对单基因进行突变,也可以在基因组水平引入广泛的变化。同时CRISPR作为招募平台,通过蛋白质融合或sg RNA蛋白相互作用,将功能域募集到特定位点,也可以在目标基因中引入突变。
3.1、 通过CRISPR产生双链断裂进行突变库的建立
利用CRISPR系统可以对靶向DNA序列进行切割的特性,并激活DNA修复机制,可以在基因组DNA中引入多样性的突变,表1总结了基于该原理的定向进化平台。通过Cas9-sg RNA复合物对DNA双链的切割作用产生双链断裂(Double-Strand Break,DSB),而DSB的修复主要通过非同源末端连接(non-Homologous End Joining,NHEJ)途径和同源性修复(HomologyDirected Repair,HDR)途径完成[45](图2(a))。其中,NHEJ不依赖同源模板[46],通常产生较小的插入或缺失,导致不可控的随机突变,对单核苷酸取代能力不足[47]。HDR途径则需要外源双链或单链寡脱氧核苷酸(double-strand/single strand DNA,ds DNA/ss DNA)提供模板,所以其擅长利用携带突变的供体模板进行单核苷酸的替换,但效率一般较低[48]。由于依赖HDR途径可以获得更为精确的人工设计突变,不会造成基因大片段的缺失,于是人们开展了提高HDR效率的研究,如调节修复机制[49,50]、合理设计供体模板等[51,52]。
3.1.1、 大肠杆菌底盘系统
在原核生物中,CRISPR介导的DNA链切割可以杀死未被编辑过的细胞,从而可以增加筛选效率又避免使用抗性标记[53]。Wang等[54]借助λRed与ds DNA/ss DNA开发了多重自动化基因组工程(Multiplex Automated Genome Engineering,MAGE),该系统可同时靶向染色体多个位置。将CRISPR-Cas9引入该系统则可以提高较大核糖体结合位点(Ribosome Binding Site,RBS)的替换效率(与MAGE相比提高了56%),并且在单轮循环中引入单突变和双突变的效率比MAGE分别高93%和90%[55]。Li等[56]报道了一种基于CRISPR-Cas9的迭代基因组编辑系统。该系统以ds DNA作为编辑模板,实现基因缺失、插入和替换的效率可接近100%,随后针对β-胡萝卜素,进行了甲基赤藓糖醇磷酸和中心代谢途径的组合优化,筛选得到的最优菌株在发酵中可产出2.0 g/L的β-胡萝卜素。
随着DNA合成与测序成本的降低,人们可以借助高通量方法来在定向进化过程中产生突变并分析表型(图2(b))。结合大规模并行寡核苷酸合成和测序,Garst等[57]开发了可在全基因组范围内产生能追踪的编辑平台(CRISPR-Enabled Trackable Genome Engineering,CREATE)。该平台的核心设计是将每个g RNA连接到同源修复盒上,该盒既可以编辑基因位点,又可以作为条形码来追踪基因型与表型关系,实现引导切割的g RNA和同源修复模板序列的偶联。借助CREATE可以对大肠杆菌耐热和耐药性的相关基因绘制超过50 000个全基因组突变,并鉴定其赋予目标表型的突变。利用迭代CREATE(i CREATE)系统[58],研究人员为得到高葡萄糖和木糖利用率且抗水解产物抑制的大肠杆菌菌株以用于3-羟基丙酸(3-Hydroxypropionic Acid,3HP)生产,针对30个基因中约40 000个突变设计文库,得到的突变株可以在高水解产物浓度下达到亲本株3HP产量的7~8倍。同时Liu等[59]又针对115个基因中约162 000个突变设计文库,优化了3HP合成代谢相关途径,使突变株3HP产量高出野生型菌株60倍。这种系统的局限性是在追踪基因型与表型的关系时,以质粒为载体的条形码稳定性较低,但可以采取基因组整合的办法解决该问题。
表1 基于双链断裂的CDE策略
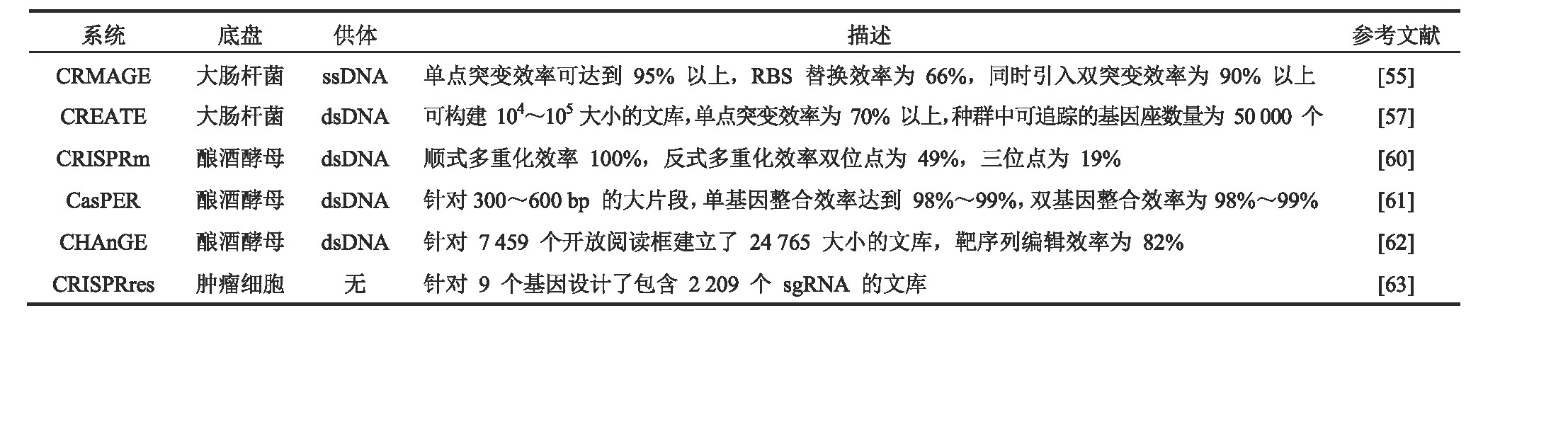
图2 DNA修复途径介导的定向进化策略
Fig.2 DNA repair pathway mediated directed evolution strategy
3.1.2、 酵母底盘系统
酵母的代谢途径工程研究对于生物燃料、化学药品的生成具有重要意义,故人们开展了酵母中的基因组多样性研究。早期人们使用的突变文库多为可以在酵母细胞内自我复制的环状质粒,这种着丝粒质粒在单细胞水平拷贝数差异很大,相当于在表型与基因型之间增加了拷贝数的干扰项,这会导致定向进化的效率大大降低。
R y a n等[60]在酵母中建立了CRISPRm(Multiplex CRISPR System),该系统以ds DNA为模板,可在二倍体或多倍体酿酒酵母基因组中创建多个DSB。另外,为增加CRISPRm的编辑效率,将sg RNA与自切割肺炎三角洲病毒核酶3′端融合以保护sg RNA免受核酸外切酶的影响。最后在单轮定向进化中筛选出纤维糊精转运蛋白变体,将纤维二糖到酒精的发酵速率提高了10倍。
对于多基因相关代谢途径的优化,可以利用CRISPR系统对酿酒酵母基因组中甲羟戊酸代谢相关的5个基因进行组合编辑[64],得到31个突变株,其中产量最高的突变菌株比野生型菌株高41倍。Cas PER (Cas9-Mediated Protein Evolution Reaction)[61]是一种CRISPR-Cas9介导的大序列定向进化方法,可以整合300~600 bp的供体模板,其首先通过易错PCR得到甲羟戊酸途径两个关键代谢酶的突变文库,再利用CRISPR系统在靶标处引入DSB后发生的HDR,使突变文库整合到靶向位点,最后得到了异戊二烯产量增加11倍的突变体。
借助高通量寡核苷酸合成与测序技术同样可以实现酵母基因组范围的定向进化研究。Guo等[65]介绍了一种基于CRISPR-Cas9的策略用于在酵母细胞中同时创建数百个无选择标记的遗传变异,并针对315个特性不明的小型开放阅读框进行评估,研究这些小型开放阅读框对环境适应性生长的必要性,讨论了该策略用于定向进化的可能性。CRISPR-Cas9与HDR辅助的基因组规模工程(CRISPR-Cas9 and Homology-DirectedRepair-Assisted Genome-Scale Engineering,CHAn GE)[62]可以实现单核苷酸精度的全基因组突变,进化出对生长抑制剂(如糠醛、乙酸)具有耐受性的菌株。在酵母中也可以使用类似大肠杆菌中CREATE的策略,利用条形码对每个引导-供体对序列进行标记,实现对突变文库的高通量测序和功能分析[66]。同时,该实验还发现利用融合蛋白Lex A-Fkh1p将供体DNA招募到断裂位点,可以将编辑效率提高5倍以上[66]。
在基因组水平对基因表达水平进行单一类型的调节(上调、下调或敲除)是不全面的,因为其无法研究具有不同表达水平基因间的相互作用对复杂表型的影响。多功能基因组水平CRISPR系统(Multi-Functional Genome-Wide CRISPR,MAGIC)[67]可以有效解决这一问题。MAGIC使用3个正交的Cas蛋白开发了三功能CRISPR系统,将基因激活、干扰和缺失整合在一起。与CHAn GE相比,在相同筛选条件下MAGIC可以发现更多糠醛耐受性相关基因间的相互作用,进化出糠醛耐受性菌株的效率更高。
3.1.3、 植物与动物底盘系统
尽管在酵母中HDR途径非常活跃[68],但是在植物和哺乳动物产生DSB后首个反应是NHEJ。主要原因是动植物中NHEJ途径发生的概率要高于HDR,且难以传递足够的供体DNA(修复模板)。目前利用NHEJ途径可以实现广泛的动植物基因缺失文库的建立[69,70,71,72,73]。尽管多数条件下NHEJ途径会导致基因的插入和缺失,使开放阅读框出现移码突变,但借助NHEJ开发定向进化平台仍然是可行的。例如,在水稻中开发的CRISPR-Cas9定向进化平台[74],可以进化SF3B1剪接体蛋白,以抵抗剪接抑制剂。其sg RNA文库靶向必需基因SF3B1,并从15 000个愈伤组织中得到6个抗剪接抑制剂(GEX1A)的SF3B1突变体。
在哺乳动物底盘中,N H E J产生的基因多样性可以用于细胞耐药性研究。CRISPRres(CRISPR-Induced Resistance in Essential Genes)系统[63]可以利用大规模的sg RNA文库进行癌细胞抗药性的遗传筛选,快速获得和鉴定必需基因中的耐药性突变。为验证系统的有效性,结合蛋白晶体结构,解析了烟酰胺磷酸核糖基转移酶突变前后与抗癌药KPT-9274的结合机制。I p s a r o等[75]对赖氨酸甲基转移酶(DOT1L)的KMT结构域设计了包含73个sg RNA的文库,并在EPZ-5676抑制剂的选择作用下得到具有VVEL293MM(两个蛋氨酸替换为两个缬氨酸,同时缺失谷氨酸和赖氨酸残基)突变的DOT1L变体,生化分析发现该突变可以使酶活增加,同时降低对EPZ-5676抑制剂的敏感性。
对于抗体工程,NHEJ无法满足精确位点突变的需求,所以人们开发了利用HDR获得高效抗体的研究。早期,研究人员利用CRISPR-Cas9,针对BRCA1第18个外显子中6个碱基[76],克隆了包含随机六聚体的HDR文库,证明了利用借助HDR对基因组区域进行饱和编辑的可能性。Mason等[77]利用单链寡核苷酸作为供体模板,将文库引入抗鸡蛋溶菌酶抗体(HEL3)重链可变域(VH)的互补决定区3 (CDRH3),使用定向进化和高通量筛选方法获得了新的CDRH3序列。将DNA供体形式由ss DNA变为可自线性化质粒,可以增加HDR的效率,再借助易错PCR对HEL3的VH区生成随机突变文库,筛选出亲和力范围在皮摩尔范围内的变体[78]。
3.2 、CRISPR系统与具有诱变功能的蛋白联用
借助CRISPR系统对基因组产生DSB后修复途径,往往会对宿主本身造成损伤,并且受到需要提供HDR供体模板的限制,而利用Cas9的引导功能,引导诱变功能蛋白可以解决这些问题[79]。碱基编辑器是由DNA靶向模块(d Cas9或n Cas9)与催化结构域(胞嘧啶或腺苷脱氨酶[80,81])组成的融合蛋白,其中靶向模块可以结合到特定的DNA位点,随后sg RNA与靶DNA结合形成“R-环”[82],导致一小段单链DNA移位,这一小段单链DNA作为催化窗口被催化结构域进行碱基替换。由于碱基编辑的DNA靶向模块使用的是d Cas9或n Cas9,所以并不会导致DSB的产生。借助目前开发的各类CRISPR融合蛋白平台,可以实现单碱基的变化、局部序列的多样化[83,84],用于生成新的蛋白质突变体或作物改良[85,86]等。表2为相应平台的汇总描述。
3.2.1、 碱基编辑工具的开发
胞嘧啶碱基编辑器(Cytosine Base Editor,CBE)可以令胞嘧啶(C)脱去氨基,使其转化为尿嘧啶(U),从而将C·G碱基对转化为T·A碱基对(图3(a))。第一代碱基编辑器由来自大鼠的胞嘧啶脱氨酶APOBEC1,通过连接肽与d Cas9连接。以PAM的远端作为位置1,其有效脱氨窗口在距PAM的4~8位。由于尿嘧啶糖基化酶(Uracil DNA Glycosylase,UDG)经常会除去尿嘧啶,导致编辑效率较低(体外脱氨效率为25%~40%,细胞内脱氨效率为0.9%~7.7%),所以在d Cas9蛋白的C末端偶联了UDG的抑制剂UGI (Uracil DNA Glycosylase Inhibitor),开发了第二代碱基编辑器,使体内编辑效率与第一代碱基编辑器相比提高了3倍。而如果将d Cas9替换为n Cas9(D10A),则可将第二代碱基编辑器效率进一步提升2~6倍[80]。借助丰富的CRISPR工具盒,人们开发了诸多针对不同PAM或者编辑窗口的碱基编辑器。如利用Sp Cas9变体(VQR-BE3、VRER-BE3)分别识别NGAN和NGCG PAM,编辑窗口分别为4~11和3~10位[90];利用不同来源的Cas蛋白(Sa-BE3、Sa KKH-BE3和Cas12a-BE)分别识别NNGRRT、NNNRRT和TTTV[90,91]。
腺嘌呤碱基编辑器(Adenine Base Editor,ABE)则是可以将T·A转化为C·G (图3(b))。与CBE类似,人们开展了一系列ABE的优化及应用[92,93,94]。例如,Wang等[95]通过对ABE进行核心模块组合优化(包括sg RNA、n Cas9、脱氨酶与核定位信号肽),获得了具有更精确的编辑窗口、更高编辑效率的s ABE系统。
表2 基于与诱变功能蛋白联用CDE策略

3.2.2 、基于碱基编辑的定向进化技术
定向进化的实现需要借助单碱基取代和局部序列多样化,创建遗传突变库[96]。CRISPR-X[83]和TAM (Targeted AID-mediated Mutagenesis)[84]是两个碱基编辑介导的产生局部序列多样化的平台。其中,CRISPR-X使用的sgRNA融合有两个MS2结合适体,可以招募MS2-AID (Activation Induced Cytidine Deaminase,AID)融合蛋白实现对局部区域的诱变(图3(c))。Hess等[83]对野生型绿色荧光蛋白进行诱变,得到S65T和Q80H的绿色荧光蛋白变体(加强型绿色荧光蛋白)。借助CRISPR-X同样可以进行耐药机制的研究:PSMB5是蛋白酶抑制剂硼替佐米的靶标,作者通过迭代进化发现了5个对硼替佐米强抗性突变与3个适度抗性突变。在TAM系统中(图3(d)),研究人员则在慢性髓性白血病细胞中用dCas9-AID-P182X (AIDx)靶标BCR-ABL基因,有效鉴定了赋予细胞伊马替尼抗性的已知突变和新突变[84]。除了耐药性研究,将碱基编辑工具与哺乳动物细胞展示系统结合使用,可以在多轮突变和选择下得到更高亲和力的抗体[97]。
单纯依靠CBE或ABE可能无法产生足够丰富的突变类型,而CBE和ABE的联合使用可以有效解决这一问题。Liu等[98]通过对编码OsACC羧基转移酶结构域基因设计141条sgRNA,结合两种CBE (eBE3和eCDA),及一种ABE(eABE),分别构建了3个碱基编辑文库。从eABE文库中获得了未报道过的I1879V突变,赋予水稻除草剂抗性,而另外两个文库未获得新的突变。碱基编辑器介导的基因进化技术(a Base-Editing-Mediated Gene Evolution,BEMGE)[99]则是将胞嘧啶碱基编辑器(r BE9)和腺嘌呤碱基编辑器(r BE14)同时转入水稻细胞,对水稻除草剂靶标基因Os ALS1进行靶向诱变,获得4个自然界中未曾被发现的、对除草剂双嘧菌钠具有抗性的蛋白变体。类似的策略同样被用于在放线菌的单碱基编辑。CRISPR-BEST (CRISPR-base Editing System)[87]包含两个独立的碱基编辑器(CRISPR-c BEST和CRISPR-a BEST),并在非模式链霉菌中通过引入终止密码子失活了奇霉素合成途径中两个拷贝的kir N基因,同时该研究借助Csy4(1类IF型核糖核酸内切酶)设计了sg RNA多重化系统,可以实现仅依靠一对启动子和终止子表达多个sg RNA,且每个sg RNA表达强度相同,显示出强大的用于代谢工程类定向进化的潜力。饱和靶向内源基因突变碱基编辑器(Saturated Targeted Endogenous Mutagenesis Editors,STEME)[88]则将CBE与ABE系统进行合并融合(图3(e)),通过将胞嘧啶脱氨酶(APOBEC3A)和腺嘌呤脱氨酶(ec Tad A-ec Tad A7.10)同时融合在n Cas9的N端,并将UGI融合至n Cas9 (D10A)的C端,实现只在一个sg RNA引导下就可以诱导靶位点C>T和A>G的同时突变,显着增加靶基因碱基突变的饱和度与多样性,最后验证STEME在植物中的定向进化能力,获得了Os ACC羧基转移酶结构域3个对氟吡甲禾灵具有抗性的新突变位点。
图3 Cas9与诱变蛋白联用介导的定向进化
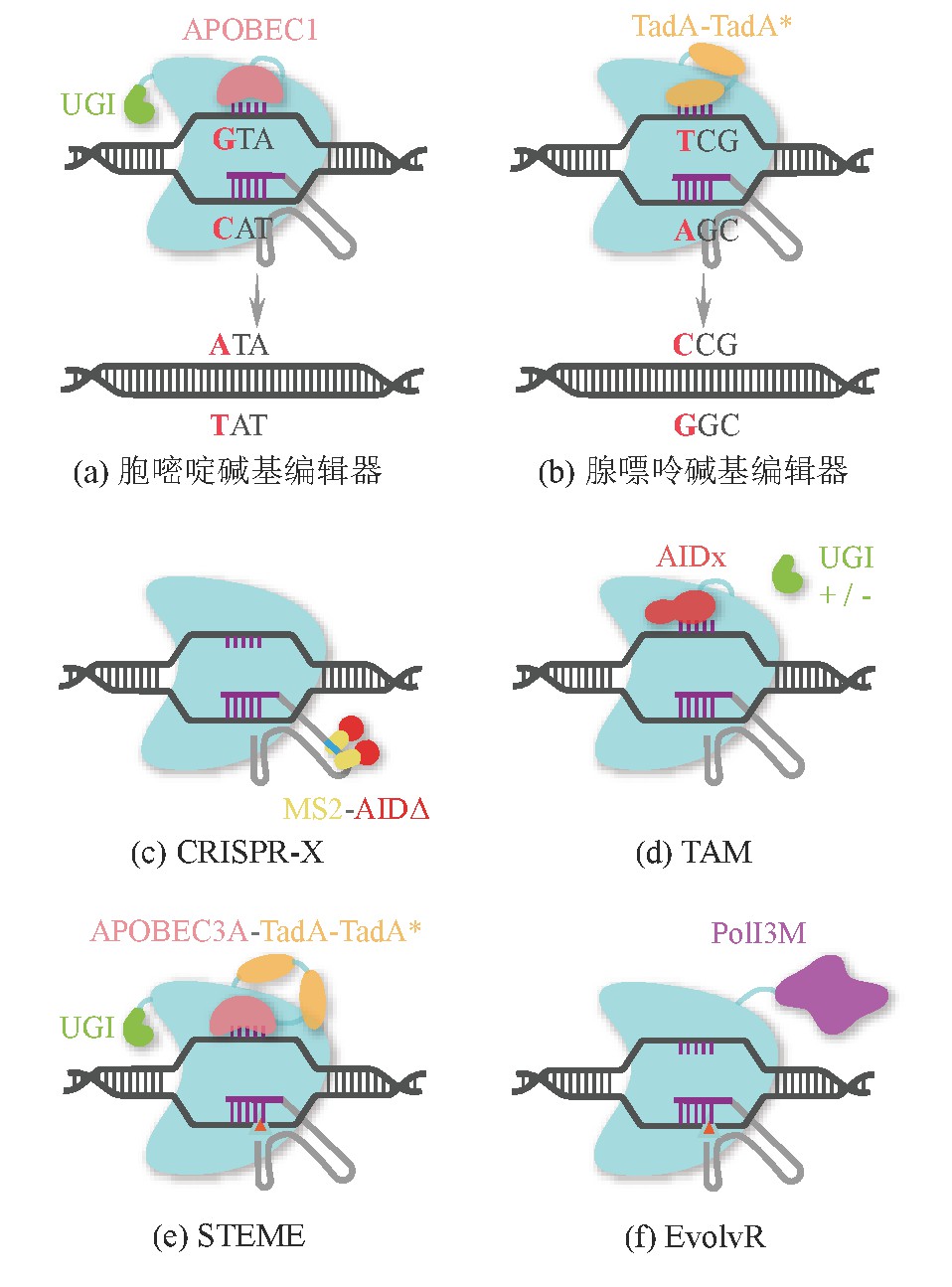
Fig.3 Cas9 and protein with mutation coupling mediated directed evolution
3.2.3、 Cas-DNA聚合酶定向进化技术
除了利用碱基编辑器进行单碱基的转换,借助CRISPR系统来招募其他诱变蛋白同样可以产生局部序列多样化。Evolv R系统[89]将n Cas9与易错DNA聚合酶I (Pol I3M)进行融合(图3(f)),利用n Cas9创建的DNA缺刻作为DNA聚合酶起始位点,诱变窗口长度、突变率和取代偏差则由聚合酶变体本身性质决定。该系统可以产生高于野生型细胞770多万倍的突变率,编辑350个核苷酸。最后使用Evolv R鉴定对抗生素壮观霉素耐药的新型核糖体突变位点。借助类似的原理,Tou等[100]在酿酒酵母中也开发了Evolv R系统。
4 、CRISPR介导的筛选与选择
对多样化突变文库进行筛选与选择是定向进化中另一个关键部分。筛选可以对每种突变体的特性进行评估,而选择则只会产生理想的突变体[101]。筛选技术可以减少错过所需突变体的机会,但降低通量;选择技术则可以用于更大的文库,但需要对突变文库施加选择压力来消除非目的突变体[102]。CRISPR系统除了具有出色的基因编辑能力外,还已应用于分子检测。而筛选和选择方法恰恰依赖分子检测等检测技术的开发,因为大多数生物分子与可直接观察的表型无关[96],需要荧光、比色或其他报告分子。例如,借助气相色谱检测产物的形成,获得具有高活性细胞色素P450酶变体[103],通过一连串酶促反应来产生与纤维素酶活相对应的荧光[104]等。目前,CRISPR系统被广泛地应用于核酸分子的检测[105,106],在2019年也出现了对非核酸靶标的检测系统,所以本节重点对CRISPR介导的筛选检测技术进行阐述,在未来展望部分则对CRISPR介导的选择进行讨论。
Liang等[107]开发了CRISPR-Cas12a和细菌变构转录因子(allosteric Transcription Factors,a T F s)介导的小分子检测平台C AT-S M e l o r(CRISPR-Cas12a-and a TF-mediated Small Molecule Detector),借助CRISPR-Cas12a的单链DNA切割能力与a TFs对小分子和双链DNA的竞争结合活性,当目的小分子存在时,a TF的构象发生改变,导致ds DNA从a TF结合域解离,ds DNA激活Cas12a对携带有荧光团-淬灭剂(Fluorophore Quencher,FQ)标记的ss DNA的切割,检测荧光信号发生变化,实现对尿酸和对羟基苯甲酸的纳摩尔水平的高通量检测。另一种CRISPR介导的传感器则是借助功能性DNA(functional DNA,f DNA)。当f DNA的靶标出现时,单链DNA激活剂与f DNA解聚,触发Cas12a对标记有FQ的单链DNA进行裂解,进而出现可检测的荧光信号,该策略也成功地用于检测ATP和Na+[108]。类似地,Li等[109]同样借助f DNA实现了对Pb2+、鲍曼不动杆菌和微小RNA的检测,再次扩充了CRISPR系统对非核酸分子的检测范围。
5 、总结与展望
CRISPR技术的出现和发展已经彻底改变人们对基因进行编辑、表达调控、分子检测的能力,出现了一系列基于CRISPR技术的拓展工具,这也促进定向进化技术的发展,并已应用于代谢工程、抗体工程、植物育种等领域(图4(a))。不同来源的CRISPR系统及其相关变体,已经可以较为有效地解决如PAM位点限制[110]、脱靶[111]等问题,为进一步多基因间协同进化(如人造基因回路或代谢通路)提供可能。利用大规模gRNA文库及高通量测序,结合DNA条形码技术,可以更高效地追踪与获得基因组层面上与突变相关的遗传信息,提高定向进化效率。同时通过机器学习与建立模型[112],有希望从获得的原始数据对进化模型进行完善,或通过逻辑线路设计实现对基因突变(如重复序列导致的缺失)的追踪与定量[113],得到普适性的系统突变库与目的性状的定量关系,帮助人们更好地借助CRISPR工具箱对定向进化进行操纵和预测。
借助CRISPR系统对DNA的切割活性或靶向活性,可以实现多样化突变文库的建立。在CRISPR介导的修复途径的诱变中,NHEJ和HDR途径产生的突变文库各有利弊,选择哪种途径要取决于宿主本身性质和研究需求。因此对修复途径进行更深入的研究,规避不同修复途径的劣势,对于CRISPR介导的定向进化非常重要。而对于Cas蛋白将诱变功能蛋白引入靶向位点的策略,可以避免出现双链断裂对宿主造成不利影响,实现对特定窗口核苷酸进行突变。另外,自2017年以来,Cas13系统也展示出巨大的应用潜力,其RNA的靶向能力将给研究者在RNA水平开发定向进化系统提供可能。与DNA文库相比,RNA文库多样性更强,但是如何将活性RNA突变体映射到DNA上并迭代遗传是其开发难点。
基于CRISPR的检测技术已经拓展到了多种小分子,这为开发新型筛选与选择系统提供了可能。如在选择方法的展示技术中,已提出的CRISPR展示系统可以构建结合有蛋白质、人工适体、随机序列库或长非编码RNA的Cas9蛋白复合物[114],在定向进化的高通量选择方法中具有广阔的应用前景。同时,借助Cas9对DNA的切割宿主致死性,可以进一步开发基于生存和生长的选择方法,而不是仅依靠药物或抗生素选择导致的淘汰,拓宽可研究表型。目前已有研究利用该性质进行微生物种群水平的选择性调控[115],以及结合致死基因,设计基于CRISPR的正负选择策略用于Cas9蛋白的定向进化[116]。由此可见,CRISPR技术在定向进化中筛选与选择策略上仍有很大的发展空间。
图4 CRISPR介导定向进化的应用与机遇
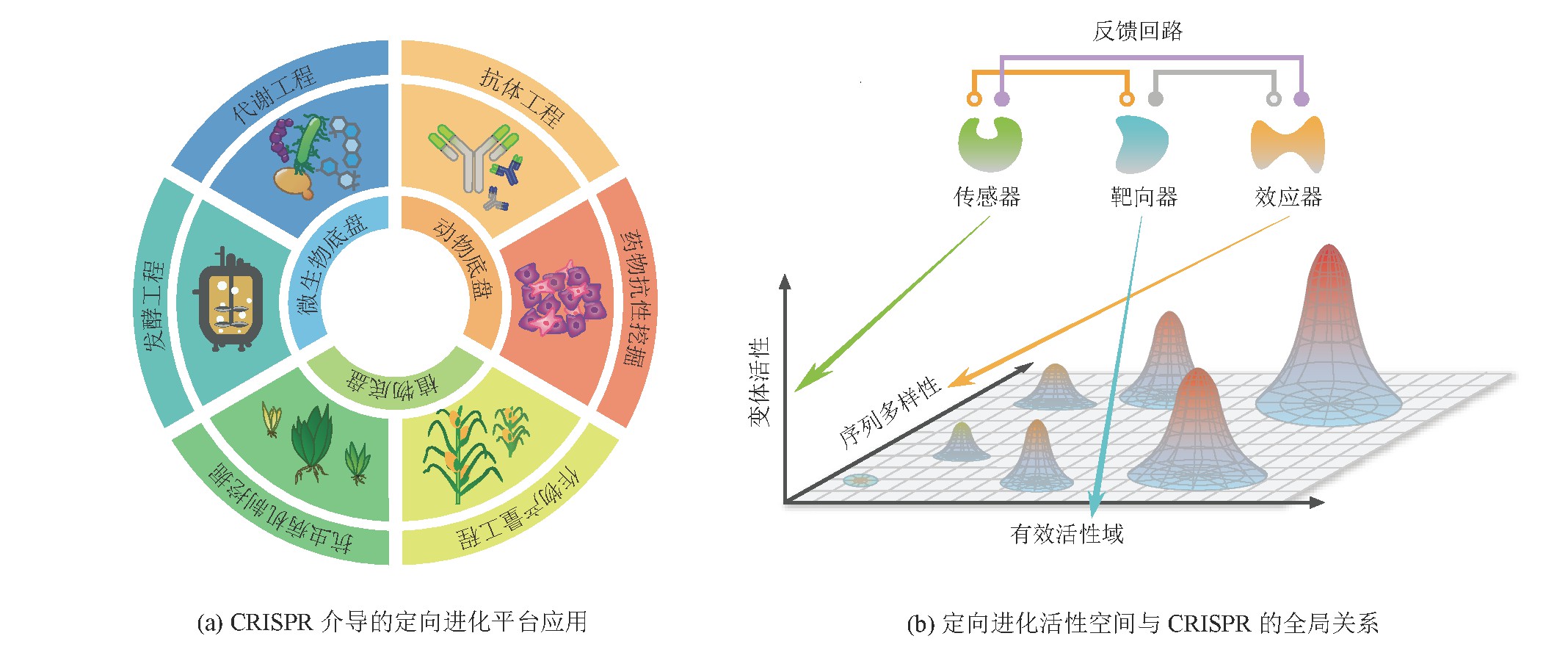
Fig.4 Application and opportunities of CRISPR-mediated directed evolution
CRISPR的多用途性,能更好地实现基因型-表型的联系,实现将建立突变文库和选择与筛选的偶联,并帮助已有定向进化技术进行改良。Cas蛋白作为模块化程度极高的核酸靶向平台,可以通过串联多种功能蛋白来满足定向进化的多种需求,实现更高的序列多样性、有效活性域可及性。Cas蛋白介导的有效活性域可及性将提高突变位点的精准性,致突变功能蛋白(如易错DNA聚合酶、脱氨酶等)将提高序列的多样性。二者共同决定了待进化分子的进化潜力。利用恰当的传感器对变体活性进行筛选,并开发基于传感器-靶向器-效应器之间反馈基因回路的新型可迭代定向进化技术(如当筛选系统检测到高活性变体在种群中占比较高时,线路会产生反馈,进而调节效应器与靶向器的效率,使得高活性变体在迭代过程中具有更小突变压力的同时,靶向器可对更广泛的位点进行探索),将是未来的发展方向之一(图4(b))。
未来,CRISPR技术将加速定向进化相关领域的发展,CRISPR工具盒将为人们提供多元化工具以解决定向进化中的问题并使定向进化技术设计进入全新的阶段。值得一提的是,也可以借助定向进化手段对CRISPR系统进行优化,而CRISPR技术又能改善定向进化策略,显示出二者潜在的紧密联系。
参考文献
[1]LiX,Song YJ,Century K,et al.A fast neutron deletion mutagenesis -based reverse genetics system for plants[J].The Plant Jourmal,2001,27():235-242.
[2Mittelsten Scheid 0,Afsar K,Paszkowski J.Release of epigenetic gene silencing by trans-acting mutations in Arabidopsis[J].Proceedings of theNational Academy of Sciences ofthe United States of America,1998, 95(2):632-637.
[3] Zhao HM,Chockalingam K. Chen ZL .Directed evolution of enzymes and pathways for industrial biocatalysis[J] Current Opinion in Biotechnology,2002,13<2):104-110.
[4] Johannes TW,Zhao HM. Directed evolution of enzymes and biosynthetic pathways[J].Current Opinion in Microbiology,2006,9(3).261-267.
[5] Butt H,Zaidi SEA,Hassan N,et al. CRISPR- based directed evolution for crop improvement[J].Trends in Biotechnology,2020,38(3):236-240.
[6] Makarova KS Haft DH,Barrangou R,et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Reviews Microbiology.2011,9(6):467-477.
[7] Koonin EV,Makarova KS ,Zhang F. Diversity, cassification and evolution of CRISPR-Cas systems[J]. Current Opinion in Microbiology,2017,37:67-78.
[8] Makarova KS,Wolf YI,Alkhnbashi OS,et al An updated evolutionary classification of CRISPR-Cas systems[J].Nature Reviews Microbiology,2015, 13(11):722-736.
[9] Shmakov S, Abudayyeh 00.Makarova KS,et al Discovery and functional characterization of diverse class 2 CRISPR-Cas systems[J] Molecular Cell,2015,60(3):385-397.
[10] Jiang FG, Doudna JA. CRISPR-Cas9 structures and mechanisms[J] Annual Review of Biophysics,2017,46(1);:505-529.
[11] Jinek M.Chylinski K,Fonfara I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science,2012,337(6096):816-821.
[12] Cong L,Ran FA,Cox D,et al.Mutiplex genome engineering using CRISPR/Cas systems[J]. Science ,2013,339(6121):819-823.
[13] Mali P,Yang LH,Esvelt KM,et al RNA-guided human genome engineering via Cas9[J] Science 2013,339(6121):823-826.
[14] Li JF,Norville JE ,Aach J,et al.Multiplex and homologous recombination-mediated genomeediting in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J].Nature Biotechnology,2013,31(8):688-691.
[15] Nekrasov V,Staskawicz B,Weigel D,et al.Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease[J].Nature Biotechnology,201
3,31(8):691-693.
[16] Ran FA,Hsu PD,Lin CY,et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing speicitylJLC.ll.2013, 154(6): 1380-1389.
[17] Mali P,Aach J,Stranges PB,et al. CAS9transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering[J] Nature Biotechnology,.2013,31(9):833-838.
[18] Gilbert LA,Larson MH,Morsut L,et al. CRISPRmediated modular RNA-guided regulation of transcription in eukaryotes[J]Cll.201.1542).442-451.
[19] Dominguez AA,Lim WA,Qi LS Beyond editing.repurposing CRISPR-Cas9 for precision genome regulation and interrogation[J.Nature Reviews Molecular Cell Biology,2016,17(1):5-15.
[20] Nishimasu H,Cong L,Yan WX,et al. Crystal structure of Staphylococcus aureus Cas9[J] Cell,2015,162(5)1113-1126.
[21]Friedland AE ,Baral R. Singhal P,et al. Characterization of Staphylococcus aureus Cas9:a smaller Cas9 for ll-in-one adeno-associated virus delivery and paired nickase applications[J]. Genome Biology,2015, 16(1):257.
[22]Hou ZG, Zhang Y,.Propson NE ,et al.Eficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis[J] Proceedings of the National Academy of Sciences of the United States of America,2013,110(39):15644-15649.
[23] Karvelis T,Gasiunas G,Miksys A,et al .crRNA and tracrRNA guide Cas9-mediated DNA interference in Streptococcus thermophilus[J] RNA Biology,2013,10(5):841-851.
[24] Kim E,Koo T,Park SW.et al.ln vivo genomeediting with a small Cas9 orthologue derived from Campylobacter jejuni[J] Nature Communications,2017,8(1):14500.
[25] Chatterjee P,Jakimo N,Jacobson JM.Minimal PAM specificity of a highly similar SpCas9ortholog[J] Science Advances,2018 .4(10):eaau0766.
[26] Kleinstiver BP,Prew MS,Tsai SQ,et al. Engineered CRISPR-Cas9 nucleases with altered PAM speifcitie[J.Nature ,2015,523(7561):481-485.
[27] Hu JH,Miller SM,Geurts MH,et al. Evolved Cas9variants with broad PAM compatibility and high DNA specificity[J].Nature,2018,556(7699;:57-63.
[28] Nishimasu H,Shi X,Ishiguro S,et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space[J] Science ,2018,361(6408):1259-1262.
[29] Miller SM.Wang T,Randolph PB,et al. Continuous evolution of SpCas9 variants compatible with non-G PAMs[J]. Nature Biotechnology ,2020.38(4):471-481.
[30] Walton RT,Christie KA,Wittaker MN,et al.Unconstrained genome targeting with nearPAMless engineered CRISPR-Cas9 variants[J]. Science,2020 ,368(6488):290-296.
[31] Slaymaker IM,Gao LY,Zetsche B,et al.Ratinally engineered Cas9 nucleases with improved specificity[J] Science,2016.351(6268):84-88.
[32] Nihongaki Y,Kawano F,Nakajima T,et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing[J] Nature Biotechnology,2015,33(7):755-760.
[33] Zetsche B,Volz SE ,Zhang F.A split-Cas9architecture for inducible genome editing and transcription modulation[J] Nature Biotechnology,2015,33(2):139-142.
[34] Gao YC,Xiong X,Wong S,et al. Complex transcriptional modulation with orthogonal and inducible dCas9 regulators[J.Nature Methods,2016,13(12)-1043-1049.
[35] Kim D,Kim J.Hur JK.et al. Genome-wide analysis reveals specificities of Cpfl endonucleases inhuman cells[J].Nature Biotechnology.2016,34(8)-863-868.
[36] Zetsche B,Gootenberg JS. Abudayyeh 00,et al. Cpfl is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J] Cll,.2015,163(3):759-771.
[37] Gao LY,Cox DBT,Yan WX,et al. Engineered Cpfl variants with altered PAM speificities[J. Nature Biotechnology,2017,35(8):789-792.
[38] Kleinstiver BP,Sousa AA,Walton RT,et al. Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene ,epigenetic and base editing[J].Nature Biotechnology,2019,37(3):276-282.
[39]Yang H.Gao P.Rajashankar KR.et al. PAMdependent target DNA recognition and cleavage by C2c1 CRISPR-Cas endonuclease[J]. Cll,2016.167(7):1814-1828.e12.
[40]Teng F,Cui TT,Feng GH,et al.Repurposing CRISPR-Cas 12b for mammalian genome engineering[J].Cell Discovery,2018,4(1):63.
[41] Ming ML,Ren QR,Pan CT,et al. CRISPR-Cas12b enables eficient plant genome engineering[J.Nature Plants,2020,6(3):202-208.
[42] Liu JJ,Orlova N,Oakes BL,et al.CasX enzymes comprise a distinct family of RNA-guided genome editors[J].Nature .2019,5667743):218-223.
[43] Burstein D,Harrington LB Strutt SC,et al New CRISPR-Cas systems from uncultivated microbes[J]. Nature ,2017 ,542(7640):237-241.
[44] Yan WX,Hunnewell P.Alfonse LE,et al.Functionally diverse type V CRISPR-Cas systems[J]. Science,2019,363(6422)-88-91.
[45] Kakarougkas A,Jeggo PA.DNA DSB repair pathway choice: an orchestrated handover mechanism[J] The British Journal of Radiology,2014.87(1035):20130685.
[46] Hefferin ML.Tomkinson AE. Mechanism of DNA double- strand break repair by non-homologous end joining[J] DNA Repair,2005,4(6):639-648.
[47] Bibikova M,Golic M,Golic KG,et al.Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases[J] .Genetics ,2002,161(3):1169-1175.
[48] Capecchi MR. Altering the genome by homologous recombination[J] Science, 1989,244(4910):1288-1292.
[49] Chu VT,Weber T,Wefers B,et al.ncreasing the fficiency of homology-directed repair for CRISPRCas9-induced precise gene editing in mammalian ells[J] Nature Biotechnology,.2015,33(5):543-548.
[50]Nambiar TS Billon P,Diedenhofen G,et al. Stimulation of CRISPR-mediated homologydirected repair by an engineered RAD 18 variant[J]. Nature Communications ,2019,10(1);.3395.
[51] Baker O,Tsurkan S,Fu J,et al.The contribution of homology arms to nuclease -assisted genome engineering[J] Nucleic Acids Research,2017,45(13):8105-8115.
[52]Li H,Beckman KA,Pessino V,et al.Design and specificity of long ssDNA donors for CRISPRbased knock-in [Z/OL].bioRxiv,2017.0dittps /oi. org/10. 1101/178905.
[53] Jiang WY,Bikard D,Cox D,et al.RNA- guided editing of bacterial genomes using CRISPR-Cas systems[J] Nature Biotechnology.2013.313):233-239.
[54] Wang HH,Isaacs FJ,Carr PA, et al.Programming cells by multiplex genome engineering and accelerated evolution[J].Nature,2009 ,460(7257):894-898.
[55] Ronda C.Pedersen LE. Sommer MOA.et al. CRMAGE:CRISPR optimized MAGE recombineering[J] Scientific Reports,2016,6(1):19452.
[56] Li YF,Lin ZQ,Huang C.et al. Metabolic engineering of Escherichia coli using CRISPRCas9 meditated genome editing[J]. Metabolic Engineering,2015,31:13-21.
[57] Garst AD,Bassalo MC ,Pines G,et al. Genomewide mapping of mutations at single- nucleotide resolution for protein,metabolic and genome engineering[J].Nature Biotechnology,2017,35(1):48-55.
[58] Liu RM,Liang LY,Garst AD,et al Directedcombinatorial mutagenesis of Escherichia coli for complex phenotype engineering[J.Metabolic Engineering.2018,47:10-20.
[59] Liu RM,Liang LY, Choudhury A,et al.terative genome editing of Escherichia coli for3-hydroxypropionic acid production[J] Metabolic Engineering.2018,47:303-313.
[60]Ryan 0W, Skerker JM,Maurer MJ.et al Selection of chromosomal DNA liraries using a multiplex CRISPR system]J]eLife ,2014.3.e03703.
[61] Jakocinnas T.Pedersen LE,Lis AV,et al. CasPER,a method for directed evolution in genomic contexts using mutagenesis and CRISPR/Cas9[J].Metabolic Engineering,2018,48.288 -296.
[62] Bao ZH,HamediRad M,Xue P,et al. Genomescale engineering of Saccharomyces cerevisiae with single -nucleotide precision[J] Nature Biotechnology,2018,36(6):505-508.
[63]Neggers JE Kwanten B, Dierckx T,et al.Target identification of small molecules using large-scale CRISPR-Cas mutagenesis scanning of essential genes[J].Nature Communications,2018,9(1):502.
[64] Jakocinas T,Bonde l,Herrgard M,et al. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae[J] Metabolic Engineering,2015,28.213-222.
[65] Guo XG,Chavez A,Tung A,et al High-throughput creation and functional profiling of DNA sequence variant libraries using CRISPR-Cas9 in yeast[J] Nature Biotechnology,2018,36(6):540-546.
[66] Roy KR,Smith JD,Vonesch SC, et al.Multiplexed precision genome editing with trackable genomic barcodes in yeast[J.Nature Biotechnology.2018.36(6):512-520.
[67] Lian JZ Schultz C,Cao MF,et al Multi-functional genome. wide CRISPR system for high throughput genotype phenotype mapping[J].Nature Communications,2019,10(1):5794.
[68] Dicarlo JE ,Norville JE ,Mali P,et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J]. Nucleic Acids Research,2013,41(7):4336-4343.
[69] Kim BH,Zhang GJ. Generating stable knockoutZebrafish lines by deleting large chromosomal fragments using multiple gRNAs[J].G3,2020, 10(3): 1029-1037.
[70] Lee H,Kim JI,Park JS,et al. CRISPR/Cas9-mediated generation of a Plac8 knockout mouse model[J].Laboratory Animal Research,2018,34(4)-279-287.
[71] Shalem O, Sanjana NE .Hartenian E,et al. Genomescale CRISPR-Cas9 knockout screening in human ells[J. Science,2014,343(6166)-84-87.
[72]Hahn F,Mantegazza O, Greiner A,et al.An eficient visual screen for CRISPR/CAS9 activity in Arabidopsis thaliana[J] Frontiers in Plant Science,2017,8:39.
[73] Zhou HB,Liu B,Weeks DP,et al.Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice[J] Nucleic Acids Research,2014,42(17):10903-10914.
[74]Butt H,Eid A,Momin AA.et al. CRISPR directed evolution of the spliceosome for resistance to splicing inhibitors[J]. Genome Biology ,2019,20(1):73.
[75]psaro JJ,Shen C. Aral E,et al. Rapid generation of drug-esistance aleles at endogenous loci using CRISPR-Cas9 indel mutagenesisl[J.PLoS One,2017,12(2):e0172177.
[76] Findlay GM,Boyle EA,Hause RJ,et al. Saturation editing of genomic regions by multiplex homologydirected repair[J.Nature ,2014,513(7516):120-123.
[7] Mason DM,Weber CR,Parola C, et al.Highthroughput antibody engineering in mammalian cells by CRISPR/Cas9-mediated homology-directed mutagenesis[J.Nucleic Acids Research,2018, 46(14):7436-7449.
[78] Parola C ,Neumeier D,Friedensohn S,et al. Antibody discovery and engineering by enhanced CRISPR-Cas9 integration of variable gene cassette libraries in mammalian cells[J].mAbs,2019.11(8):1367-1380.
[79] Hess GT,Tycko J,Yao D,et al. Methods and applications of CRISPR- mediated base editingin eukaryotic genomes[J]. Molecular Cell,2017,68(1):26 -43.
[80] Komor AC,Kim YB,Packer MS .et al. Programmable editing of a target base in genomic DNA without double -stranded DNA cleavage[J.Nature,2016 ,533(7603):420-424.
[81] Gaudelli NM,Komor AC ,Rees HA,et al Programmable base editing of A-T to G-C in genomic DNA without DNA cleavage[J]. Nature,2017,551(7681):464-471.
[82] Nishimasu H,Ran FA,Hsu PD,et al.Crystal structure of Cas9 in complex with guide RNA and target DNA[J]Cell,2014, 156(5):935-949.
[83] Hess GT,Fresard L,Han K,et al. Directed evolution using dCas9- targeted somatic hypermutation in mammalian cells[J]. Nature Methods ,2016,13(12):1036- 1042.
[84] Ma YQ,Zhang JY,Yin WJ,et al. Targeted AIDmediated mutagenesis(TAM)enables fficient genomic diversification in mammalian ells[J] Nature Methods,2016,13(12):1029-103
[85] Shimatani Z,Kashojia S, Takayama M.et al. Targeted base editing in rice and tomato using a CRISPR- Cas9 cytidine deaminase fusion[J] Nature Biotechnology.2017,35(5):441-443.
[86]Zong Y,Wang YP,Li C,et al.Precise base editing in rice,wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nature Biotechnology,2017.35(5):438-440.
[87] Tong YJ,Whitford CM,Robertsen HL,et al.Highly eficient DSB-free base editing for streptomycetes with CRISPR-BEST[J] Proceedings of the National Academy of Sciences of the United States of America,2019, 116(41):20366-20375.
[88] Li C,Zhang R,Meng XB,et al. Targeted,random mutagenesis of plant genes with dual cytosine and adenine base editors[J]. Nature Biotechnology,2020 ,38(7):875-882.
[89] Halperin SO,Tou CJ,Wong EB,et al. CRISPRguided DNA polymerases enable diversification of all nucleotides in a tunable windowlJ.Nature,2018,560(7717).248-252.
[90] Kim YB,Komor AC,Levy JM,et al.Increasingthe genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions[J].Nature Biotechnology,2017,35(4):371-376.
[91] Li XS,Wang Y,Liu YJ,et al Base editing with a Cplil-cytidine deaminase fusion[J].Nature Biotechnology,2018,36(4):324-327.
[92]Yan F,Kuang Y,Ren B,et al.Highly eficient A:T to GC base editing by Cas9n-guided tRNA adenosine deaminase in rice[J]. Molecular Plant,2018.11(4):631-634.
[93] Kang BC,Yun JY,Kim ST,et al.Precision genome engineering through adenine base editing in plants[J.Nature Plants ,2018,4(7):427-431.
[94] Li C,Zong Y,Wang YP,et al.Expanded base editing in rice and wheat using a Cas9-adenosine deaminase fusion[J] Genome Biology, 2018,19(1):59.
[95] Wang FP,Zhang CW,Xu W,et al. Developing high-afficiency base editors by combining optimized synergistic core components with new types of nuclear localization signal peptide[J].The Crop Journal,2020 ,8(3):408-417.
[96] Packer MS,Liu DR. Methods for the directed evolution of proteins[J]. Nature Reviews Genetics,2015,16(7):379-394.
[97] Liu LD,Huang M,Dai PF,et al.Intrinsic nucleotide preference of diversifying base editors guides antibody ex vivo afinity maturation[J.Cell Reports .2018,25(4):884-892. e3.
[98] Liu XS,Qin RY,Li J,et al.A CRISPR-Cas9-mediated domain-specific base- editing screen enables functional assessment of ACCase variants in rice[J]. Plant Biotechnology Journal,2020,18(9):1845-1847.
[99]Kuang Y,Li S,Ren B,et al. Base-editing-mediated artfcial evolution of OsALS1 In Planta to develop novel herbicide-tolerant rice germplasms[J].Molecular Plant,2020,13(4):565-572.
[100] Tou CJ,Schaffer DV,Dueber JE. Targeted diversification in the S. cerevisiae genome with CRISPR- guided DNA polymerase J] .ACS Synthetic Biology,2020,9(7):1911-1916.
[101] Leemhuis H,Kelly RM,Dijkhuizen L Directed evolution of enzymes:library screening strategies[J]. IUBMB Life 200.613)22228.
[102]Xiao H,Bao ZH,Zhao HM.High throughput screening and selection methods for directed enzyme evolution[J].Industrial and Engineering Chemistry Research,2015,54(16):4011-4020.
[103] Coelho PS ,Brustad EM,Kannan A,et al Olefin cyclopropanation via carbene transfer catalyzed by engineered cytochrome P450 enzymes[J] Science,2013,339(6117):307-310
[104] Ostafe R,Prodanovic R,Ung WL,et al.A highthroughput ellulase screening system based on droplet microfluidics[J]. Biomicro fluidics .2014,8(4):041102.
[105] Kellner MJ,Koob JG Gootenberg JS, et al SHERLOCK:.nucleic acid detection with CRISPR nucleases[J]. Nature Protocols,2019,14(10):2986-3012.
[106] Chen JS,Ma E,Harrington LB,et al.CRISPRCas12a target binding unleashes indiscriminate single -stranded DNase activity[J] Science,2018,360(6387):436-439.
[107] Liang MD,Li ZL,Wang WS,et al.A CRISPRCas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules[J].Nature Communications .2019,10(1):3672.
[108]Xiong Y,Zhang JJ,Yang ZL,et al.Functional DNA regulated CRISPR-Cas 12a sensors for pointof-care diagnostics of non-nucleic-acid targets[J]. Journal of the American Chemical Society ,2020,142(1):207-213.
[109] Li JJ,Yang sS, Zuo C, et al.Applying CRISPRCas 12a as a signal amplifier to construct biosensors for non-DNA targets in ultralow concentrations[J].ACS Sensors,2020,5(4):970-977.
[110]Ma DC,Xu ZM,Zhang ZY,et al. Engineer chimeric Cas9 to expand PAM recognition based on evolutionary information[J] Nature Communications,2019,10(1):560.
[111]Liu Q,He D,Xie L .Prediction of off-target specificity and cells-pecific fitness of CRISPRCas system using attention boosted deep learning and network-based gene feature[J].PLoS Computational Biology,2019,15(10):e1007480.
[112] Benes D,Rodriguez-Paton A, Sosik P. Directed evolution of biocircuits using conjugative plasmids and CRISPR-Cas9.design and in silico experiments[J] Natural Computing,2017.16(3):497-505.
[113] Huang YJ,Yang S, Chen WH,et al.A synthetic genetic circuit enables precise quantification of direct repeat deletion in bacteria[J].ACS Synthetic Biology,2020.9(5):1041-1050
[114] Shechner DM,Hacisuleyman E, Younger ST,et al.Multiplexable ,locus. specific targeting of long RNAs with CRISPR- display[J] Nature Methods,2015,12(7):664-670.
[115] Hamilton TA,Pellegrino GM,Therrien JA.et al. Eficient inter-species conjugative transfer of a CRISPR nuclease for targeted bacterial klling[J.Nature Communications,2019,10(1):4544.
近年来随着人类基因组测序的完成和全基因组测序技术的不断发展,复杂基因组中未知基因功能的探索对生物学的定点研究、临床医学及基因治疗都有着不可替代的作用。长期以来,基于同源重组机制的基因敲除技术已在基因功能研究中被广泛采用,但是该技术在实际工...
随着基因工程技术的不断发展,基于核酸酶的基因编辑技术得以开发利用,其原理是通过核酸酶特异性地识别并切割靶DNA双链,激发细胞内源性的修复机制,从而实现基因定向改造[。...
基因编辑技术是分子生物学研究中的重要手段,通过对目的片段序列的改变,从而影响基因的功能。它的出现推进分子生物学的进程,提高了对于基因结构功能的研究效率。...
CRISPR-Cas系统是细菌、古细菌对抗病毒进化出来的免疫机制,2012年,首次将CRISPR-Cas9系统成功应用于大肠杆菌基因组编辑,随后这一系统被广泛应用到哺乳动物和人类细胞中,从此基因编辑进入了新时代。...
基因定点敲除技术通过对染色体上基因特异位点进行定点突变,使其功能丧失,进而达到研究其功能的目的。一些利用基因敲除技术创建的动物疾病模型对医学研究的发展做出了重要贡献。传统基因敲除技术于上世纪80年代末由Smithies等[1]创立,通过同源重组手段...