
宿主消化道中生存着大量的微生物, 经过长期的共进化, 这些微生物与宿主之间形成了相对稳定的互利共生关系。一方面, 宿主消化道为各种微生物提供相对稳定的生存环境和持续的营养物质供应; 另一方面, 消化道微生物也直接或间接参与宿主的发育、营养、免疫等诸多生理生化过程。
近年来, 食物组成对消化道微生物群落结构和功能的影响成为研究热点。如 Li 等通过对受试人员食谱中添加十字花科蔬菜发现其显著影响了人类粪便样品中的微生物群落结构。Kocherginskaya 等通过使用两种不同成分的饲料喂养公牛探究其瘤胃中细菌类群变化, 研究结果表明两种饮食条件下公牛瘤胃内微生物群落差异显著, 饲喂玉米的公牛瘤胃中的微生物在数量和种类上都明显高于饲喂干草的公牛瘤胃中的微生物。不仅在陆生脊椎动物, 在水生脊椎动物鱼类中同样有相似的研究报道, 如Ring等以 5 种不同食物组成的饲料对大西洋鲑鱼进行为期 28d 的投喂实验, 对其中肠和后肠的微生物菌群进行分析, 结果显示不同饮食条件下大西洋鲑的肠道菌群结构存在差异。McKellep 等对大西洋鲑分别投喂含有大豆饼、菊粉和氧四环素的饲料, 结果显示后肠中附着菌群存在差异, 其中投喂大豆饼组鱼体后肠内附着菌群不仅数量上更多, 而且具有更高的菌群结构多样性。以上研究结果表明无论在陆生脊椎动物还是水生脊椎动物, 宿主食物组成都是影响其消化道微生物群落的重要因素。在水生脊椎动物中, 草食性鱼类以其能以陆生或水生植物为主要食物来源而成为重要研究对象, 本文以淡水草食性鱼类为研究对象, 探索这一特殊食性鱼类的消化道微生物菌群结构。
草鱼(Ctenopharyngodon idellus)和团头鲂(Me-galobrama amblycephala) 为典型的草食性鱼类,且草鱼是目前世界上产量最大的淡水经济鱼类团头鲂是我国特有的优良淡水鱼类, 它们不仅在我国淡水养殖中占据重要地位, 在维持淡水生态系统的平衡方面也有重要作用。本研究以湖泊天然水体养殖的草鱼(Cteno-pharyngodon idellus)和团头鲂(Megalobrama ambly-cephala)为研究对象, 采用 PCR-DGGE 指纹分析结合 DNA 测序技术比较分析其消化道微生物群落结构特征, 希望能为深入阐述草食性鱼类消化道内特定微生物菌群参与营养物质代谢提供参考。
1、 材料与方法
1.1 采样点概况
武汉市武湖(N 30°47?, E 114°28?), 位于湖北省武汉市黄陂区境内, 为长江中游中型浅水富营养化湖泊, 湖区面积约 20 km2, 最大水深 4.2 m, 平均水深 2.6 m。水生植被覆盖率为 30%—40%, 据夏文凯等研究, 武湖水生维管束植物主要有 4 科 5 种,沉水植物主要有聚草(Myriophyllum spicatum)、苦草(Vallisneria spiralis)、金鱼藻(Ceratophyllum demersum)、菹草(Potamogeton crispus)。
1.2 样品采集
本实验所用草鱼、团头鲂于 2012 年 11 月 21 日采自武汉市武湖(表 1), 选取大小相近个体各三条,活鱼充氧运回实验室后立即在无菌条件下进行解剖,分别剪取前、中、后肠, 用无菌水冲洗肠内容物并收集于 2 mL 无菌离心管中。
1.3 肠道微生物总 DNA 提取
草鱼和团头鲂消化道微生物中 DNA 提取参考Ni 等, 即向每管肠道内容物中加入 1200 ?L 裂解液(10 mmol/L Tris-Cl; 0.5% SDS; 100 mmol/L EDTA;0.1 mg/mL 蛋白酶 K; 50 μg/mL RNase A), 于 55℃中水浴裂解 12h; 室温离心后取上清转入新无菌离心管中采用常规酚-氯仿法进行抽提, 随后用 4℃保存的70%酒精洗涤 3 次后自然风干, 风干后的 DNA 重悬于100 ?L 无菌双蒸水中, 保存于–20℃备用。
1.4 PCR 扩增
采用细菌16S rRNA基因通用引物F357-GC (5?-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAGCAG-3?)和 R518(5?-ATTACCGCGGCTGCTGG-3?)对肠道内容物样品中细菌类群进行 PCR-DGGE 分析。PCR 反应体系为每 25 ?L 反应混合液包含: 2.5 U Taq DNA 聚合酶(Fermentas, 美国), 80 ?mol/LdNTP (Fermentas,美国), 2 mmol/L MgCl2(Fermentas, 美国), 正反向引物各 0.2 μmol/L, 1 × buffer (Fermentas, 美国),细菌 DNA 模版 2 μL。PCR 反应在 S1000TM热循环仪(Bio-Rad, 美国)上进行, 反应条件为: 94℃预变性10min; 随后进行 10 个循环的降落 PCR (94℃变性45s, 68—59℃退火 30s, 每个循环降低 1 ℃ , 72℃延伸 1min); 然后再进行 25 个循环的常规 PCR 扩增(94℃变性 45s, 58℃退火 30s, 72℃延伸 1min); 最后72℃延伸 10min。取 5 ?L PCR 产物用浓度为 1.5%的琼脂糖胶 100 V 稳压电泳 30min 以检测 PCR 扩增效果。
1.5 变性梯度凝胶电泳(DGGE)分析
PCR 扩增所得产物在 9%(w/v)胶浓度的聚丙烯酰胺凝胶上进行电泳, 变性范围为 40%—70%, 电泳条件为 60℃条件下 120 V 预电泳 10min, 然后 100V 稳压电泳 12h。凝胶用 1 × SYBR Gold 染色 30min后在 BIO-RAD Gel Doc XR+凝胶成像系统(Bio-rad,美国)下成像。
1.6 测序
从变性梯度凝胶上回收需要测序的条带, 加100 ?L 无菌水溶解, 取 2 ?L DNA 模版用不带 GC夹板的引物 F357 和 R518 进行 PCR 扩增, PCR 扩增条件同上, PCR 产物用 DNA 凝胶回收试剂盒(Axygen, 美国)进行回收纯化, 纯化后的 DNA 片段用 pMD18-T 载体(TaKaRa, 日本)转化入 Escherichiacoli DH5α 菌株培养后进行 PCR 检测, 合格的菌斑送北京诺赛基因组研究中心有限公司进行测序。
1.7 数据分析
采用 Quantity One 4.6.2(Bio-Rad, 美国)软件导出变性梯度凝胶电泳图谱条带丰度数据矩阵。借助XLSTAT-Pro 7.5 软件依据条带丰度值做 UPGMA 聚类分析, 并运用 canoco4.5 软件对数据进行 PCA 分析。测序所得序列运用 NCBI 中 VecScreen 工具除去载体, 再次去除引物后将所得序列结果在 RDP 数据库中进行比对。运用 ClustalX(version 1.83)和 MEGA 安装包(4.0)构建系统发生树, 采用邻接法(NJ)进行分析, 并进行 1000 次重复的 bootstrap 验证。
2、结果
2.1 DGGE 指纹图谱
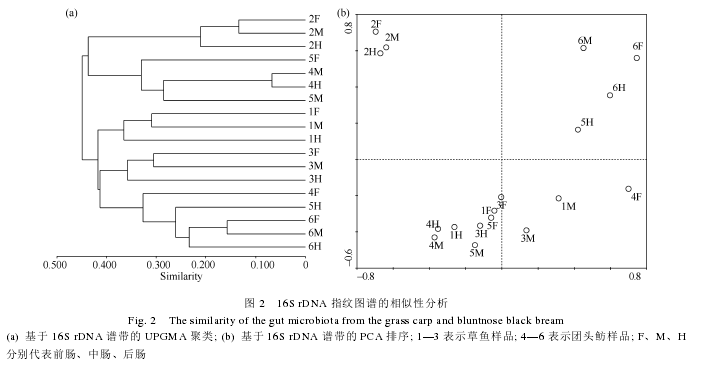
针对细菌 16S rRNA 基因进行的 DGGE 图谱分析共检测到 91 种 DGGE 带型(图 1)。其中有 30 条谱带仅在单个泳道检出, 没有检测到在所有泳道都出现的共有条带。草鱼消化道内检测到的平均谱带数为(33.3±2.8)条, 团头鲂消化道检测到的平均谱带数为(38.0±2.5)条。
2.2 消化道微生物群落结构比较
UPGMA 聚类分析显示草鱼消化道微生物群落并没有完全与团头鲂消化道微生物群落分开; 相比于草鱼和团头鲂消化道微生物群落之间的差异, 草鱼和团头鲂个体间差异更加明显, 而且草鱼消化道微生物群落的个体分化更为明显, 如 1 号草鱼样品、2 号草鱼样品、3 号草鱼样品分别聚为不同的聚类枝,而团头鲂则只有 6 号样品聚为一支, 并嵌入到 4 号前肠和 5 号后肠聚的进化枝中; 4 号团头鲂其他样品与 5 号团头鲂其他样品则没有很好地分开。
PCA 排序结果同样显示除了 1 号草鱼样品和 6 号团头鲂样品与其他样品不同之外, 其他的样品都没有很好地区分开, 这一结果同样说明草鱼和团头鲂消化道微生物群落结构并没有明显地分开。
2.3 DGGE 谱带测序分析
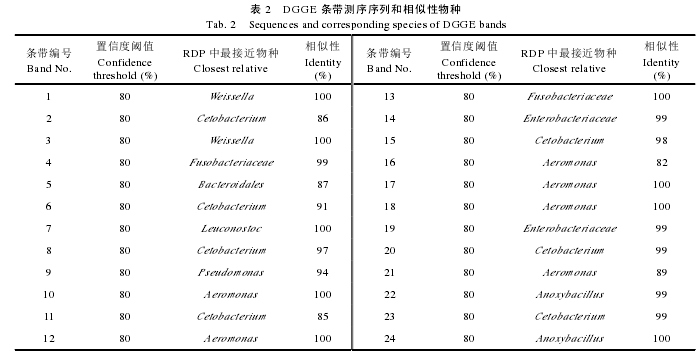
本实验一共测得 24 条 DGGE 条带对应的 DNA序列(表 2)。将所得序列进行 RDP 数据库比对, 置信度阈值为 80%。结果显示草鱼消化道内菌群主要是 γ-变形菌门 (γ-Proteobac-teria)、梭杆菌门(Fusobacteria)还有少量厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes); 团头鲂消化道内菌群与草鱼相似, 主要为 γ-变形菌门、梭杆菌门和厚壁菌门, 但未检测到拟杆菌门细菌。其中, 气单胞菌属和 Cetobacterium 属细菌是草鱼和团头鲂消化道中的优势菌群; 另外, 团头鲂消化道内还检测到魏斯氏菌属、明串珠菌属和假单胞菌属细菌,但未检测到拟杆菌门细菌。同时结合 RDP 和 NCBI数据库比对结果运用 MEGA 安装包(4.0)构建 NJ 系统发生树。
3、 讨论
宿主消化道内生活着大量的微生物, 就人类而言, 消化道内微生物细胞在数量上至少是人类体细胞数量的十倍。这些微生物生态系统在宿主体内发挥着重要的生理功能。目前为止, 国内外学者已对动物消化道微生物开展了广泛深入的研究, 如Rawls 等以无菌斑马鱼为模型研究其消化道微生物时发现, 斑马鱼消化道内微生物能够调节肠道212 个基因的表达, 且其中一部分能够刺激上皮细胞增殖、促进营养物质代谢和先天性免疫应答。尽管目前已经证实消化道微生物是维持宿主健康不可或缺的因素, 但消化道内微生物群落结构并不是固定不变的, 宿主的食物组成、生活环境、健康状况等因素都会影响宿主的消化道微生物群落结构,而食物组成对宿主消化道微生物群落结构的影响目前已开展了广泛的研究, 并证实宿主的食性和食物组成会对群落结构造成明显的影响, 如 Ring等对北极嘉鱼(Salvelinus alpinus L.)分别投喂等比例替代的糊精和菊粉饲料, 研究结果表明投喂两种食物的北极嘉鱼后肠中微生物组成存在差异, 投喂糊精组鱼体后肠中优势菌群为葡萄球菌属(Staphyloco-ccus)、假单胞菌属 (Pseudomonas)、微球菌属 (Mi-crococcus)、水栖嗜冷杆菌(Psychrobacter glacincola)和链球菌属(Streptococcus), 而投喂菊粉组优势菌群为葡萄球菌属(Staphylococcus)、链球菌属(Strepto-coccus)、肉杆菌属(Carnobacterium)和芽孢杆菌属(Bacillus)。Syvokien和 Mick?nien?对波罗的海沿岸的 10 种海洋鱼类的消化道微生物群落进行周年跟踪研究, 数据显示不同食性鱼类之间的肠道微生物菌群存在显著差异。Ward 等通过 16S rDNA 克隆文库方法对南极地区南极鱼科的杂食性鱼类Notothenia coriiceps 和专一肉食性鱼类 Chaenoce-phalus aceratus 消化道微生物群落进行分析, 结果显示杂食性的N. coriiceps肠道微生物多样性比肉食性的 C. aceratus 丰富。这些研究结果证实了宿主食性和食物组成是影响其消化道微生物群落结构的重要因素。颜庆云等对以浮游生物为主要食物来源的鲢(Hypophthalmichthys molitrix)、鳙(Aristichthysnobilis)肠含物进行 PCR-DGGE 分析, 指纹图谱的聚类结果显示鲢、鳙并没有完全分开, 而是存在交叉。
本实验研究结果也显示同为草食性的草鱼和团头鲂消化道微生物群落结构并没有明显的不同, 这再次证明食性相同的鱼类消化道微生物群落结构类似,并暗示它们消化道内微生物群落对营养物质的代谢方式可能类似。
Han 等运用克隆文库方法分析了投喂商业饲料的混养池塘内草鱼肠道内容物、池塘水体、沉积物、商业饲料和芦苇的细菌组成, 研究发现草鱼肠道内主要菌群为变形菌门、厚壁菌门和放线菌门。
Wu 等以池塘饲喂黑麦草草鱼为实验对象, 通过高通量测序研究发现草鱼消化道内存在核心微生物群落, 为变形菌门、厚壁菌门和放线菌门。本实验的测序结果显示草鱼消化道内菌群主要是 γ-变形菌门、梭杆菌门还有少量厚壁菌门和拟杆菌门; 团头鲂消化道内菌群与草鱼相似, 主要为 γ-变形杆菌、梭杆菌门和厚壁菌门, 但未检测到拟杆菌门细菌,这些与前面研究人员的结果基本一致。而且检测到气单胞菌属和 Cetobacterium 属为草鱼和团头鲂消化道内共有的优势菌群, 这一研究结果与 Ni 等对池塘饲料养殖草鱼和洞庭湖野生草鱼的研究结果一致。以上研究表明来自不同生境的草鱼消化道主要微生物群落组成基本相同, 不具有生境特异性,推测其消化道内存在核心微生物组成。在研究杂食性鱼类中, 薛恒平等报道杂食性鱼类消化道内优势菌群为弧菌和气单胞菌; 周金敏通过对肉食性鱼类黄颡鱼肠道菌群分析发现其肠道内的优势菌群为气单胞菌属、肠杆菌科和不动杆菌属; 李学梅等对三种室内饲养鱼类肠道微生物群落进行PCR-DGGE 分析显示, 偏肉食性的斑点叉尾 肠道中菌群主要属于变形杆菌门, 而偏杂食性的银鲫和异育银鲫肠道中主要为梭杆菌门和气单胞属菌。以上针对杂食性和肉食性鱼类的研究结果与本研究中草食性鱼类消化道内微生物组成还是有所区别。综合以上分析, 本文推测草食性鱼类消化道内优势微生物类群基本维持不变, 宿主食性可能是维持鱼类消化道微生物群落结构的重要因素。
另外, 从本实验的研究结果还发现, 草鱼消化道微生物群落个体分化明显, 其中草鱼 2 号样本的谱带数明显高于其他两个样本; 相似的, 2 号团头鲂样本的谱带数明显低于其他两样本。且切胶测序结果同样发现草鱼和团头鲂个体间存在差异, 已有研究表明动物体消化道微生物能够受宿主发育阶段、饮食、健康状况和生活环境等因素影响。如 Fujimura等研究表明宿主的健康状况和肠道微生物相互影响, 且宿主机体的生态失衡能够改变肠道微生物群落组成。Moran 等对来自新西兰东北部海岸的海洋草食性鱼类雪梨舵鱼开展的研究发现, 雪梨舵鱼肠道细菌群落组成受鱼体不同发育阶段影响。本实验所反映的草鱼和团头鲂消化道微生物均存在个体差异, 其原因还有待进一步深入探究。随着高通量测序技术的飞速发展, 宏基因组测序已经广泛应用于动物体肠道微生物研究, 这将为研究鱼类肠道菌群个体差异提供更好的研究手段, 以期更加深入的揭示消化道微生物与鱼源宿主的精密关系。
总之, 本实验通过 PCR-DGGE 技术比较了草鱼和团头鲂消化道微生物群落结构并对特定条带进行回收测序, 检测到两种鱼消化道内主要微生物组成相似, 消化道微生物群落结构不存在显著性差异,但检测到草鱼和团头鲂个体间存在差异。
日本渔业协同组合是日本渔民在自发的基础上自愿集合、自我管理的组织结构,在日本国内渔业资源管理中充当着非常重要的角色,是连接政府、渔民的第三方中介组织。由于日本国情、制度及文化传承等方面的影响,日本渔民已习惯于加入到组织中进行渔业的日常作业...
高密度、集约化的养殖方式已成为中国淡水渔业支柱产业之一,这种养殖方式创造了巨大经济产值和社会效益的同时也对环境造成了巨大的负面影响。淡水池塘养殖是一种受人为调控影响较大的营养型养殖系统,高密度的投饵养殖方式导致残饵、粪便、排泄物等在池塘底...
鳞片是鱼类皮肤的衍生物,分盾鳞、硬鳞和骨鳞3种,除软骨鱼类和硬骨硬鳞鱼类外,其余有鳞片的鱼类都被骨鳞。骨鳞不仅可用来鉴定鱼类的年龄,还可以根据鳞片矿物元素含量监测鱼类生存环境的变化。鳞片(下文所指的鳞片均为骨鳞)约占鱼体重量的3%~5%,...
细鳞鲑(Brachymystaxlenok)隶属于鲑形目鲑科鲑亚科。在我国主要分布在东北地区黑龙江流域、绥芬河、图们江、鸭绿江,新疆的额尔齐斯河,河北省北部白河及滦河上游以及秦岭北麓渭河流域。李思忠根据幽门盲囊、侧线鳞和最小性成熟年龄等形态学和生理学特征,...
在观赏水族中,海水观赏鱼由于其色彩艳丽,形态多姿,在中高档名贵观赏鱼中大受欢迎[1-2].但目前海水观赏鱼存在着饲养困难、易死亡、海水获得难等问题,其最主要原因是海水水族箱中硝化功能在水族箱中不易建立,水处理能力较弱,海水水质不够稳定[3].水族箱...