为了对目前基于 NGS 的微生物检测数据分析方法进行全面评估,我们将分析系统简化为 6 个核心环节,如图 1 所示,分别为:质量控制(A)、比对方法优化(B)、与参考基因组进行比对(C)、序列拼接(D)、与微生物基因组进行比对(E)、下游分析(F)。其中质量控制(环节 A)属于提高微生物检测精度的方法,通常 NGS 产生的测序数据会存在低质量序列、低复杂度序列等,会影响后续分析。因此需要通过质量控制软件对样本测序数据进行处理。比对方法优化(环节 B)属于提高微生物检测速度的方法,由于 NGS 产生上百万片段,数据分析在单个节点上的计算时间可能需要数周,因此需要采用设计算法的查询和匹配效率,以降低数据分析所需的运算量,在数据分析环节上提高速度。序列比对(环节C,E)为微生物检测的核心步骤[12],将样本数据 和 人 类 参 考 基 因 组(human reference geneset,HRG)或 微 生 物 参 考 基 因 组(microbe referencegeneset,MRG)进行比对,清除与HRG比对成功的序列,保留与MRG比对成功的序列,最后获得已知和未知的微生物基因。序列拼接(环节D)将检测出的微生物基因短reads拼接成完整微生物基因序列,从而进行下游分析(环节 F),如可视化[13]、单核苷酸多态性(single nucleotide polymorphisms,SNP)分析[14]等,才能获得基因序列的全部信息,完成微生物检测的整个流程。
2基于NGS的微生物检测数据分析方法。
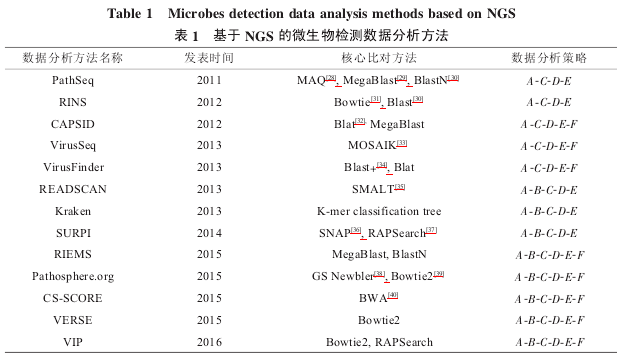
从2011年开始,基于NGS的微生物检测数据分析方法得到了快速的发展。目前已经发表的数据分析方法包括PathSeq[15]、RINS[16]、CAPSID[17]、VirusSeq[18]、 VirusFinder[19]、 READSCAN[20]、Kraken[21]、SURPI[22]、RIEMS[23]、Pathosphere.org[24]、CS-SCORE[25]、VERSE[26]和VIP[27]等,如表1所示。
参照前文提到的数据分析策略,可以发现不同的微生物检测计算分析方法主要体现在比对方法优化(环节 B)以及下游分析(环节 F)。早期的数据分析方法如PathSeq、RINS 等,是最初微生物检测数据分析的典型解决方案,并未专门对比对方法进行优化,也缺少下游分析的步骤,属于“基础型”检测方法。而 VirusSeq、VirusFinder、CaPSID 等,在“基础型”的基础上,增加了如病毒结合位点分析等下游分析(环节 F),其检测功能得到了完善,称为“功能型”检测方法。而 READSCAN、Kraken、CS-SCORE在“基础型”的基础上,增加了比对方法优化(环节 B),其处理速度大大增加,属于“速度型”检测方法。近几年出现的 SURPI、RIEMS、Pathosphere.org、VERSE 和 VIP,同时包含比对方法优化和下游分析,能够提供完整的分析流程,属于“完整型”检测方法。
除了数据分析策略带来的基础特点外,由于采用了不同的软件、处理流程等,不同的基于 NGS的微生物检测数据分析方法还有着不同的优势,如图2 所示。
原文出处:周子寒,彭绍亮,伯晓晨,李非. 基于高通量测序技术的微生物检测数据分析方法[J]. 生物化学与生物物理进展,2017,(01):58-69.
相关内容推荐
-
微生物组大数据分析的方法流程与发展趋势
微生物群落是地球上生命基本元素(C、N和S等)进行生物地球化学循环的主要驱动力,与人类健康、环境保护以及工农业生产等密切相关。近十年来,随着高通量测序的广泛应用,微生物组学成为新兴概念和热点。微生物组与不同的生存环境结合,诞生人体微生物组,宿...
相关标签: