蜘蛛丝拥有优异的机械和生物学性能,是一种理想的多功能材料。由于蜘蛛自相捕食和产丝量小等,通过驯养蜘蛛获取大量蛛丝纤维的难度较高,因此采用生物技术的方法重组表达蜘蛛丝蛋白成为获取仿生蜘蛛丝纤维的主要途径。
到目前为止,在蛛丝仿生领域仍未取得突破,仿生的拟蛛丝纤维性能远不及天然蛛丝。研究表明蛛丝蛋白的高分子量特性和成丝模式是决定丝纤维性能的重要因素,现已成为研究的热点。典型的蛛丝蛋白由氨基端(N terminal,NT)、羧基端(C terminal,CT)以及占到>90%的重复区(Re-peat,R)构成。NT 和 CT 高度保守,在蛛丝蛋白的分泌、储存和成丝等过程中起重要的调节作用。
随着 pH 的降低(中性到酸性),NT 二聚化,形成蛋白网络;CT 则能促进蛛丝蛋白的排列和可溶性的提高等,具体功能尚不清楚;R 主要与蜘蛛丝性能相关。另外,蛛丝蛋白纤维化还受到盐离子的影响,如氯化钠能够维持主壶腹腺丝蛋白(major ampullate spidroin,MaSp)NT 单体的稳定性等。由于主壶腹腺体容易分离,分泌的主壶腹腺丝性能突出等优点,长期以来一直受到青睐。
2012 年本课题组获得首条次壶腹腺丝蛋白(Minorampullate spidroin,MiSp)全长编码序列,经比对分析得知 MiSp 和 MaSp 的 CT 同源性较高,但MaSp CT 含有两个保守半胱氨酸(Cysteins),通过二硫键相互作用,形成平行二聚体;MiSp CT 则没有 Cys,二聚化机制和生物学功能尚未得到充分证实。
为了进一步研究蜘蛛丝蛋白 MiSp 重复区 R和羧基端 CT 的二级结构和功能,本实验通过克隆并在大肠杆菌 Rosetta 2(DE3)中分别表达R1R2CT和 R1R2 两个组合模块蛋白,借助 Ni-NTA 亲和色谱纯化,利用圆二色谱(circular dichroism,CD)测定并分析了不同 pH 条件下的重组蛋白 R1R2和 R1R2CT 二级结构特性;采用扫描电子显微镜(scanning electron microscope,SEM) 表征 R1R2 和R1R2CT 重组蛋白在不同 pH 及离子条件下的聚集和成丝情况,为成丝机理的研究奠定基础。
1 材料与方法
1.1 材料
质粒抽提试剂盒、DNA 胶回收试剂盒均购自上海生工生物工程有限公司;EcoRⅠ、HindⅢ、BsaⅠ限制性内切酶和 phusion High-Fidelity PCRMM buffer 购自 NEB 公司 (美国);T4 DNA 连接酶购自 Fermentas 公司 (美国);6×his 抗体购自天根公司;Ni-NTA 树脂购自 Qiagen 公司(德国)。
1.2 方法
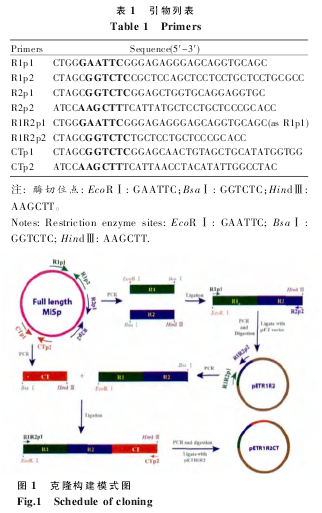
1.2.1 克隆构建
以 MiSp 全长基因为模板 (GenBank 登录号:
JX513956),以 R1p1 和 R1p2 为引 物 PCR 扩增R1,以 R2p1 和 R2p2 为引物 PCR 扩增 R2。BsaⅠ酶切 R1 和 R2,酶切片段回收后由 T4 连接酶体外连接 R1 和 R2;以连接产物为模板,以 R1p1 和R2p2 为引物扩增 R1R2;R1R2 由 EcoRⅠ和 Hind Ⅲ酶切,胶回收后的酶切片段与拥有相同酶切位点的载体连接。以 R1R2 为模板,以 R1R2p1 和R1R2p2 为引物扩增片段 R1R2,以 CTp1 和 CTp2为引物扩增 CT;R1R2 和 CT 分别由 BsaⅠ酶切,酶切片段回收后体外连接,连接产物用作模板,以R1R2p1 和 CTp2 为引物扩增 R1R2CT, 产 物经EcoRⅠ和 HindⅢ酶切后与载体连接,引物序列见表 1,具体克隆构建过程如图 1。
1.2.2 融合蛋白的表达
将测序正确的重组质粒转化于 Rosetta 2(DE3)感受态细胞。挑取单克隆接种于 5 mL LB 培养基(含卡那霉素 30 mg/L)。200 r/min,37 ℃过夜培养。将 5 mL 过夜培养物接种于 500 mL 新鲜LB 培养基 (含卡那霉素 30 mg/L),37 ℃震荡培养至 OD600为 0.9 左右,温度降至 25 ℃时加入终浓度为 0.3 mmol/L 的 IPTG,25 ℃诱导培养 4 h,离心 10 min (8 000 r/min,4 ℃)收集菌体,加入 30 mL20 mmol/L Tris pH 8.0 重悬菌体,200 W 超声破碎(超声 5 s,间隔 8 s)60 次,低温高速离心 30 min(12 000 r/min,4 ℃)收集上清。
1.2.3 蛋白表达产物纯化和分析
上清中重组蛋白与 Ni-NTA 结合,由含有 50mmol/L 咪唑的 20 mmol/L Tris pH 8.0 进行完全洗涤,最后由含有 300 mmol/L 咪唑的 20 mmol/LTris pH 8.0 洗脱蛋白。纯化过程中,收集穿柱液(Flow through)、洗涤液(Wash)、洗脱液(Elution),并各取出 20 μL,加入 5 μL 的 5X SDS-PAGE 上样缓冲液,100 ℃水浴 10 min,取适量样品经 12%SDS-PAGE 电泳后进行染色和脱色分析。样品经12% SDS-PAGE 凝胶电泳后进行恒流电转移,转移到 PVDF 膜,以 6×his 组氨酸标签作为检测抗原,对表达的融合蛋白进行 Western blot 来进一步检测,具体方法参照 Invitrogen 操作手册。
1.2.4 重组蛋白二级结构测定
实验中采用 CD 测定所有蛋白的二级结构,均使用上海交通大学分析测试中心的 JASCO J-815 圆二色谱仪。终浓度为 0.1 g/L 的重组蛋白R1R2 和 R1R2CT 分别溶于 pH 5.5、6.5 和 7.5 的PBS 磷酸盐溶液中,从 190~260 nm 的波长范围收集信号,测试所用比色皿大小为 1 mm。
1.2.5 扫描电子显微镜测试分析
终浓度为 10 μmol/L 的重组蛋白 R1R2 和R1R2CT 分别溶于 6 种不同 pH 和盐离子的溶液中: 20 mmol/L PBS pH 5.5、20 mmol/L PBS pH 6.5、20 mmol/L PBS pH 7.5、20 mmol/L PBS pH 5.5(含有 154 mmol/L NaCl)、20 mmol/L PBS pH 6.5(含有154 mmol/L NaCl)、20 mmol/L PBS pH 7.5 ( 含 有154 mmol/L NaCl),于 37 ℃ 250 r/min 震荡培养12 h,培养液(纤维)由东华大学化学化工与生物工程学院日立 TM-1000 台式扫描电镜测试分析。
2 结果
2.1 重组子的鉴定
R1R2 和 R1R2CT 分别与经改造的载体 pHTH连接,重组子质粒送上海生工生物工程有限公司正反向测序,测序结果经比对和分析确定无突变,可用于后续的重组蛋白表达。
2.2 重组蛋白表达和纯化
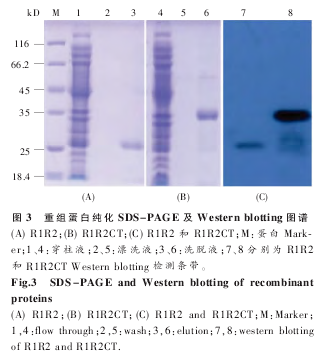
R1 和 R2 主要由甘氨酸 (Glycine) 和丙氨酸(Alanine) 组成,CT 序列中 Gly 和 Ala 较少,R1R2和 R1R2CT 的氨基酸序列见图 2。超声破碎经IPTG 诱 导表 达 的 R1R2 和 R1R2CT Rosetta 2(DE3)菌体,高速离心后的上清由 Ni-NTA 柱纯化(Qiagen 纯化方法)。目的蛋白与 Ni-NTA 结合效率较高,洗脱后重组蛋白由 SDS-PAGE 检测,图谱显示目的蛋白可达到较好的纯度 (图 3A、B);Western blotting 图谱进一步证明重组蛋白的大小和完整性(图 3C)。
2.3 二级结构测定
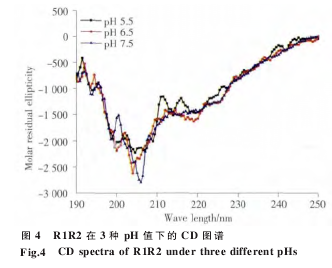
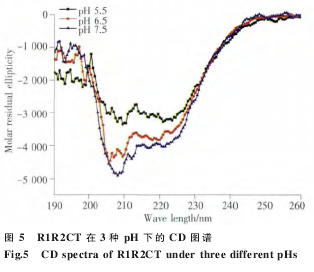
我们采用 CD 技术分别测定于 pH 7.5、6.5 和5.5 的 PBS 溶液中重组蛋白 R1R2 和 R1R2CT 的二级结构特征。R1R2 在 3 种不同的 pH 中结构相似,主要为无规卷曲构象(图 4)。R1R2CT 在 208 nm和 222 nm 处呈现负峰,是典型的螺旋峰型(图 5)。
我们的 NMR 结果证实 CT 为五 α 螺旋结构,且随着 pH 值的降低,峰值信号向上偏移(未发表)。
2.4 扫描电镜分析
重组模块蛋白 R1R2、R1R2CT 分别溶于 6 种不同 pH 值和离子条件下的溶液中:20 mmol/LPBS pH 5.5、20 mmol/L PBS pH 6.5、20 mmol/LPBS pH 7.5、20 mmol/L PBS pH 5.5 +154 mmol/LNaCl、20 mmol/L PBS pH 6.5 +154 mmol/L NaCl、20 mmol/L PBS pH 7.5+154 mmol/L NaCl,终浓度为 10 μmol/L.蛋白溶液经 250 r/min,37 ℃震荡培养 12 h 后,电镜结果显示,R1R2 和 R1R2CT 在pH 7.5 和 pH 6.5 条件下均无可见的纤维产生。pH5.5 时,R1R2CT 形成纤维束,表面形态光滑、形态整齐; 而 R1R2 则形成较为松散的纤维,表面较为粗糙,呈扁平条带状。加入 154 mmol/L 氯化钠后,R1R2CT 表面粗糙,蛋白聚集不均匀,形成纤维形态较差; RIR2 形成的纤维量较少,但形态与无氯化钠相似,表面形态较无氯化钠时粗糙且不平整。
3 讨论
蛛丝蛋白由蜘蛛腹部腺体分泌产生,如 MaSp由主壶腹腺分泌、MiSp 由次壶腹腺分泌以及鞭毛状丝蛋白(Flag)由鞭毛状腺分泌等,主壶腹腺和次壶腹腺结构相似。蛛丝蛋白在纤维化过程中 pH 值从 7.6 降到 6.3(当前可测定的最低 pH值),同时离子浓度和种类亦发生变化,最后在剪切力作用下形成高品质的丝纤维。2007 年,PloSONE 报道了首条 MaSp 全长基因组序列,蛋白序列分析表明,MaSp 由 NT、CT 以及占到 95%以上的重复区 R 组成条 MiSp 全长基因组序列,与 MaSp 相似,MiSp 亦由 NT、CT 和 R 组成,R 占主要部分。6种蛛丝纤维分别拥有不同的机械性能,主要由不同的重复区 R决定;NT 和 CT 在蛛丝蛋白的成丝过程中主要起到调节、促溶以及利于纤维蛋白排列等作用。
MiSp 重 复 区 域 R 主 要 由 poly -Alanine(polyA),Glycine -Glycine -X (X 为 其 他 氨 基 酸 ,GGX)以及 Glycine-Alanine(GA)等模块组成,组成的次壶腹腺丝拥有类似拖丝的机械性能。结构分析发现,polyA 和 GA 均能形成 β-sheet 结构,在固态丝纤维中整齐排列形成晶体区域,决定丝素蛋白的强度。GGX 形成 310-helix,形成丝纤维的无定型区域,决定丝纤维的延展性能。另外,MiSp中还带有特殊的 Spacer 区域,但到目前为止,该模块的功能尚不清楚,我们推测该模块与延展性相关。在自然进化中,重复区变化较大,但是CT 在序列和结构上高度保守,研究发现 MaSp CT可以稳定丝蛋白,阻止过早的聚集,并且能够调整重复区的排列而形成表面形态更好的丝纤维。
2010 年,NATURE 报道了 MaSp CT 的高级结构,解释了它在溶液中的结构形态和它与丝蛋白储存和自组装转变的密切相关性。另外,MaSp CT 在调整重复区 R 二级结构方面扮演着重要的角色,这对于丝纤维的机械性能具有很重要的作用。
为了进一步研究 R 和 CT 模块的功能,我们构建了 R1R2 和 R1R2CT 两个重组克隆。通过BsaⅠ限制性内切酶的介导,我们将 R1 和 R2 以及 CT 三段无缝连接(图 2),排除了由酶切位点引入外源氨基酸的干扰。由于 R1、R2 以及 CT 中均含有稀有密码子,因此我们选择 Rosetta 2 (DE3)作为表达宿主菌体来消除稀有密码子的影响。表达及纯化结果表明 (图 3),重组蛋白 R1R2 和R1R2CT 均可在 Rosetta 2 (DE3)中大量表达,采用Ni-NTA 纯化柱纯化可得到纯度较高的重组蛋白(图 3)。Western blotting 结果也证实了重组蛋白R1R2 和 R1R2CT 的完整性和纯度。R1R2 主要由ploy-A、GA 以及 GGX 组成,CD 图谱显示(图 4 和5),R1R2 重组蛋白在 PBS 溶液中主要为无规卷曲构象,R1R2CT 则拥有 α 螺旋的两个特征负峰值。
我们的其他实验数据表明,MiSp CT 为五 α 螺旋构象,随着 pH 的降低,峰图向上偏移,表明二级结构出现细微的变化 (未发表)。R1R2 为无规卷曲,R1R2CT 为 α 螺旋构象,我们推测 CT 可以促进重复区 α 螺旋等构象的形成。我们在 3 种不同的 pH 值及有无 154 mmol/L NaCl 的条件下,37 ℃250 r/min 震荡培养上述两种重组蛋白 12 h,样品经 SEM 分析 (图 6) 表明,在 pH 7.5 和 6.5 时,R1R2CT 和 R1R2 均无法形成纤维,pH 值降到 5.5时,可形成纤维。R1R2CT 可形成表面形态良好的重组纤维,且纤维成纤维束状(图 6 中显示为 4 根单纤维的混合),单根纤维表面光滑,呈立体圆形,大小均匀。只有重复区的 R1R2 重组蛋白则不同,形成的纤维为扁平带状,大小不均匀。文献记载MaSp 重复区 R 重组蛋白无法形成纤维,只有加上 CT 后才能形成可见纤维。通过本实验,我们证实单独的 MiSp R1R2 重组蛋白也可形成纤维,只是纤维形态较 R1R2CT 不平整,呈条带状。
因此,CT 有利于丝蛋白的排列组装而形成较为紧密的重组纤维。NaCl 能够维持 MaSp NT 单体稳定性,不利于丝蛋白的纤维化。这里我们进一步证实,在 154 mmol/L NaCl 存在的条件下,R1R2和 R1R2CT 均可形成纤维,但形成纤维形态不均匀,无法呈现出立体圆形。
本次研究通过对 R1R2 和 R1R2CT 的表达、纯化以及二级结构和成丝倾向的分析,证实了在不同 pH 值的 PBS 中 R1R2 为无规卷曲构象,而R1R2CT 则主要为螺旋构象。pH 值 6.5~7.5 不利于丝素蛋白的纤维化,pH 值为 5.5 时,纤维化形态较好。另外,我们还分析了 NaCl 对成丝的影响,结果表明 NaCl 不利于高品质纤维的形成。本研究为成丝机理的研究以及通过基因工程方法仿生蜘蛛丝奠定了基础。
参考文献(References):
[1] GILMAN J J. Strength of spider silk[J]. Science, 1996, 272(5258):17a.
[2] VOLLRATH F. Strength and structure of spiders silks[J]. Jour-nal of Biotechnology, 2000, 74(2): 67-83.
[3] LEWIS R V. Spider silk: ancient ideas for new biomaterials[J].Chemical Reviews, 2006, 106(9): 3762-3774.
[4] KLUGE J A, RABOTYAGOVA O, LEISK G G, et al. Spidersilks and their applications[J]. Trends in Biotechnology, 2008,26(5): 244-251.
[5] RISING A. Controlled assembly- a prerequisite for the use ofrecombinant spider silk in regenerative medicine? [EB/OL].Acta Biomaterialia
[6] HAUPTMANN V, WEICHERT N, MENZEL M, et al. Native-sized spider silk proteins synthesized in planta via intein -based multimerization[J]. Transgenic Research, 2013, 22(2): 369-377.
[7] XIA X X, QIAN Z G, CS K I, et al. Native-sized recombinantspider silk protein produced in metabolically engineered Es -cherichia coli results in a strong fiber[J]. Proceedings of the Na-tional Academy of Sciences of USA, 2010, 107 (32): 14059-14063.
[8] AYOUB N A, GARB J E, KUELBS A, et al. Ancient proper-ties of spider silks revealed by the complete gene sequence ofthe prey-wrapping silk protein (AcSp1)[J]. Molecular Biologyand Evolution, 2013, 30(3): 589-601.
[9] CHEN G, LIU X, ZHANG Y, et al. Full-length minor ampul-late spidroin gene sequence[J]. PLoS one, 2012, 7(12): e52293.
[10] AYOUB N A, GARB J E, TINGHITELLA R M, et al. Blueprintfor a high-performance biomaterial: full-length spider draglinesilk genes[J]. PLoS one, 2007, 2(6): e514.
[11] HAGN F. A structural view on spider silk proteins and theirrole in fiber assembly[J]. Journal of Peptide Science, 2012, 18(6): 357-365.
[12] GAINES W A, SEHORN M G, MARCOTTE W R. SpidroinN -terminal domain promotes a pH -dependent association ofsilk proteins during self-assembly[J]. The Journal of BiologicalChemistry, 2010, 285(52): 40745-40753.
[13] ANDERSSON M, HOLM L, RIDDERSTRALE Y, et al. Mor-phology and composition of the spider major ampullate glandand dragline silk[J]. Biomacromolecules, 2013, 14(8): 2945-2952.
[14] HAGN F, EISOLDT L, HARDY J G, et al. A conserved spidersilk domain acts as a molecular switch that controls fibre as-sembly[J]. Nature, 2010, 465(7295): 239-242.
[15] VOLLRATH F. Biology of spider silk[J]. International Journalof Biological Macromolecules, 1999, 24(2-3): 81-88.。
[16] JEFFERY F, LA MATTINA C, TUTON-BLASINGAME T, etal. Microdissection of black widow spider silk -producingglands [J]. Journal of Visualized Experiments (Video Journal),2011, (47): 2382.
[17] ORTIZ R, CESPEDES W, NIEVES L, et al. Small ampullateglands of Nephila clavipes[J]. The Journal of Experimental Zo-ology, 2000, 286(2): 114-119.
[18] EISOLDT L, THAMM C, SCHEIBEL T. Review the role ofterminal domains during storage and assembly of spider silkproteins[J]. Biopolymers, 2012, 97(6): 355-361.
[19] HUDSPETH M, NIE X, CHEN W, et al. Effect of loading rateon mechanical properties and fracture morphology of spidersilk[J]. Biomacromolecules, 2012, 13(8): 2240-2246.
[20] JENKINS J E, SAMPATH S, BUTLER E, et al. Characterizingthe secondary protein structure of black widow dragline silkusing solid-state NMR & X-ray diffraction[J]. Biomacromolecules,2013, 14(10): 3472-3483.
[21] PARKHE A D, SEELEY S K, GARDNER K, et al. Structuralstudies of spider silk proteins in the fiber[J]. Journal of MolecularRecognition, 1997, 10(1): 1-6.
[22] YAMAGUCHI E, YAMAUCHI K, GULLION T, et al. Struc-tural analysis of the Gly-rich region in spider dragline silk us-ing stable-isotope labeled sequential model peptides and sol-id-state NMR[J]. Chemical Communications, 2009(28): 4176-4178.
[23] COLGIN M A, LEWIS R V. Spider minor ampullate silk pro-teins contain new repetitive sequences and highly conservednon-silk-like "spacer regions"[J]. Protein Science, 1998, 7(3):667-672.
[24] TOKAREVA O, JACOBSEN M, BUEHLER M, et al. Struc-ture-function-property-design interplay in biopolymers: spidersilk[EB/OL]. Acta Biomaterialia
[25] RAMMENSEE S, SLOTTA U, SCHEIBEL T, et al. Assemblymechanism of recombinant spider silk proteins[J]. Proceedingsof the National Academy of Sciences of USA, 2008, 105(18):6590-6595.
[26] VEZY C, HERMANSON K D, SCHEIBEL T, et al. Interfacialrheological properties of recombinant spider -silk proteins [J].Biointerphases, 2009, 4(3): 43-46.