第三章 口服益生菌对血液透析病人的肠道菌群及短链脂肪酸的调节作用。
3.1 前言。
肠道短链脂肪酸(short chain fatty acids,SCFAs)是肠道菌群代谢产物中最主要的标志物之一,由肠道菌群分解碳水化合物产生,是结肠中的基础组分,是肠道内为消化的碳水化合物[62],具有刺激结肠收缩的功能[63],多数研究的是乙酸、丙酸和丁酸,这三个酸是占肠道内短链脂肪酸的绝大部分[64。其中,乙酸、丙酸可被人体吸收,进入肝脏代谢供能。丁酸作为结肠细胞的主要能量来源,可被其迅速吸收[65],对维持结肠黏膜正常功能有重要作用,可抑制炎症反应及致癌作用,加强结肠防御屏障。肠道内 SCFAs 的含量多少说明了肠道菌群是否有活力,还代谢肠上皮细胞,所以短链脂肪酸对于肠道环境很重要。下面分别介绍这三种酸,乙酸来自于盲肠参与身体重要器官的代谢[66] [67]。丙酸是为肝脏代谢提供能量的[68]。丁酸是被结肠的上皮细胞吸收利用,为结肠和盲肠提供动力[69],对结肠和盲肠的细胞分化和生长起到了不可复制的作用[ 70]。肠道菌群中的厚壁菌门主要代谢产物就是丁酸,上述表明,SCFAs 对改善肠道菌群平衡及肠道功能是有重要作用的[71],并且短链脂肪酸还与肠道的健康有关联[72]。副干酪乳杆菌 L9 就可以通过肠道菌群中的短链脂肪酸来调节肠道环境。但是该菌株对肠道内哪种短链脂肪酸产生影响还不明确。但它提供了一种思路,就是用益生菌这类活的微生物,每天的足量摄入来达到改善肠道环境[73],促进肠道发育、调节肠道菌群的作用。另外益生菌的代谢产物主要为乙酸和极少量甲酸、丙酸等有机酸类物质,这些物质可促进肠管蠕动,从而产生通便感觉,也使肠道润活起来。上述这些症状的改善很大原因是益生菌对肠道短链脂肪酸有调节作用,使肠道菌群得到改善,相互作用,有益菌也在增多,从而可以使肠道内的酸碱度平衡,肠道致病菌减少[74]。有一项研究发现[75],胃肠疾病患者的粪便丁酸含量是要低于健康人的水平,但连续服用酵母菌 6 天后,患者粪便中的丁酸含量明显升高,肠道内的总 SCFAs 的水平也有所提高。SCFAs含量的变化成为了研究益生菌对肠道影响的重要指标[76]。因此本章通过对食用口服益生菌的实验对象粪便 SCFAs 测定,研究口服益生菌对肠道菌群的影响。

正常人肠道内寄生着大量微生物,其至少包含 500 个菌种,细菌总量超过1011 个,这些微生物统称为肠道菌群,正常肠道菌群以双歧杆菌、大肠杆菌、类杆菌、粪肠球菌等专性厌氧菌为主[ 77]。双歧杆菌、乳酸菌等益生菌与肠粘膜上皮细胞相互作用,通过与病原菌进行营养竞争,调节肠道微生态的组成,可促进厌氧革兰阳性菌的生长,抑制革兰阴性菌的生长,并可增强宿主吞噬细胞的活性,增强细胞免疫功能[78、77]。近年来,科学研究表明,肥胖[79]、糖尿病[80–83]、便秘以及炎症性肠病等很多疾病都与肠道菌群相关[84],导致其体内肠道菌群存在的数量与组成形式发生明显的改变。如美国 Gordon 实验室在肥胖关系和肠道微生物研究方面做了大量工作,他们证实肠道微生物会直接参与了宿主能量的摄取、利用和储存。另外,肠道菌群也与心脑血管疾病,精神疾病,胃肠疾病等疾病有关。国内外研究学者认识到人类健康和疾病与肠道菌群的密切关系,许多研究分析了肠道菌群本身以及其代谢产物在人体能量代谢、胃肠道功能和对营养物质的吸收等方面发挥着重要作用[85]。肠道菌群通过发酵多糖和蛋白质,产生小分子物质,促进营养物质被肠道吸收。健康的肠道菌群通过竞争性争夺营养、分泌细菌素、发酵产酸降低肠道内 pH等,来抑制有害菌生长,以维持肠道内环境稳态。当肠道完整性遭到破坏时,有害菌大量生长繁殖,可能造成细菌移位,引发感染。肠道作为人体最大的免疫器官,对免疫调节有重要作用[86]。正常的菌群与肠壁内的淋巴细胞以肠黏膜为界线,二者相互作用并处于平衡状态,两者平衡若被破坏,则会导致疾病的发生。肠道菌群不仅可调控肠道活动,还可影响宿主的脑功能和行为。肠道菌群通过肠-脑轴对宿主的应激反应、焦虑抑郁、认知能力产生重要影响,从而调控宿主行为。平衡的肠道菌群可促进宿主身心健康,而失调的肠道菌群则可能引发肠-肾疾病[87]。因此研究肠道菌群的变化具有重要意义,本章将采用 16s 高通量测序方法研究口服益生菌对实验对象肠道菌群的变化影响。
3.2 材料与方法。
3.2.1 主要试剂。
3.2.1.1 DNA 提取试剂。
PBS:NaCl 8.0 g;Na2HPO4?12H2O 2.9 g KCl 0.2 g;KH2PO40.2 g;溶于 800 mL去离子水,盐酸调 pH 到 7.4,再超纯水定容到 1 L;121℃灭菌 15min 备用。
10%Tris-SDS:Tris 6.06 g;浓盐酸 2.1 mL;0.5 M EDTA 4 0 mL;加 40 mL 超纯水,溶解后;调 pH=9.0,定容到 250 mL;再加 50 mL 10%SDS,混匀。
3 mol/L 乙酸钠:三水合醋酸钠 40.8 g,加 40 mL 超纯水溶解,冷却至室温后,用冰醋酸调 pH=5.2,超纯水定容到 100 mL,121℃灭菌 15min 备用。
氯仿:异戊醇(24:1):氯仿 96 mL,异戊醇 4 mL;均匀摇晃,静置于棕色试剂瓶中,备用。1 M Tris-HCl:Tris 12.14 g;加入超纯水 80 mL,用浓 HCl 调至 pH=8.0,定容至 100 mL,121℃灭菌 15min 备用。70%乙醇:70 ml 无水乙醇,30 ml 超纯水。
10×TE 缓冲液(pH8.0):1 M Tris-HCl (pH=8.0) 10 mL; 0.5 M EDTA (pH=8.0) 2mL,定容到 100 mL,灭菌,稀释 10 倍使用。
RNA 酶 RT405-02 规格 1 ml3.2.1.2 短链脂肪酸试剂。庚酸乙醚溶液:庚酸 71 μL,无水乙醚定容 100 ml,放冰上备用。1 M 盐酸备用。
标品制备:乙酸标品 2.9 μL、丙酸标品 3.7 μL、丁酸标品 4.6 μL、异丁酸标品4.6 μL,异戊酸标品 5.5 μL,并用庚酸/乙醚溶液分别定容至 10 ml 并倍比稀释为 5mM、2.5 mM、1.25 mM、0.625 mM、0.312 mM、0.156 mM 的各酸标准溶液放冰上备用。
3.2.2 主要设备。
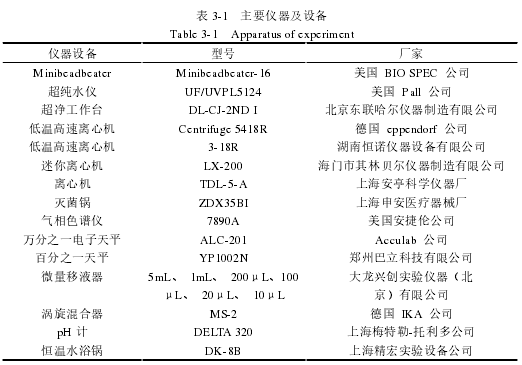
实验使用仪器及设备如表 3-1 所示:
3.2.3 研究样品。
样品选取的是基线 V0 期和干预两个月 V2 期和干预六个月 V6 期,共 60 个样品。
3.2.4 粪便短链脂肪酸提取与测定实验步骤。
3.2.4.1 实验材料。
乙酸、丙酸、丁酸、乙醚(以上分析纯),庚酸、盐酸,2 ml 螺口离心管(海门订购),微量进样器(蓝弋),冰盒,容量瓶,直径为 2-3 mm 的玻璃珠3.2.4.2 实验步骤。
(1)样品预处理① 准备预处理管,于 2 ml 螺口离心管内加入适量玻璃珠,用毛细管柱 FFAP加入 1 ml 含有内标庚酸(5 mmol/L)的乙醚溶液,及 50 μl 的 1M 盐酸,样品预先称重,于-20℃保存。
② 加入粪便样品(约 0.1-0.2 g),再称重,记录加入粪便重量。
③ 配平,采用 minibBB 破碎,30s 两次。
④ 离心 10000rpm,2min, 冰上转移上清液至新的螺口离心管。-20 度保存待用。
注:乙醚极易挥发,所有步骤均冰上操作,动作迅速,及时盖盖,(2)样品测定用自动进样器吸取 2 μl 样品进样,采用毛细管柱 FFAP 跑样,火焰离子检测器进行检测。气相程序如下:Detect =230℃,Inject= 230℃,Aux=230℃,Colomn:
50℃ 1min,50-140℃ 10℃/min,140℃ 1min,140-240℃ 30℃/min,240℃ 2min(3)标准曲线制作配制含有 5 mmol/L 乙酸、5 mmol/L 丙酸、5 mmol/L 丁酸、5 mmol/L 庚酸的乙醚溶液,采用倍比稀释的方法,得到分别含有 2.5,1.25,0.625,0.312,0.156 mmol/L的乙酸、丙酸、丁酸、庚酸的标准溶液。采用(2)中程序跑样,作出标准曲线。
(4)短链脂肪酸含量计算按照上述步骤,测定得到乙酸、丙酸、丁酸、异丁酸和异戊酸的峰面积。分别计算得到乙酸、丙酸、丁酸、异丁酸和异戊酸与内标庚酸的峰面积比,将该值带入回归方程,即可求得样品上清液中乙酸、丙酸、丁酸、异丁酸和异戊酸的浓度,换算后除以样品的取样重量可得到每毫克粪便样品中上述五种短链脂肪酸的含量。
3.2.5 16S 高通量测序方法与步骤。
3.2.5.1 粪便提取 DNA。
(1)实验室准备工作:
①将无活菌的实验用品及试剂放入超净台中,紫外灯照射。
②打开低温离心机,调到 15000rpm,5min,4℃(制冷 15-20min 左右)。
(2)准备器材:
① 1000 μl 枪头两盒。
② 拧盖的 2 ml 离心管。
③ 玻璃珠(2 ml 离心管,0.3 g 左右)。
(3)实验步骤:(在冰盒中进行)超净台操作(除离心),使用灭菌离心管和枪头。
① 取 200 μl 样品液匀浆至拧盖的 2 ml 离心管。
② 加入 1 mlPBS,用漩涡振荡器混合均匀(或用手上下颠倒),离心(15000rpm,5min,4℃)。
③ 弃 1 ml 上清,重复 2 次。
④ 弃 1 ml 上清,加入 0.3 g 左右玻璃珠,300 μlTris-SDS,500 μlTE 饱和酚。
⑤ 震荡呈乳白色,在均质机(Beat-Bitter)中均质 30s,离心(15000rpm,5min,4℃)。
⑥ 将上层悬浊液 400 μl(2×200)转移到一个新的离心管中(圆头),分别加入 400μl(酚:仿:异戊醇=25:24:1)(200 μlTris 酚,200 μl 氯仿/异戊醇)。
⑦ 手摇(10-20 次)(上下颠倒即可,轻柔),混匀呈白色,离心(15000rpm,5min,4℃)。
⑧ 将 250 μl(2×125) 上层悬浊液转移到新的 1.5 ml 离心管中,加入 25 μl3M 的乙酸钠(pH=5.3),加入 300 μl 异丙醇,手摇混匀后离心(15000rpm,5min,4℃)。
⑨ 弃上清(直接倒掉即可),加入 500 μl 70%乙醇,手摇混匀,离心(15000rpm,5min,4℃)。
⑩ 弃上清(直接倒掉即可),再用小离心机(12000rpm,1min,常温),用枪头(斜)吸去多余液体,开盖,室温干燥 15min(注意计时)。加入 100 μlTE, 5μl RNA 酶,弹混(轻),小离心机轻微离心(到 6000r 立即停止), 37℃恒温箱放置 10min。
3.2.5.2 16s 多样性材料与方法。
(1)测序实验步骤。
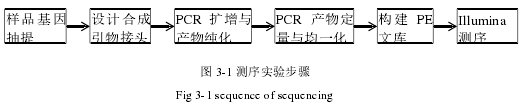
① DNA 抽提。
根据 E.Z.N.A.? soil 试剂盒 (Omega Bio-tek,Norcross,GA,U.S.)说明书进行总 DNA 抽提,DNA 浓度和纯度利用 NanoDrop2000 进行检测,利用 1%琼脂糖凝胶电泳检测 DNA 提取质量。
② PCR 扩增。
引物:用 338F (5’-ACTCCTACGGGAGGCAGCAG-3’):
806R (5’-GGACTACHVGGGTWTCTAAT-3’) 。
扩增区域:V3-V4 可变区进行 PCR 扩增。
扩增程序为:95℃ 预变性 3min,27 个循环(95℃ 变性 30s,55℃ 退火 30s,72℃ 延伸 30s),最后 72℃延伸 10min(PCR 仪:ABI GeneAmp? 9700 型)。扩增体系为 20 μl,4 μl 5*FastPfu 缓冲液,2 μl 2.5mM dNTPs,0.8 μl 引物(5 μM),0.4 μlFastPfu 聚合酶;10 ng DNA 模板。PCR 采用 TransGen AP221-02:TransStartFastpfu DNA Polymerase;PCR 仪:ABI GeneAmp? 9700 型;全部样本重复 3 遍,将同一样本的 PCR 产物混合,然后用 2%琼脂糖凝胶进行电泳检测,然后使用AxyPrepDNA 凝胶回收试剂盒,并切胶回收 PCR 产物,用 Tris-HCl 缓冲液洗脱,之后进行 2%琼脂糖电泳检测; 氢氧化钠变性,产生单链 DNA 片段。试剂:
TruSeqTM DNA Sample Prep Kit。
③ Miseq 文库构建数据处理。
原始测序序列使用 Trimmomatic 软件质控,使用 FLASH 软件进行拼接:引物允许 2 个碱基的错配,去除模糊碱基;根据重叠碱基 overlap 将两端序列进行拼接。
去除无法拼接的序列。使用的 UPARSE 软件,根据 97%的相似度对序列进行 OTU聚类;使用 UCHIME 软件剔除嵌合体。利用 RDP classifier (http://rdp.cme.msu.edu/)对每条序列进行物种分类注释,比对 Silva 数据库(SSU123),设置比对阈值为70%。通过 PCR 将 Illumina 官方接头序列添加至目标区域外端;④ Illumina Miseq 测序。
使用 2%琼脂糖凝胶回收 PCR 产物,利用 AxyPrep DNA Gel Extraction Kit 进行纯化,再用 Tris-HCl 进行洗脱,然后进行 2%琼脂糖电泳检测。利用 QuantiFluor?-ST (Promega,USA) 进行检测定量。根据 Illumina MiSeq 平台 (Illumina, SanDiego,USA)标准操作规程将纯化后的扩增片段构建 PE 2*300 的文库。1) 要使引物碱基与 DNA 片段一端与互补,所以要将其固定在芯片上;2) 以 DNA 片段为模板,芯片上固定的碱基序列为引物进行 PCR 合成,在芯片上合成目标待测 DNA片段;3) 变性和退火完成后,芯片上的 DNA 片段的另一端,并且随机与附近的另一个引物形成互补,也要将其固定住;4) PCR 扩增,产生 DNA 簇;5) DNA 扩增子线性化成为单链;6) 加入带有4种荧光标记的dNTP和改造后的DNA聚合酶,使得每次的循环均只合成一个碱基;7) 再用激光扫描反应板的表面,以便读取每条模板序列在第一轮的反应中在反应板的表面所聚合的核苷酸种类;8) 还要将“终止基团”和“荧光基团”进行化学切割,继续进行聚合第二个核苷酸的进程;9) 最后要统计分析这几轮收集到的荧光信号的结果,从而获知模板DNA片段中的序列。
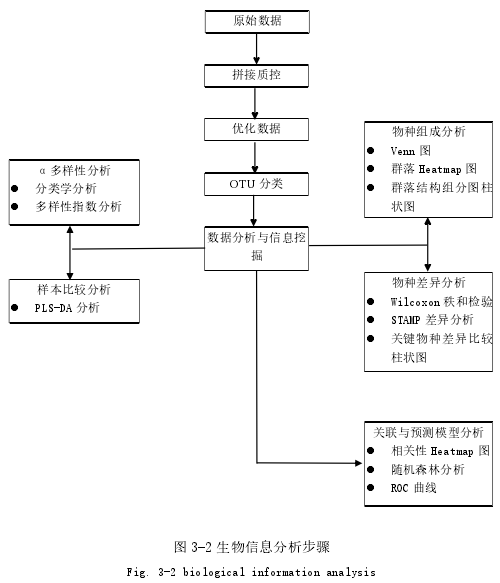
(2)生物信息分析步骤。
Miseq 测序首先根据 overlap 关系的拼接,和对序列质量进行质控和过滤之后将样本区别后进行 OTU 聚类分析和物种分类学水平分析。在 OUT 的基础上可以进行对实验样品的多种多样性指数分析,也可根据 OTU 聚类分析的结果,对测序进行深度的检测。根据所得到的分类学信息,可以在各个分类水平上对实验样品进行群落结构的分析统计。得到了上述分析之后,能够对多样本之间的群落组成信息进行多元分析和差异显着性检验等一系列更为深入详细的统计学分析和直观性可视化分析,图 3-2 如下:
第4章双鸭山市农村公共文化服务体系存在的问题及原因4.1存在问题4.1.1农村公共文化设施陈旧,总量偏少随着社会各界对农村公共文化服务建设的重视程度加深,双鸭山市建立的多个文化设施在上级部门的支持下得到了快速发展。但是目前而言,文化站的场地...
第三章沈北新区失地农民安置现状及存在问题3.1失地农民安置现状3.1.1失地农民现状失地农民主要集中分布于沈北新区近郊处,主要有沈北新区道义街道、虎石台街道、辉山街道、新城子街道等一系列街道村屯。这此街道紧连沈北新区区中心,经济基础相对较好...
第4章养猪业可持续发展存在问题及成因分析4.1养猪业可持续发展存在问题结合可持续发展理论及养猪业访谈调查结果,我国目前影响养猪业可持续发展存在的主要问题有以下几点:4.1.1产业环境缺乏压力我国养猪业所存在最为关键的问题,在于养猪业是一种...
第1章绪论1.1研究背景当前,随着城镇化进程的快速发展,造成了大规模的城市扩建行动。当城市扩展到城中村、周边村时,自然而然产生了大量的失地农民,他们因为想种地却无地可种、想上班却无班可上、想买房却无钱购买而成为了一个庞大的特殊群体,现在...
第7章结论7.1主要结论玉米是牡丹江市主栽粮食作物,其生产资源条件得天独厚,产业开发空间巨大,农产品市场前景好,玉米产业已经成为牡丹江市的主导产业之一。但从目前来看,牡丹江市玉米产业发展潜力还远未充分发掘出来,玉米产业发展存在一些问题。...
第3章牡丹江市大豆产业发展现状3.1牡丹江市大豆生产情况大豆产业是牡丹江市重要的粮食作物,又是重要的油料作物,是部分农民收入重要来源。在2009年数据来看,全市现有耕地面积641万亩,2009年全市大豆播种面积348万亩,大豆总产量在50万吨左右...
第四章农村信息化的未来发展趋势4.1农村信息化发展现状及趋势4.1.1当前我国农业发展的特点解决三农问题的主要手段就是农村的信息化,这也同时是建设社会主义新农村的重中之重。从古今中外的发展历程与经验看,农村信息化主要经历了三个阶段:一是以建设...
第五章湖南林业可持续发展的对策湖南省是我国南方的重点林区,实现全省林业的可持续发展具有重要的战略意义。5.1树立生态文明理念在新的历史形势下,实现林业可持续发展必须切实转变旧有思想观念,以生态文明的理念统领林业的可持续发展,全面加强林业...
第二章研究的目的、意义、内容、技术路线及方法2.1研究的目的、意义在国民经济发展中,林业的经济效益和社会效益是不容忽视的,这也是一个国家是否繁荣昌盛,是否达到文明和发达水平的一个重要标志。一段时期以来,湖南作为南方的重点林区,现有的森林...
农村垃圾处理与城市垃圾处理同等重要,农村生活垃圾如果不能得到及时的处理或者处理不当,可能滋生一些病毒细菌,这将直接影响着人们的健康状况,它将是全面建成小康社会道路上的拦路虎。...